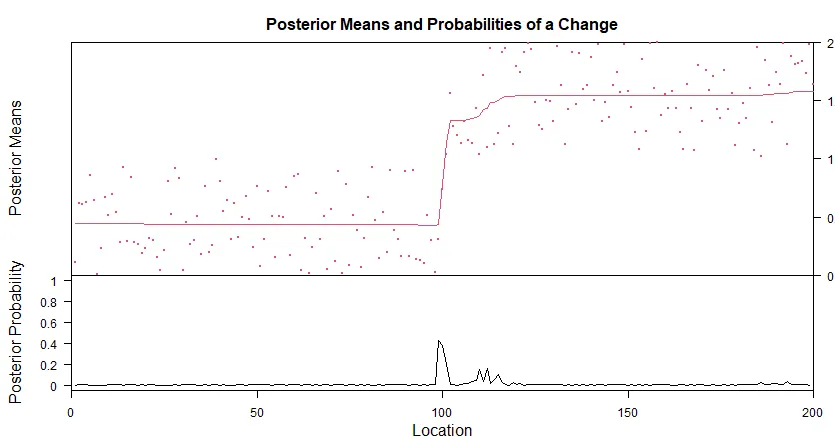
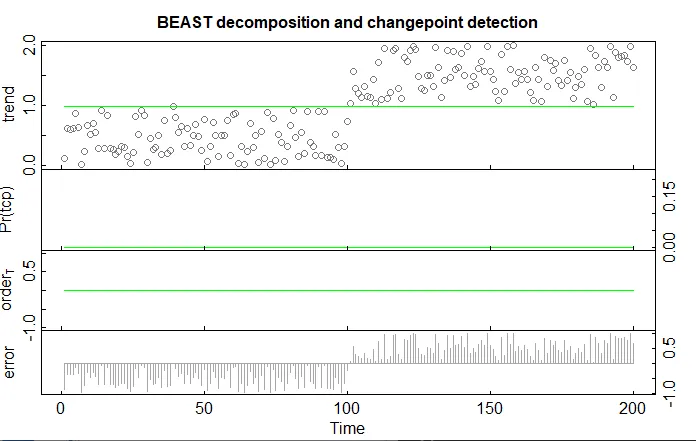
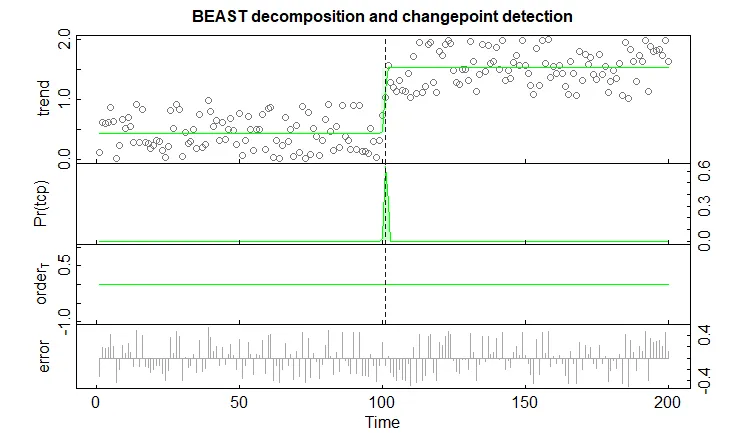
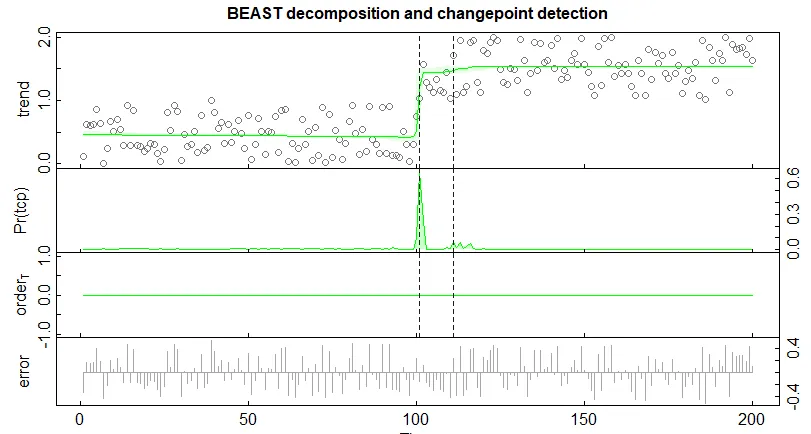
我想运行一个MCMC线性高斯多个变点模型,以检测连续值时间序列向量的变点。
在这样做时,我考虑使用MCMCregressChange函数,但我有几个问题:
(1) 如何获得这些模型的对数边际似然?
(2) MCMCregressChange函数和MCMCresidualBreakAnalysis函数有什么区别?
R脚本如下所示。如果你能帮我解决这个问题,我会非常高兴。
library(MCMCpack)
set.seed(1234)
n <- 100
x1 <- runif(n, min = 0, max = 1)
x2 <- runif(n, min = 1, max = 2)
X <- c(x1,x2)
B0 <- 0.1
sigma.mu=sd(X)
sigma.var=var(X)
model0 <- MCMCregressChange(X ~ 1, m=0, b0=mean(X), mcmc=100, burnin=100, verbose = 1000,
sigma.mu=sigma.mu, sigma.var=sigma.var, marginal.likelihood="Chib95")
model1 <- MCMCregressChange(X ~ 1, m=1, b0=mean(X), mcmc=100, burnin=100, verbose = 1000,
sigma.mu=sigma.mu, sigma.var=sigma.var, marginal.likelihood="Chib95")
model2 <- MCMCregressChange(X ~ 1, m=2, b0=mean(X), mcmc=100, burnin=100, verbose = 1000,
sigma.mu=sigma.mu, sigma.var=sigma.var, marginal.likelihood="Chib95")
print(BayesFactor(model0, model1, model2))
plotState(model0)
plotChangepoint(model0)
plotState(model1)
plotChangepoint(model1)
plotState(model2)
plotChangepoint(model2)