我在使用 autoplot 绘图的 R 教程中看到了这个。他们绘制了载荷和载荷标签:
autoplot(prcomp(df), data = iris, colour = 'Species',
loadings = TRUE, loadings.colour = 'blue',
loadings.label = TRUE, loadings.label.size = 3)
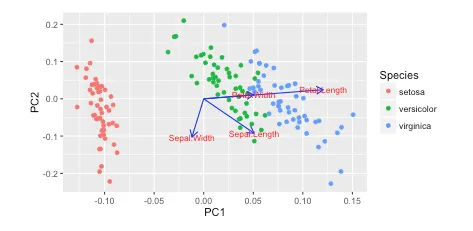 https://cran.r-project.org/web/packages/ggfortify/vignettes/plot_pca.html
https://cran.r-project.org/web/packages/ggfortify/vignettes/plot_pca.html
我更喜欢使用 Python 3 、matplotlib、scikit-learn 和 pandas 进行数据分析。但是,我不知道如何添加它们?
如何使用matplotlib绘制这些向量?
我一直在阅读Recovering features names of explained_variance_ratio_ in PCA with sklearn,但还没有弄清楚。
以下是我在Python中绘制的方式。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn.preprocessing import StandardScaler
from sklearn import decomposition
import seaborn as sns; sns.set_style("whitegrid", {'axes.grid' : False})
%matplotlib inline
np.random.seed(0)
# Iris dataset
DF_data = pd.DataFrame(load_iris().data,
index = ["iris_%d" % i for i in range(load_iris().data.shape[0])],
columns = load_iris().feature_names)
Se_targets = pd.Series(load_iris().target,
index = ["iris_%d" % i for i in range(load_iris().data.shape[0])],
name = "Species")
# Scaling mean = 0, var = 1
DF_standard = pd.DataFrame(StandardScaler().fit_transform(DF_data),
index = DF_data.index,
columns = DF_data.columns)
# Sklearn for Principal Componenet Analysis
# Dims
m = DF_standard.shape[1]
K = 2
# PCA (How I tend to set it up)
Mod_PCA = decomposition.PCA(n_components=m)
DF_PCA = pd.DataFrame(Mod_PCA.fit_transform(DF_standard),
columns=["PC%d" % k for k in range(1,m + 1)]).iloc[:,:K]
# Color classes
color_list = [{0:"r",1:"g",2:"b"}[x] for x in Se_targets]
fig, ax = plt.subplots()
ax.scatter(x=DF_PCA["PC1"], y=DF_PCA["PC2"], color=color_list)
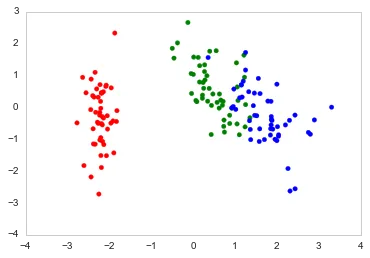

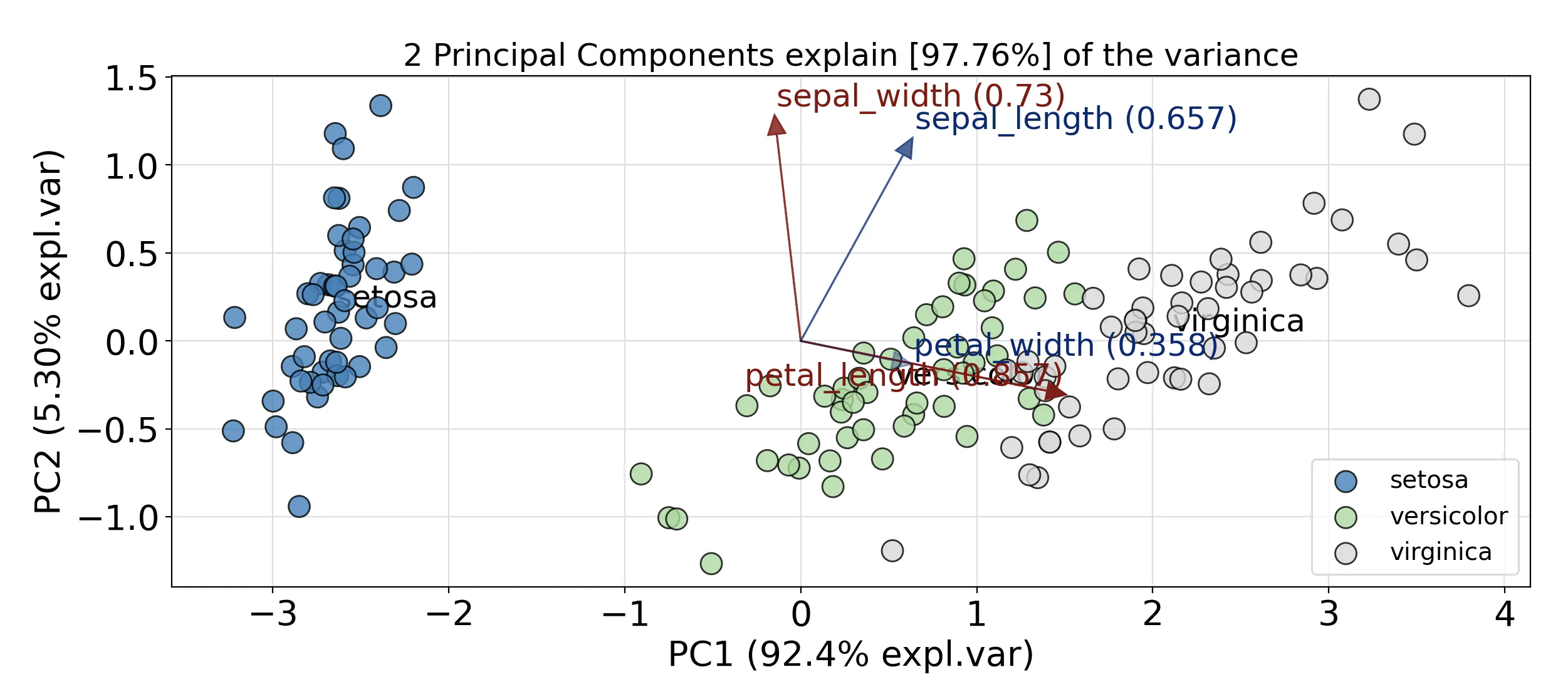
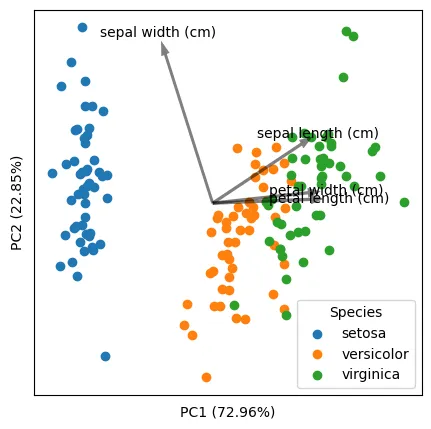
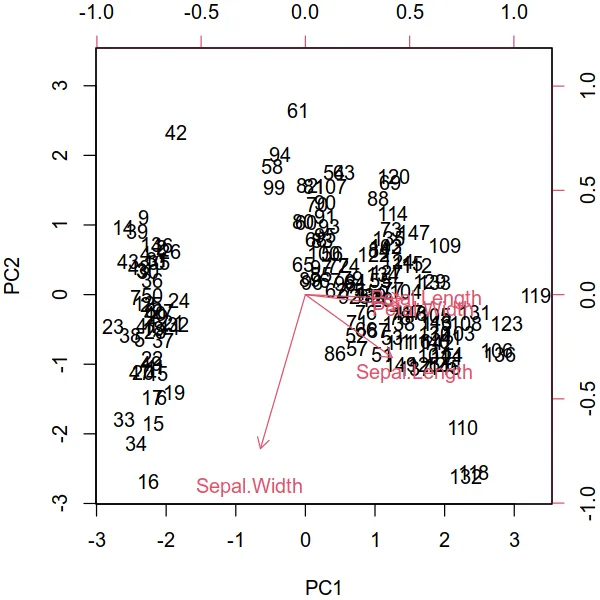
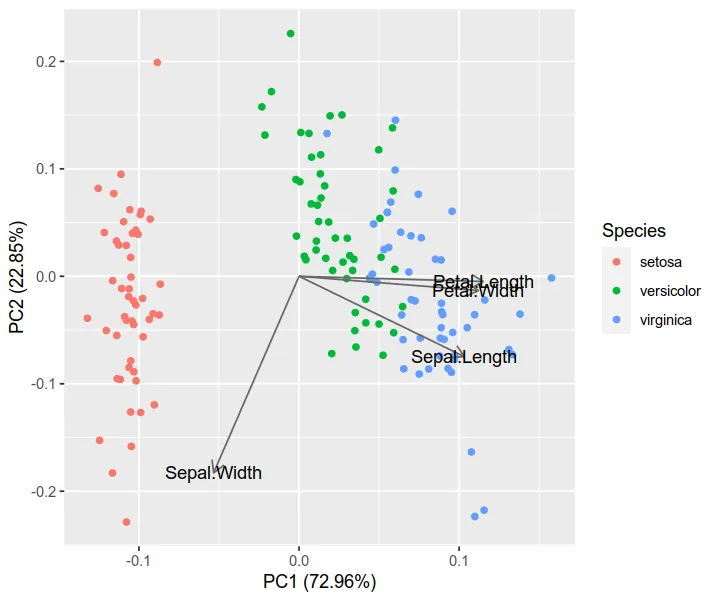
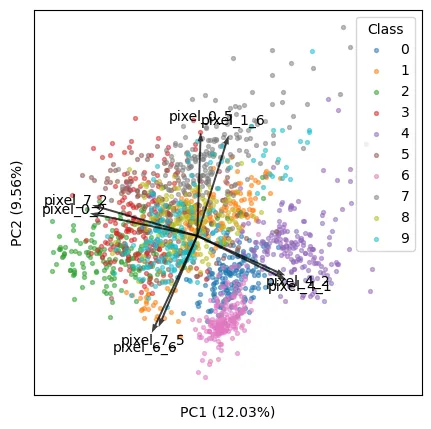
score代表原始数据的降维坐标(在 PC1 和 PC2 中),而coeff代表每个特征的特征向量。这是正确的吗? - Sos