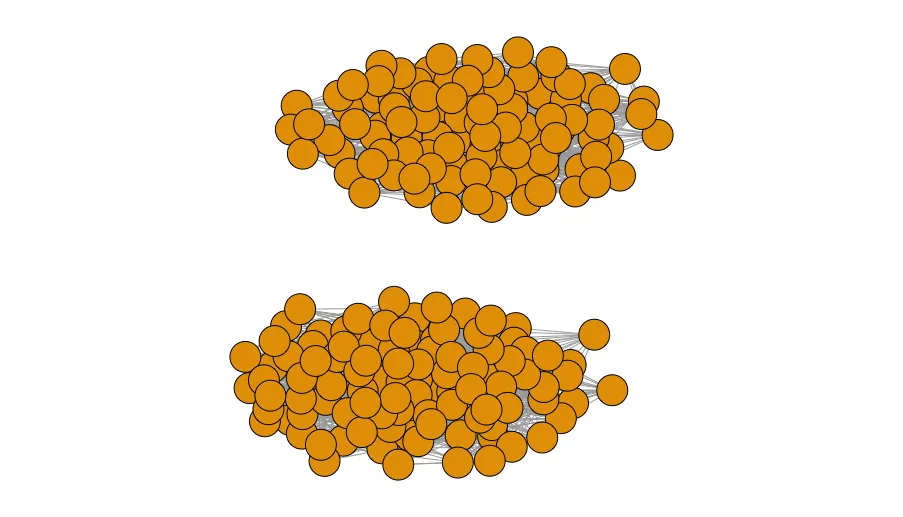
在布局功能中可能有一种简单的方法来实现这个,但你也可以在创建布局后直接更改节点坐标。如果你看一下coords,你会发现它只是一个节点坐标矩阵。你可以使用集群标签来编程地将两个节点集聚在一起:
require(igraph)
require(dplyr)
er_graph <- erdos.renyi.game(100, 5/20)+erdos.renyi.game(100, 5/20)
set.seed(40)
coords <- layout.fruchterman.reingold(er_graph)
plot(er_graph,layout=coords, vertex.label=NA)
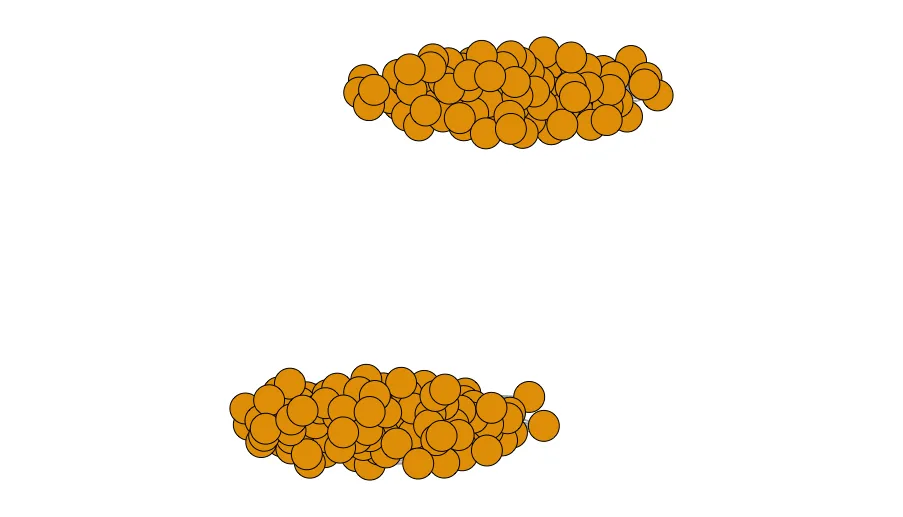
将聚类点靠近一起:首先,我们将聚类标签添加到坐标上,并设置参数f来规定我们希望消除的聚类之间距离的百分比。然后,我们从每个节点中减去f倍该聚类的平均坐标与两个聚类的平均坐标之间差值的乘积。
coords = data.frame(coords, clust=clusters(er_graph)$membership)
f = 0.6
coords = coords %>%
mutate(X1 = ifelse(clust==1, X1 - f*(mean(X1[clust==1]) - mean(X1)), X1 - f*(mean(X1[clust==2]) - mean(X1))),
X2 = ifelse(clust==1, X2 - f*(mean(X2[clust==1]) - mean(X2)), X2 - f*(mean(X2[clust==2]) - mean(X2))))
coords = as.matrix(coords[,1:2])
plot(er_graph,layout=coords, vertex.label=NA)