我正在使用来自sklearn的LaasoCV选择最佳模型以进行交叉验证。我发现,如果我使用sklearn或matlab统计工具箱,交叉验证会给出不同的结果。
我使用了matlab并复制了http://www.mathworks.se/help/stats/lasso-and-elastic-net.html中提供的示例来获得如此的图形: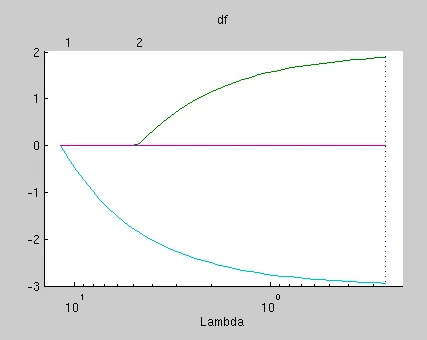 然后我保存了matlab数据,并尝试使用sklearn的laaso_path来复制该图形,我得到了:
然后我保存了matlab数据,并尝试使用sklearn的laaso_path来复制该图形,我得到了:
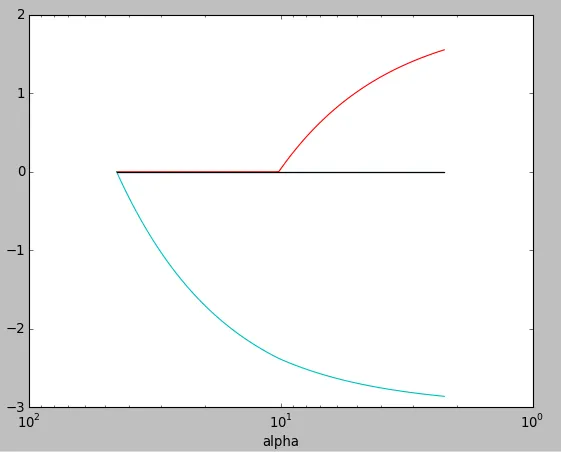 尽管这两个图形之间存在某些相似之处,但也存在某些差异。据我所知,matlab中的lambda参数和sklearn中的alpha参数是相同的,但在这个图中似乎存在一些差异。有人能指出哪一个正确还是我忽略了某些内容?此外,获得的系数也是不同的(这是我的主要关注点)。
尽管这两个图形之间存在某些相似之处,但也存在某些差异。据我所知,matlab中的lambda参数和sklearn中的alpha参数是相同的,但在这个图中似乎存在一些差异。有人能指出哪一个正确还是我忽略了某些内容?此外,获得的系数也是不同的(这是我的主要关注点)。
Matlab代码:
我使用了matlab并复制了http://www.mathworks.se/help/stats/lasso-and-elastic-net.html中提供的示例来获得如此的图形:
 然后我保存了matlab数据,并尝试使用sklearn的laaso_path来复制该图形,我得到了:
然后我保存了matlab数据,并尝试使用sklearn的laaso_path来复制该图形,我得到了:
 尽管这两个图形之间存在某些相似之处,但也存在某些差异。据我所知,matlab中的lambda参数和sklearn中的alpha参数是相同的,但在这个图中似乎存在一些差异。有人能指出哪一个正确还是我忽略了某些内容?此外,获得的系数也是不同的(这是我的主要关注点)。
尽管这两个图形之间存在某些相似之处,但也存在某些差异。据我所知,matlab中的lambda参数和sklearn中的alpha参数是相同的,但在这个图中似乎存在一些差异。有人能指出哪一个正确还是我忽略了某些内容?此外,获得的系数也是不同的(这是我的主要关注点)。Matlab代码:
rng(3,'twister') % for reproducibility
X = zeros(200,5);
for ii = 1:5
X(:,ii) = exprnd(ii,200,1);
end
r = [0;2;0;-3;0];
Y = X*r + randn(200,1)*.1;
save randomData.mat % To be used in python code
[b fitinfo] = lasso(X,Y,'cv',10);
lassoPlot(b,fitinfo,'plottype','lambda','xscale','log');
disp('Lambda with min MSE')
fitinfo.LambdaMinMSE
disp('Lambda with 1SE')
fitinfo.Lambda1SE
disp('Quality of Fit')
lambdaindex = fitinfo.Index1SE;
fitinfo.MSE(lambdaindex)
disp('Number of non zero predictos')
fitinfo.DF(lambdaindex)
disp('Coefficient of fit at that lambda')
b(:,lambdaindex)
Python 代码:
import scipy.io
import numpy as np
import pylab as pl
from sklearn.linear_model import lasso_path, LassoCV
data=scipy.io.loadmat('randomData.mat')
X=data['X']
Y=data['Y'].flatten()
model = LassoCV(cv=10,max_iter=1000).fit(X, Y)
print 'alpha', model.alpha_
print 'coef', model.coef_
eps = 1e-2 # the smaller it is the longer is the path
models = lasso_path(X, Y, eps=eps)
alphas_lasso = np.array([model.alpha for model in models])
coefs_lasso = np.array([model.coef_ for model in models])
pl.figure(1)
ax = pl.gca()
ax.set_color_cycle(2 * ['b', 'r', 'g', 'c', 'k'])
l1 = pl.semilogx(alphas_lasso,coefs_lasso)
pl.gca().invert_xaxis()
pl.xlabel('alpha')
pl.show()