欢迎更新基准测试!
df <- data.frame(
ID1 = rep(LETTERS, each = 10000),
ID2 = sample(1000, length(LETTERS) * 10000, replace = TRUE)
)
f_TIC1 <- function() {
lst <- split(df, ~ID1)
lst[[1]] <- lst[[1]][sample(1:nrow(lst[[1]]), 1), ]
Reduce(
function(x, y) {
y <- subset(y, !ID2 %in% x$ID2)
rbind(x, y[sample(nrow(y), 1), ])
},
lst
)
}
library(igraph)
library(dplyr)
f_TIC2 <- function() {
g <- df %>%
arrange(sample(n())) %>%
graph_from_data_frame() %>%
set_vertex_attr(
name = "type",
value = names(V(.)) %in% df$ID1
)
type.convert(
setNames(
rev(
stack(
max_bipartite_match(g)$matching[unique(df$ID1)]
)
), names(df)
),
as.is = TRUE
)
}
f_TIC3 <- function() {
lst <- with(df, split(ID2, ID1))
v <- c()
for (k in seq_along(lst)) {
u <- lst[[k]][!lst[[k]] %in% v]
v <- c(v, u[sample(length(u), 1)])
}
type.convert(
data.frame(ID1 = names(lst), ID2 = v),
as.is = TRUE
)
}
f_GKi1 <- function() {
. <- split(df$ID2, df$ID1)
data.frame(ID1 = type.convert(names(.), as.is=TRUE),
ID2 = Reduce(function(x, y) {c(x, sample(y[!y %in% x], 1))}, c(list(NULL), .)))
}
f_GKi2 <- function() {
. <- split(df$ID2, df$ID1)
x <- df$ID2[0]
for(y in .) {
y <- y[!y %in% x]
x <- c(x, y[sample.int(length(y),1)])
}
data.frame(ID1 = type.convert(names(.), as.is=TRUE), ID2 = x)
}
library(fastmatch)
library(dqrng)
f_GKi3 <- function() {
. <- split(df$ID2, df$ID1)
x <- df$ID2[0]
for(y in .) {
y <- y[!y %fin% x]
x <- c(x, y[dqsample.int(length(y),1)])
}
data.frame(ID1 = type.convert(names(.), as.is=TRUE), ID2 = x)
}
f_GKi4 <- function() {
. <- split(df$ID2, df$ID1)
x <- vector(typeof(df$ID2), length(.))
for(i in seq_along(.)) {
y <- .[[i]]
y <- y[!y %fin% x[seq_len(i-1)]]
x[i] <- y[dqsample.int(length(y),1)]
}
data.frame(ID1 = type.convert(names(.), as.is=TRUE), ID2 = x)
}
f_Onyambu <- function() {
data <- df[order(df$ID1, df$ID2),]
n <- 1
st <- table(data[[1]])
s <- min(st)
m <- length(st)
size <- min(m*n, s)
samples <- sample(s, size)
index <- rep(seq(s), each = n, length = size) * s - s + samples
data[index, ]
}
bm <- microbenchmark::microbenchmark(
f_TIC1(),
f_TIC2(),
f_TIC3(),
f_GKi1(),
f_GKi2(),
f_GKi3(),
f_GKi4(),
f_Onyambu()
)
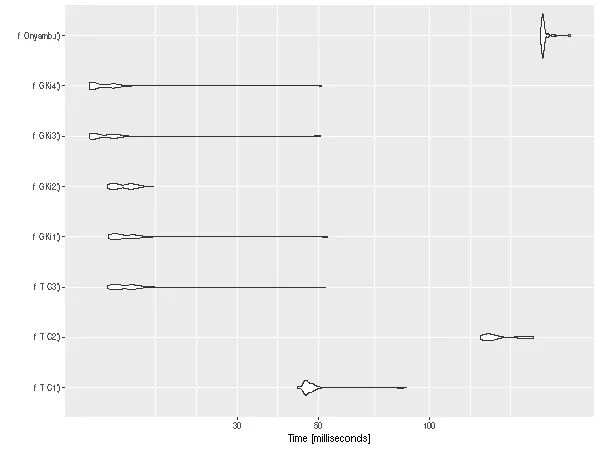
ggplot2::autoplot(bm)
bm
目前最快的是 GKi3 和 GKi4,其次是 TIC3、GKi1 和 GKi2,它们几乎相等,因为它们使用了与 TIC1 相同的逻辑,该逻辑在 GKi1 中进行了优化,并在 TIC3 和 GKi2 中重新使用。

ID1组是否都包含相同的值?或者ID1 == "B"是否可能有不同于1:5的ID2值,例如2:6?如果它们都是相同的,我建议对不重复的值进行采样,然后添加ID1。 - Martin Galc(1, 4:5)中进行抽样,因为2已经被选择并因此被移除。 - tmfmnk