为了完整起见,这里提供了一个解决方案,使用
scipy.stats.pearsonr (
文档)来创建一个p值矩阵。接下来创建一个布尔掩码,传递给seaborn(或者另外与numpy
np.triu结合使用以隐藏相关性的上三角)。
def corr_sig(df=None):
p_matrix = np.zeros(shape=(df.shape[1],df.shape[1]))
for col in df.columns:
for col2 in df.drop(col,axis=1).columns:
_ , p = stats.pearsonr(df[col],df[col2])
p_matrix[df.columns.to_list().index(col),df.columns.to_list().index(col2)] = p
return p_matrix
p_values = corr_sig(df)
mask = np.invert(np.tril(p_values<0.05))
完整过程及示例
首先创建一些样本数据(3个相关变量;3个不相关的变量):
import pandas as pd
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
from scipy import stats
num_samples = 100
mu = np.array([5.0, 0.0, 10.0])
r = np.array([
[ 3.40, -2.75, -2.00],
[ -2.75, 5.50, 1.50],
[ -2.00, 1.50, 1.25]
])
y = np.random.multivariate_normal(mu, r, size=num_samples)
df = pd.DataFrame(y)
df.columns = ["Correlated1","Correlated2","Correlated3"]
for i in range(2):
df.loc[:,f"Uncorrelated{i}"] = np.random.randint(-2000,2000,len(df))
df.loc[:,"Near Invariant"] = np.random.randint(-99,-95,num_samples)
用于方便的绘图函数
主要用于美化热力图。
def plot_cor_matrix(corr, mask=None):
f, ax = plt.subplots(figsize=(11, 9))
sns.heatmap(corr, ax=ax,
mask=mask,
annot=True, vmin=-1, vmax=1, center=0,
cmap='coolwarm', linewidths=2, linecolor='black', cbar_kws={'orientation': 'horizontal'})
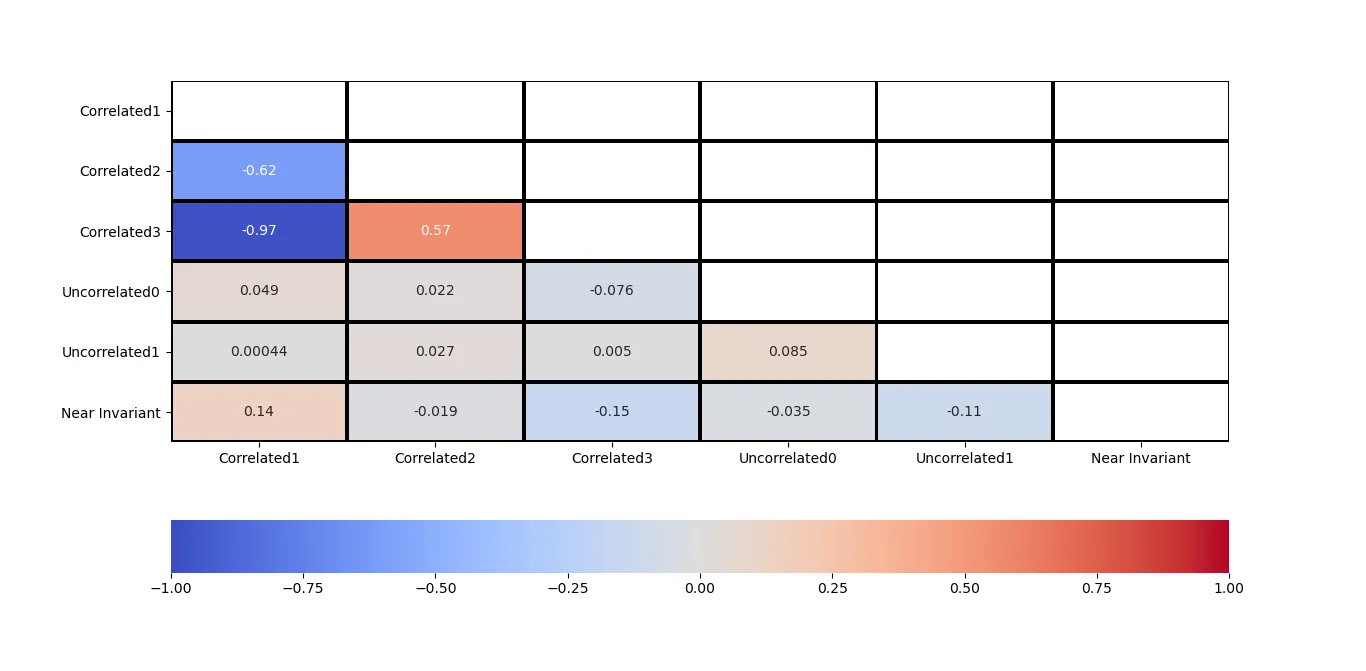
所有相关性的例子数据的相关性图
为了让您了解在此示例相关矩阵中未过滤显著相关性(p值<0.05)的情况下相关性的外观。
# Plotting without significance filtering
corr = df.corr()
mask = np.triu(corr)
plot_cor_matrix(corr,mask)
plt.show()
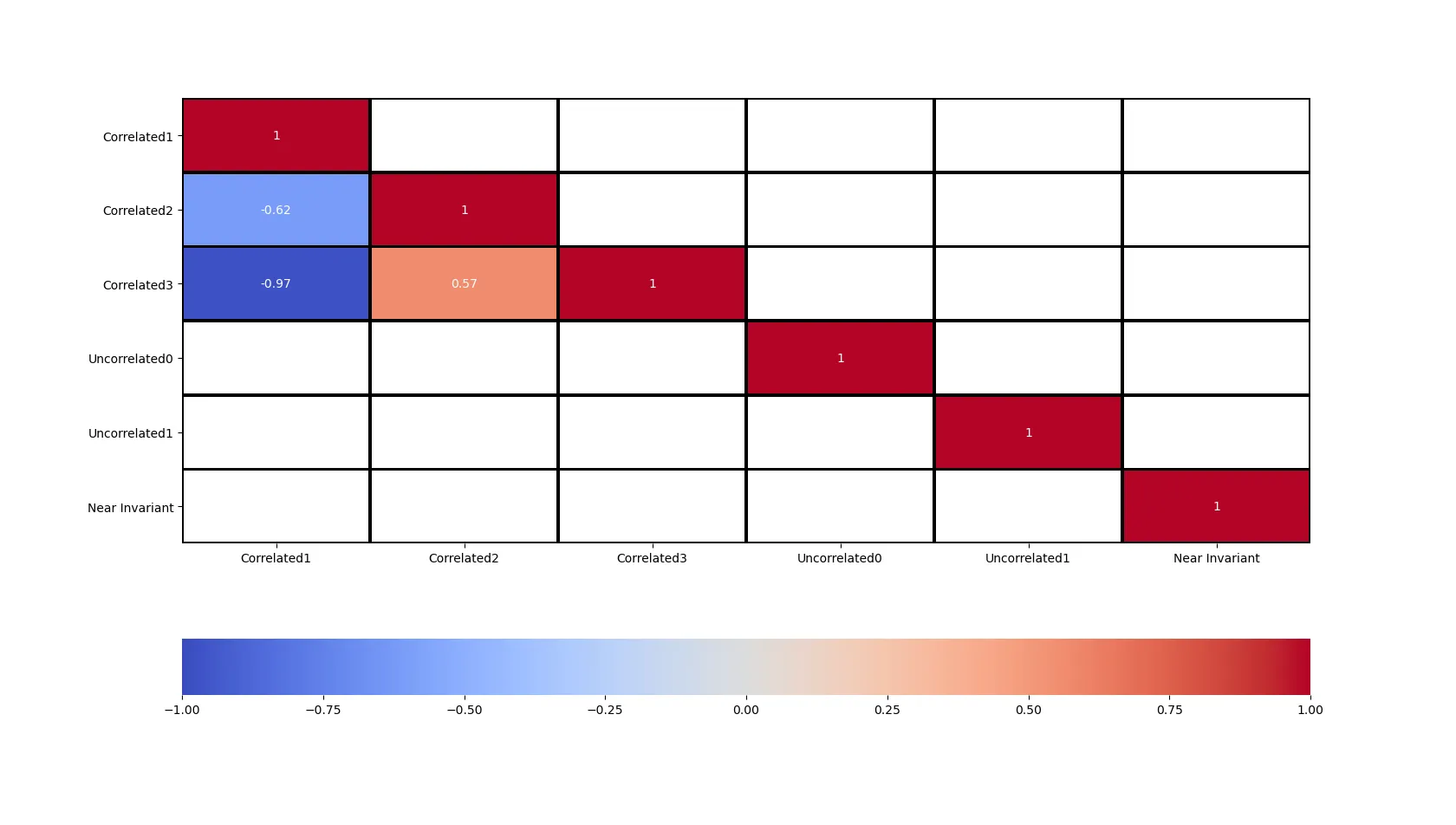
只显示显著相关性的示例数据相关图
最终绘制的图仅包含显著性水平小于0.05的相关系数。
corr = df.corr()
p_values = corr_sig(df)
mask = np.invert(np.tril(p_values<0.05))
plot_cor_matrix(corr,mask)
结论
虽然在第一个相关矩阵中有一些相关系数 (r) 大于 0.05 (根据 OP 提问中的评论进行过滤),这并不意味着 p 值是显著的。因此,重要的是区分 p 值和相关系数 r。
我希望这个答案将来对其他人寻找使用 sns.heatmap 绘制显著相关性的方法有所帮助。


a = result.corr(),那么heatmap(a, mask=mask & (a>0.05))? - ImportanceOfBeingErnestmask | (a > 0.05)?由于没有提供最小可复现示例,我无法在此处测试任何内容。 - ImportanceOfBeingErnest.corr()只给出了相关系数,而没有p值(从针对零的统计测试中获得)。 - Björn