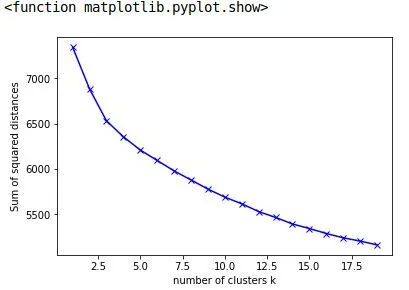
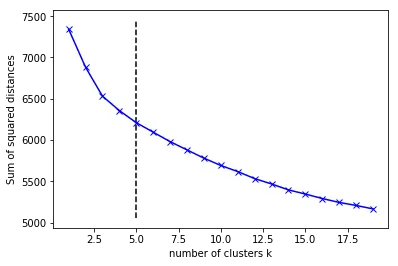
对于所有想要自行实现的人,这里提供一个简单的基本实现。它非常适合我的用例(200个集群作为计算边界),距离计算非常基础,基于2D空间中点到点的计算,但可以适应任何其他数字量。
我认为Kevin的库在技术上更加更新和更好地实现了。
import KMeansClusterer
from math import sqrt, fabs
from matplotlib import pyplot as plp
import multiprocessing as mp
import numpy as np
class ClusterCalculator:
m = 0
b = 0
sum_squared_dist = []
derivates = []
distances = []
line_coordinates = []
def __init__(self, calc_border, data):
self.calc_border = calc_border
self.data = data
def calculate_optimum_clusters(self, option_parser):
if(option_parser.multiProcessing):
self.calc_mp()
else:
self.calculate_squared_dist()
self.init_opt_line()
self.calc_distances()
self.calc_line_coordinates()
opt_clusters = self.get_optimum_clusters()
print("Evaluated", opt_clusters, "as optimum number of clusters")
self.plot_results()
return opt_clusters
def calculate_squared_dist(self):
for k in range(1, self.calc_border):
print("Calculating",k, "of", self.calc_border, "\n", (self.calc_border - k), "to go!")
kmeans = KMeansClusterer.KMeansClusterer(k, self.data)
ine = kmeans.calc_custom_params(self.data, k).inertia_
print("inertia in round", k, ": ", ine)
self.sum_squared_dist.append(ine)
def init_opt_line(self):
self. m = (self.sum_squared_dist[0] - self.sum_squared_dist[-1]) / (1 - self.calc_border)
self.b = (1 * self.sum_squared_dist[0] - self.calc_border*self.sum_squared_dist[0]) / (1 - self.calc_border)
def calc_y_value(self, x_calc):
return self.m * x_calc + self.b
def calc_line_coordinates(self):
for i in range(0, len(self.sum_squared_dist)):
self.line_coordinates.append(self.calc_y_value(i))
def calc_distances(self):
for i in range(0, self.calc_border):
y_value = self.calc_y_value(i)
d = sqrt(fabs(self.sum_squared_dist[i] - self.calc_y_value(i)))
length_list = len(self.sum_squared_dist)
self.distances.append(sqrt(fabs(self.sum_squared_dist[i] - self.calc_y_value(i))))
print("For border", self.calc_border, ", calculated the following distances: \n", self.distances)
def get_optimum_clusters(self):
return self.distances.index((max(self.distances)))
def plot_results(self):
plp.plot(range(0, self.calc_border), self.sum_squared_dist, "bx-")
plp.plot(range(0, self.calc_border), self.line_coordinates, "bx-")
plp.xlabel("Number of clusters")
plp.ylabel("Sum of squared distances")
plp.show()
def calculate_squared_dist_sliced_data(self,output, proc_numb, start, end):
temp = []
for k in range(start, end + 1):
kmeans = KMeansClusterer.KMeansClusterer(k, self.data)
ine = kmeans.calc_custom_params(self.data, k).inertia_
print("Process", proc_numb,"had the CPU,", "calculated", ine, "in round", k)
temp.append(ine)
output.put((proc_numb, temp))
def sort_result_queue(self, result):
result.sort()
result = [r[1] for r in result]
flat_list= [item for sl in result for item in sl]
return flat_list
def calc_mp(self):
output = mp.Queue()
processes = []
processes.append(mp.Process(target=self.calculate_squared_dist_sliced_data, args=(output, 1, 1, 50)))
processes.append(mp.Process(target=self.calculate_squared_dist_sliced_data, args=(output, 2, 51, 100)))
processes.append(mp.Process(target=self.calculate_squared_dist_sliced_data, args=(output, 3, 101, 150)))
processes.append(mp.Process(target=self.calculate_squared_dist_sliced_data, args=(output, 4, 151, 200)))
for p in processes:
p.start()
for p in processes:
p.join()
results = [output.get() for p in processes]
self.sum_squared_dist = self.sort_result_queue(results)