很明显,
a = a[a[:, 0].argsort()] 是所有竞争分组算法的瓶颈,感谢
Vincent J 澄清了这一点。超过80%的处理时间都花费在了这个
argsort 方法上,没有简单的方法可以替换或优化它。
numba 包可以加速许多算法,希望未来有人能对
argsort 进行改进。假设第一列的索引很小,那么分组的
剩余部分可以显著改进。
TL;DR
大多数分组方法的剩余部分包含 np.unique 方法,当组的值很小时,它相当缓慢和冗余。用 np.bincount 替换它更有效率,后者可以在 numba 中进一步改进。以下是一些关于如何改进剩余部分的结果:
def _custom_return(unique_id, a, split_idx, return_groups):
'''Choose if you want to also return unique ids'''
if return_groups:
return unique_id, np.split(a[:,1], split_idx)
else:
return np.split(a[:,1], split_idx)
def numpy_groupby_index(a, return_groups=False):
'''Code refactor of method of Vincent J'''
u, idx = np.unique(a[:,0], return_index=True)
return _custom_return(u, a, idx[1:], return_groups)
def numpy_groupby_counts(a, return_groups=False):
'''Use cumsum of counts instead of index'''
u, counts = np.unique(a[:,0], return_counts=True)
idx = np.cumsum(counts)
return _custom_return(u, a, idx[:-1], return_groups)
def numpy_groupby_diff(a, return_groups=False):
'''No use of any np.unique options'''
u = np.unique(a[:,0])
idx = np.flatnonzero(np.diff(a[:,0])) + 1
return _custom_return(u, a, idx, return_groups)
def numpy_groupby_bins(a, return_groups=False):
'''Replace np.unique by np.bincount'''
bins = np.bincount(a[:,0])
nonzero_bins_idx = bins != 0
nonzero_bins = bins[nonzero_bins_idx]
idx = np.cumsum(nonzero_bins[:-1])
return _custom_return(np.flatnonzero(nonzero_bins_idx), a, idx, return_groups)
def numba_groupby_bins(a, return_groups=False):
'''Replace np.bincount by numba_bincount'''
bins = numba_bincount(a[:,0])
nonzero_bins_idx = bins != 0
nonzero_bins = bins[nonzero_bins_idx]
idx = np.cumsum(nonzero_bins[:-1])
return _custom_return(np.flatnonzero(nonzero_bins_idx), a, idx, return_groups)
所以numba_bincount的工作方式与np.bincount相同,它的定义如下:
from numba import njit
@njit
def _numba_bincount(a, counts, m):
for i in range(m):
counts[a[i]] += 1
def numba_bincount(arr):
M = np.max(arr)
counts = np.zeros(M + 1, dtype=int)
_numba_bincount(arr, counts, len(arr))
return counts
使用方法:
a = np.array([[1,275],[1,441],[1,494],[1,593],[2,679],[2,533],[2,686],[3,559],[3,219],[3,455],[4,605],[4,468],[4,692],[4,613]])
a = a[a[:, 0].argsort()]
>>> numpy_groupby_index(a, return_groups=False)
[array([275, 441, 494, 593]),
array([679, 533, 686]),
array([559, 219, 455]),
array([605, 468, 692, 613])]
>>> numpy_groupby_index(a, return_groups=True)
(array([1, 2, 3, 4]),
[array([275, 441, 494, 593]),
array([679, 533, 686]),
array([559, 219, 455]),
array([605, 468, 692, 613])])
性能测试
在我的电脑上(有10个不同的ID),对1亿个项目进行排序需要约30秒。现在,让我们测试剩余部分中的方法运行需要多长时间:
%matplotlib inline
benchit.setparams(rep=3)
sizes = [3*10**(i//2) if i%2 else 10**(i//2) for i in range(17)]
N = sizes[-1]
x1 = np.random.randint(0,10, size=N)
x2 = np.random.normal(loc=500, scale=200, size=N).astype(int)
a = np.transpose([x1, x2])
arr = a[a[:, 0].argsort()]
fns = [numpy_groupby_index, numpy_groupby_counts, numpy_groupby_diff, numpy_groupby_bins, numba_groupby_bins]
in_ = {s/1000000: (arr[:s], ) for s in sizes}
t = benchit.timings(fns, in_, multivar=True, input_name='Millions of events')
t.plot(logx=True, figsize=(12, 6), fontsize=14)
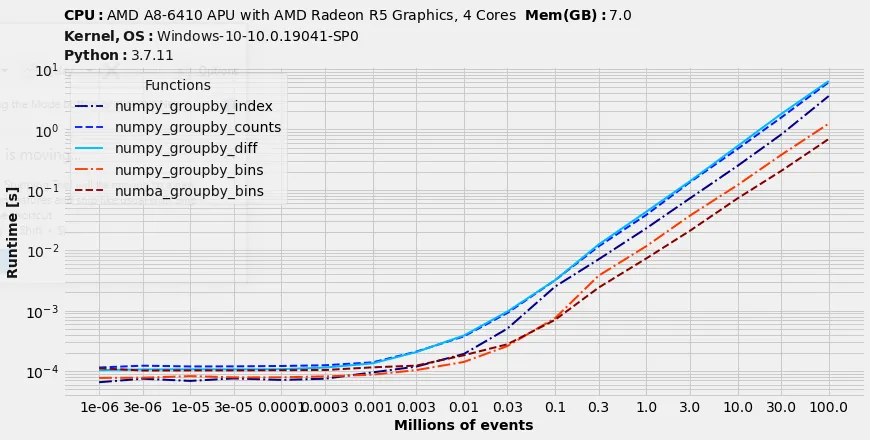
毫无疑问,使用numba提供的bincount函数是处理包含小ID数据集的最佳选择。它可以将排序后的数据分组减少约5倍,相当于总运行时间的10%。

a = a[a.T[0,:].argsort()]代替。 - Behzad Shayegha = a[a[:, 0].argsort()])是一个问题。首先,它是O(n log n)的。其次,它消耗了80%的执行时间。numpy或numba的开发人员不太可能很快对np.argsort进行优化。因此,只能加速剩下的20%。进一步优化的主要方向是用np.bincount替换np.unique,如果在numba中重新设计,可以进一步改进。注意:bincount不适用于大ID。 - mathfuxnp.split,因为它会创建一个空的 Pythoniclist并在每个 numpyic 数组切片上调用list.append(在底层实现中)。 - mathfux