我有一个输入文件,其中包含大约50000个聚类以及每个聚类中存在的若干因素(总共约1000万条记录),以下是一个更小的示例:
set.seed(1)
x = paste("cluster-",sample(c(1:100),500,replace=TRUE),sep="")
y = c(
paste("factor-",sample(c(letters[1:3]),300, replace=TRUE),sep=""),
paste("factor-",sample(c(letters[1]),100, replace=TRUE),sep=""),
paste("factor-",sample(c(letters[2]),50, replace=TRUE),sep=""),
paste("factor-",sample(c(letters[3]),50, replace=TRUE),sep="")
)
data = data.frame(cluster=x,factor=y)
在另一个问题的帮助下,我设法生成了如下所示的因素共现饼图:
counts = with(data, table(tapply(factor, cluster, function(x) paste(as.character(sort(unique(x))), collapse='+'))))
pie(counts[counts>1])
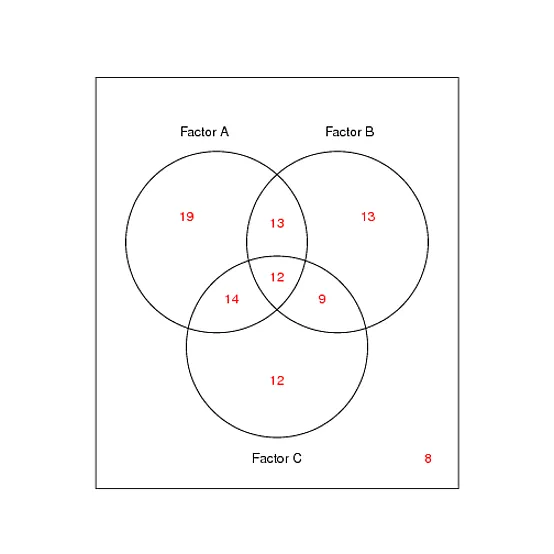
但现在我想要一个显示因子共现的维恩图。最好可以设置每个因子的最小计数阈值。例如,对于不同因子的维恩图,每个因子在每个群集中都必须出现n>10次才能被考虑。
我尝试使用aggregate生成计数表,但无法使其工作。
venneuler库的这个例子,或者是在《统计软件杂志》上使用venn库的简短文章(Murdoch, 2004)。如果这只是关于R编程,那么应该迁移到SO。 - Andy W