我正在使用
数据(响应
glmnet R软件包进行岭回归。我发现,通过glmnet :: glmnet函数获得的系数与我使用相同lambda值计算定义系数时获得的系数不同。有人可以解释一下为什么吗?数据(响应
Y和设计矩阵X)都已缩放。library(MASS)
library(glmnet)
# Data dimensions
p.tmp <- 100
n.tmp <- 100
# Data objects
set.seed(1)
X <- scale(mvrnorm(n.tmp, mu = rep(0, p.tmp), Sigma = diag(p.tmp)))
beta <- rep(0, p.tmp)
beta[sample(1:p.tmp, 10, replace = FALSE)] <- 10
Y.true <- X %*% beta
Y <- scale(Y.true + matrix(rnorm(n.tmp))) # Y.true + Gaussian noise
# Run glmnet
ridge.fit.cv <- cv.glmnet(X, Y, alpha = 0)
ridge.fit.lambda <- ridge.fit.cv$lambda.1se
# Extract coefficient values for lambda.1se (without intercept)
ridge.coef <- (coef(ridge.fit.cv, s = ridge.fit.lambda))[2:(p.tmp+1)]
# Get coefficients "by definition"
ridge.coef.DEF <- solve(t(X) %*% X + ridge.fit.lambda * diag(p.tmp)) %*% t(X) %*% Y
# Plot estimates
plot(ridge.coef, type = "l", ylim = range(c(ridge.coef, ridge.coef.DEF)),
main = "black: Ridge `glmnet`\nred: Ridge by definition")
lines(ridge.coef.DEF, col = "red")
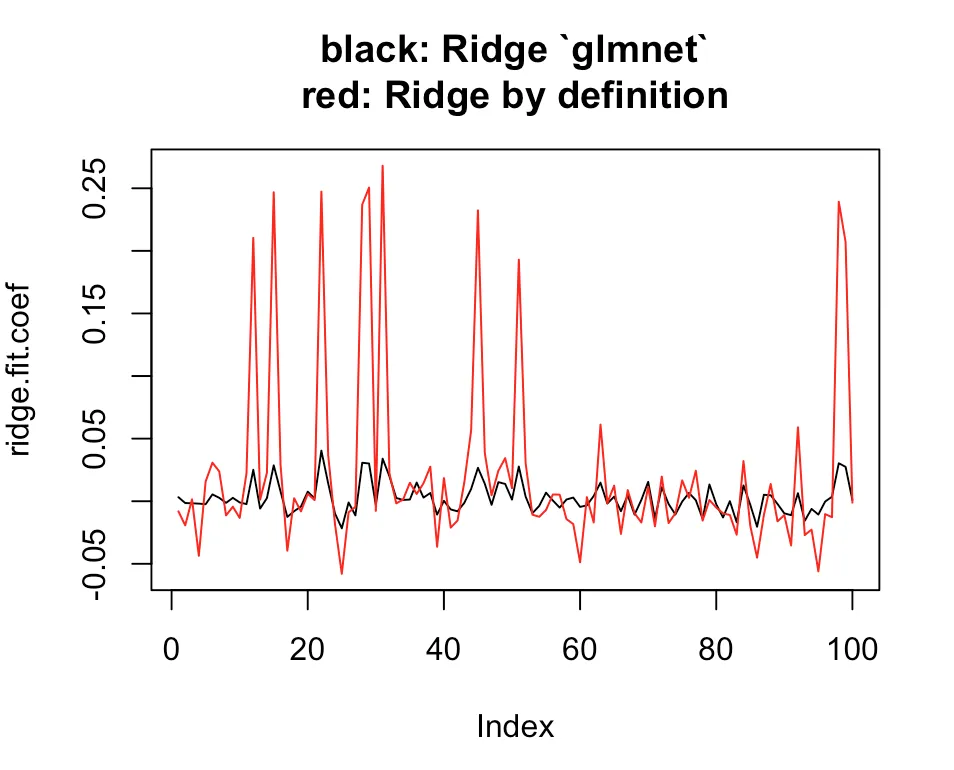
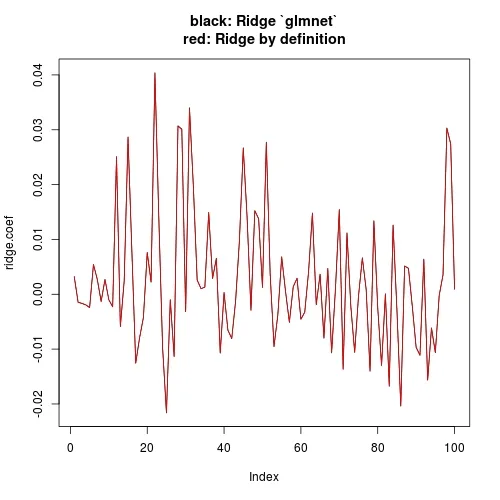
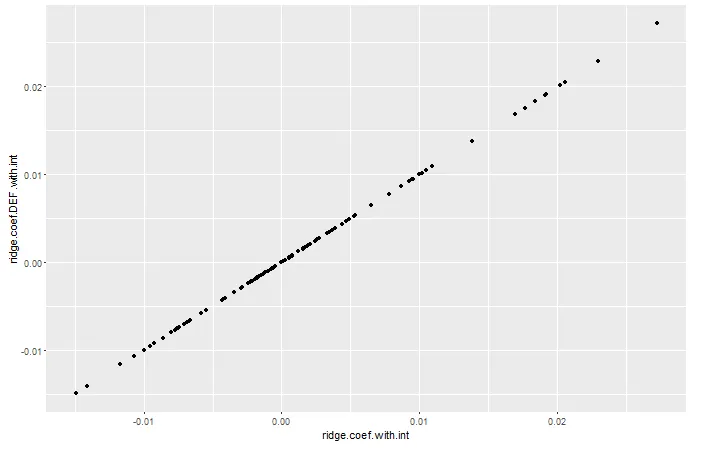
glmnet函数对于给定的lambda(惩罚项)值返回的系数与直接使用相同的lambda值解决回归系数得到的系数不一样。 - eipi10100*ridge.fit.lambda进行“手动”计算,其结果与使用solve(t(X) %*% X + 100*ridge.fit.lambda * diag(p.tmp)) %*% t(X) %*% Y和glmnet中的ridge.fit.lambda得到的系数(几乎)完全相同。 - eipi10