在(线性回归)模型中,应该有一种方法来报告系数与总体平均数(在多元情况下,“所有其他条件相等”)之间的差异,并为每个系数得到一个: $ \beta_{North} = 40 $ $ \beta_{Central} = -10 $ $ \beta_{South} = -30 $
显然跳过截距。
这对我来说似乎最直观。但是,我无论如何都想不出如何使用R的“对比编码”来做到这一点。(而且,这似乎会搞乱变量名)。
设置我的模拟/ mwe的参数
m_inc <- 90
b_n <- 40
b_c <- -10
b_s <- -30
sd_prop <- 0.5 #sd as share of mean
pop_per <- 1000
模拟数据
set.seed(100)
n_income <- rnorm(pop_per, m_inc + b_n, (m_inc + b_n)*sd_prop)
c_income <- rnorm(pop_per, m_inc + b_c, (m_inc + b_s)*sd_prop)
s_income <- rnorm(pop_per, m_inc + b_s, (m_inc + b_s)*sd_prop)
noise_var <- rnorm(pop_per*3, 0, (m_inc + b_s)*sd_prop)
i_df <- tibble(
region = rep( c("n", "c", "s"), c(pop_per, pop_per, pop_per) ),
income = c(n_income, c_income, s_income),
noise_var
) %>%
mutate(region = as.factor(region))
i_df %>% # Summary by group using purrr
split(.$region) %>%
purrr::map(summary)
看起来已经足够接近了。
现在我想要“建立收入模型”以便控制其他因素并通过地区比较差异。为了说明这个问题,让我们将南部设为基准组。我设置了默认的contr.treatment,以防您对其进行了重置。
i_df <- i_df %>% mutate(region = relevel(region, ref="s"))
options(contrasts = rep ("contr.treatment", 2))
(
basic_lm <- i_df %>% lm(income ~ region + noise_var, .)
)
标准做法:拦截器(intercept)是(大致上)“基础组”南方的均值,而系数regionc和regionn则分别表示这些地区的相对调整,大约为+20和+70。
这是标准的“虚拟编码”或“处理编码”,在R中是默认设置。
我们可以将此默认设置(针对无序变量)调整为称为“总和对比编码”的东西,适用于无序和有序变量。
options(contrasts = rep ("contr.sum", 2))
(
basic_lm_cc <- i_df %>% lm(income ~ region + noise_var, .)
)
现在看起来我们得到了所需的调整系数,但是:
- 地区名称丢失了;我怎么知道哪个是哪个?
- 显然报告的是 s(南部)和 c(中部)的调整系数。不太直观。
无论如何重新设置地区以设置特定基本组(我尝试过)... 系数都不会改变。
我找到了一个解决方案,但这不是“正确的方法”。我让结果(收入)变量减去平均值,并强制截距为0:
i_df %>%
mutate(m_inc = mean(income)) %>%
lm(income - m_inc ~ 0 + region + noise_var, .)
太好了!这正是我想要的,而且变量名也奇迹般地保留下来了。但这似乎是一种奇怪的方法。还要注意,使用上面的代码,无论是求和对比矩阵还是处理对比矩阵,都将出现这组系数。
如何使用对比编码或其他工具以“正确”的方式完成此操作?

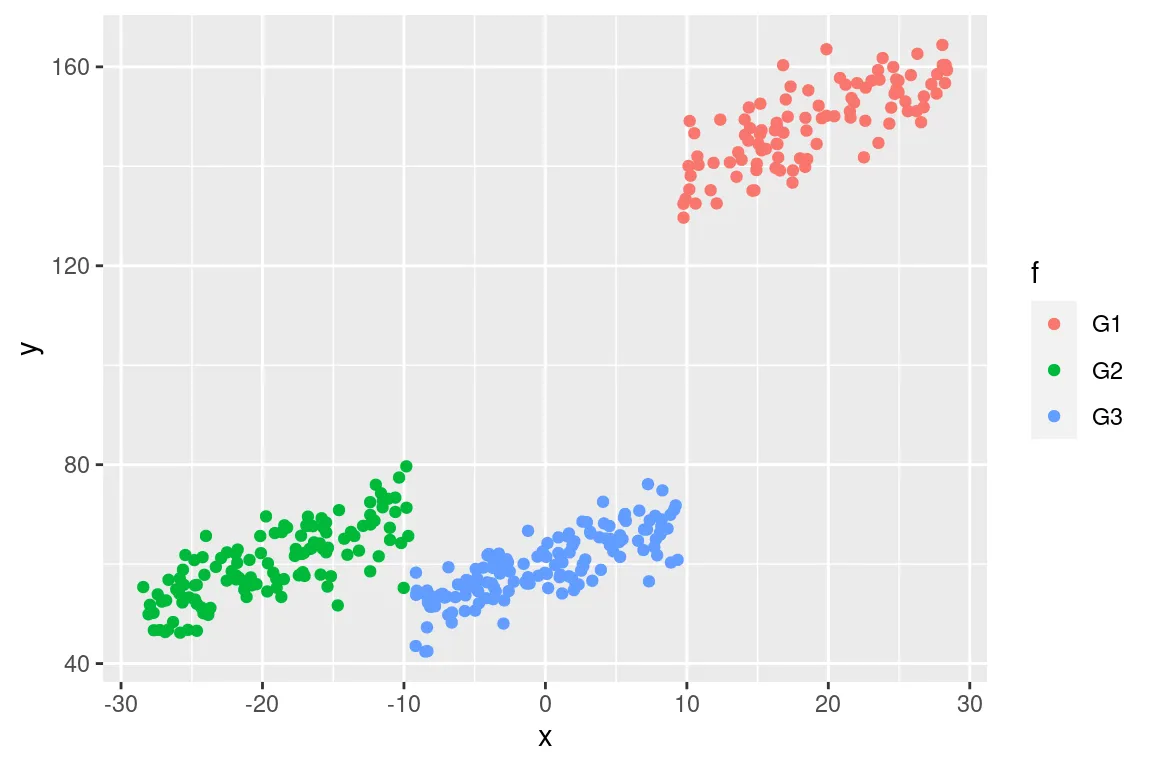


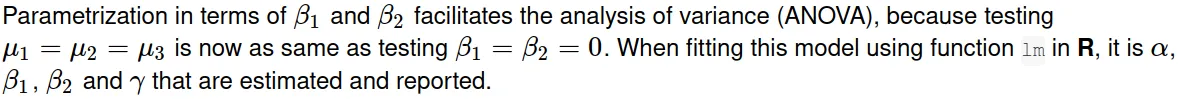



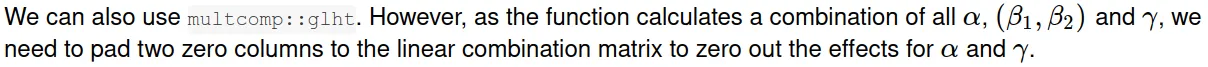

emmeans::emmeans(basic_lm, ~region, offset=-mean(i_df$income))给出了正确的值,但是标准误差却不对。 - Ben Bolker