发布前,我进行了大量搜索并找到了这个问题,可能正是我的问题所在。然而,我尝试了答案中提出的方法,但不幸的是,它没有解决我的问题,由于我是一个新成员,无法添加评论以请求进一步解释。
无论如何,我想在Python中使用scikit-learn中的高斯过程,在一个简单但真实的案例中开始(使用scikit-learn文档中提供的示例)。我有一个名为X的2D输入集(8对2个参数),我有8个相应的输出,收集在1D数组y中。
我定义了一个输入测试空间x:
这是我的结果:
无论如何,我想在Python中使用scikit-learn中的高斯过程,在一个简单但真实的案例中开始(使用scikit-learn文档中提供的示例)。我有一个名为X的2D输入集(8对2个参数),我有8个相应的输出,收集在1D数组y中。
# Inputs: 8 points
X = np.array([[p1, q1],[p2, q2],[p3, q3],[p4, q4],[p5, q5],[p6, q6],[p7, q7],[p8, q8]])
# Observations: 8 couples
y = np.array([r1,r2,r3,r4,r5,r6,r7,r8])
我定义了一个输入测试空间x:
# Input space
x1 = np.linspace(x1min, x1max) #p
x2 = np.linspace(x2min, x2max) #q
x = (np.array([x1, x2])).T
然后我实例化了GP模型,将其拟合到我的训练数据(X,y)上,并在我的输入空间x上进行1D预测y_pred:
from sklearn.gaussian_process import GaussianProcessRegressor
from sklearn.gaussian_process.kernels import RBF, ConstantKernel as C
kernel = C(1.0, (1e-3, 1e3)) * RBF([5,5], (1e-2, 1e2))
gp = GaussianProcessRegressor(kernel=kernel, n_restarts_optimizer=15)
gp.fit(X, y)
y_pred, MSE = gp.predict(x, return_std=True)
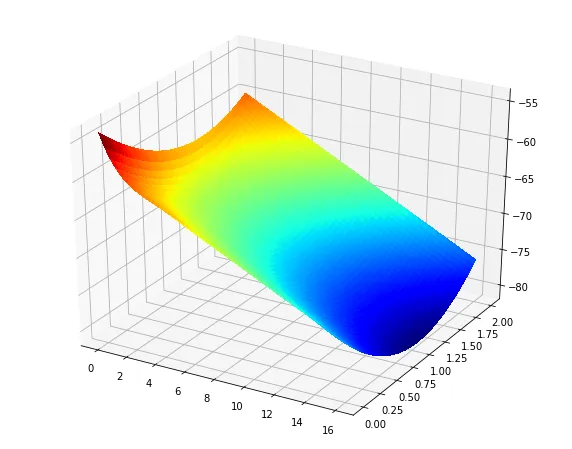
然后我制作了一个三维图:
fig = pl.figure()
ax = fig.add_subplot(111, projection='3d')
Xp, Yp = np.meshgrid(x1, x2)
Zp = np.reshape(y_pred,50)
surf = ax.plot_surface(Xp, Yp, Zp, rstride=1, cstride=1, cmap=cm.jet,
linewidth=0, antialiased=False)
pl.show()
这是我的结果:
我得到的结果如下:
当我修改内核参数时,我得到了类似于上面提到的海报的结果:
这些图甚至不能与原始训练点的观察结果匹配(下方响应在[65.1,37]处获得,最高在[92.3,54]处获得)。
我对2D GPs还比较新(也不久开始使用Python),所以我认为我在这里遗漏了什么......任何答案都将有帮助并且非常感激,谢谢!
![RBF[5,5]](https://istack.dev59.com/0I6Eb.webp)
![RBF[10,10]](https://istack.dev59.com/3q7vn.webp)