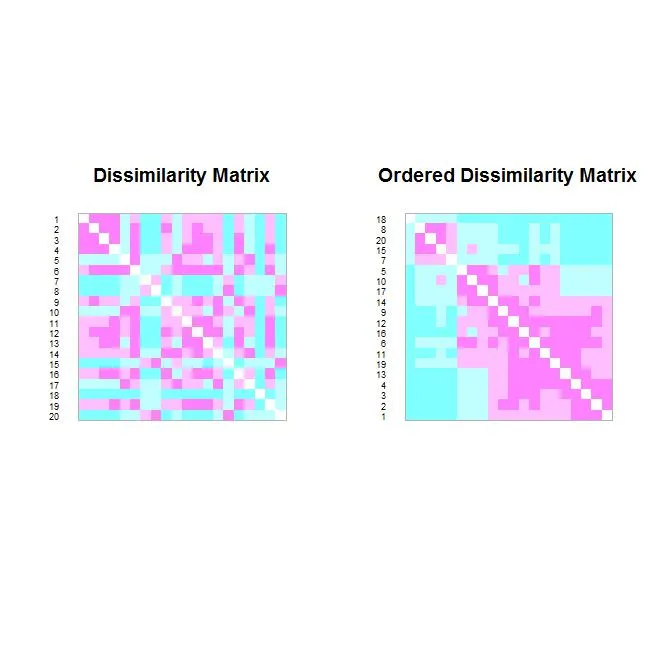
我希望在我写的一篇文章中展示一个距离矩阵,我正在寻找一个好的可视化方式。
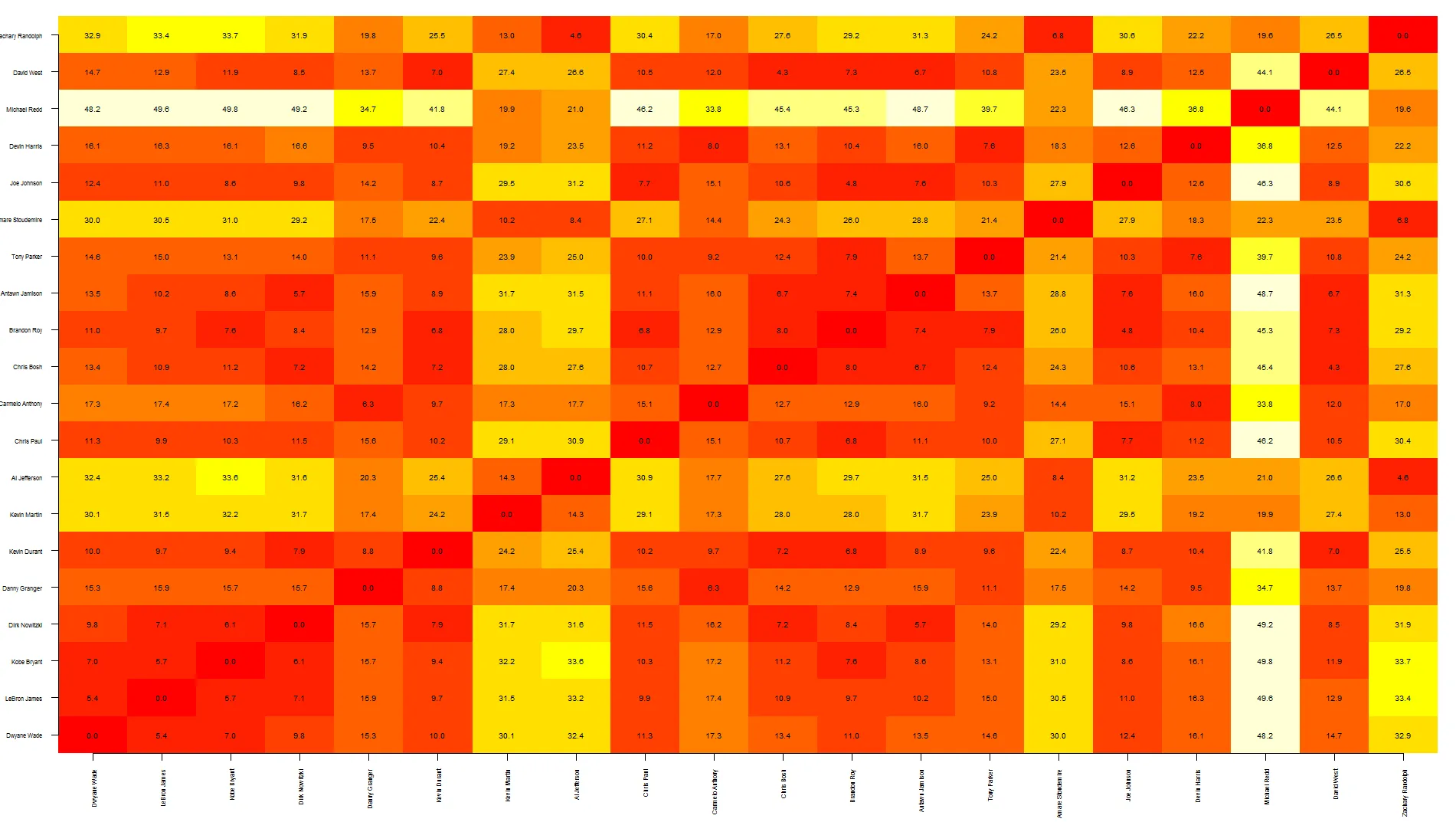
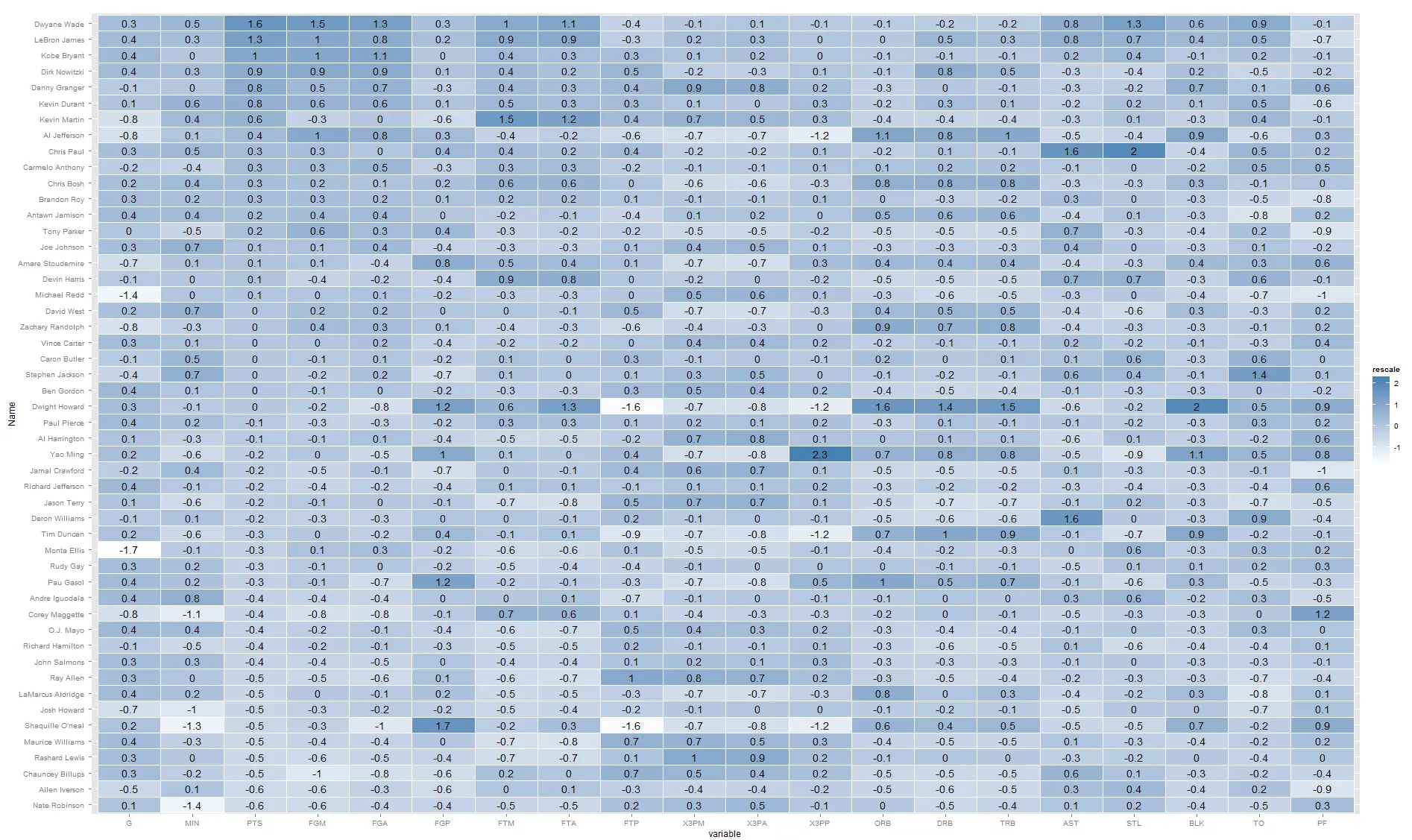
到目前为止,我发现了气球图(我在这里使用了它, 但我认为在这种情况下它不起作用),热力图(这是一个很好的例子,但它们不允许在表格中呈现数字,如果我错了请纠正我。也许将一半的表格着色,另一半显示数字会很酷),最后是相关椭圆图(这里是一些代码和示例- 使用形状很酷,但我不确定如何在这里使用它)。
还有各种聚类方法,但它们会聚合数据(这不是我想要的),而我想呈现所有数据。
数据示例:
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
dist(nba[1:20, -1], )
我愿意接受任何想法。