你得到那个奇怪的数组是因为它将每个序列视为一个条目并尝试对其进行独热编码,可以使用一个示例来说明:
import pandas as pd
from sklearn.preprocessing import OneHotEncoder
df = pd.DataFrame({'sequence':['AQAVPW','AMAVLT','LDTGIN']})
enc = OneHotEncoder()
seq = np.array(df['sequence']).reshape(-1,1)
encoded = enc.fit(seq)
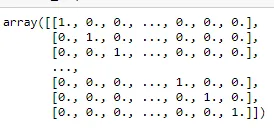
encoded.transform(seq).toarray()
array([[0., 1., 0.],
[1., 0., 0.],
[0., 0., 1.]])
encoded.categories_
[array(['AMAVLT', 'AQAVPW', 'LDTGIN'], dtype=object)]
由于您的条目是唯一的,因此您会得到这个全零矩阵。如果使用 pd.get_dummies,可以更好地理解这一点。
pd.get_dummies(df['sequence'])
AMAVLT AQAVPW LDTGIN
0 0 1 0
1 1 0 0
2 0 0 1
有两种方法可以做到这一点,一种方法是简单地计算氨基酸出现的频次,并将其用作预测因子。希望我能正确地记得氨基酸(很久以前学过):
from Bio import SeqIO
from Bio.SeqUtils.ProtParam import ProteinAnalysis
pd.DataFrame([ProteinAnalysis(i).count_amino_acids() for i in df['sequence']])
A C D E F G H I K L M N P Q R S T V W Y
0 2 0 0 0 0 0 0 0 0 0 0 0 1 1 0 0 0 1 1 0
1 2 0 0 0 0 0 0 0 0 1 1 0 0 0 0 0 1 1 0 0
2 0 0 1 0 0 1 0 1 0 1 0 1 0 0 0 0 1 0 0 0
另一种方法是将序列拆分,并通过位置进行编码,这需要序列长度相等,并且有足够的内存支持:
byposition = df['sequence'].apply(lambda x:pd.Series(list(x)))
byposition
0 1 2 3 4 5
0 A Q A V P W
1 A M A V L T
2 L D T G I N
pd.get_dummies(byposition)
0_A 0_L 1_D 1_M 1_Q 2_A 2_T 3_G 3_V 4_I 4_L 4_P 5_N 5_T 5_W
0 1 0 0 0 1 1 0 0 1 0 0 1 0 0 1
1 1 0 0 1 0 1 0 0 1 0 1 0 0 1 0
2 0 1 1 0 0 0 1 1 0 1 0 0 1 0 0
pd.Series(ProteinAnalysis("AA-QVT").count_amino_acids()).sum(),结果为5。您可能需要再次检查所有序列。 - StupidWolf