我有一个数据集,看起来像这样:
gene AE CM PR CD34
DDR1 8.020745 7.851901 7.520458 7.948326
RFC2 7.778460 8.175659 7.978560 8.845708
PAX8 9.566903 9.589379 9.405003 8.853069
GUCA1A 5.320824 5.318613 5.333363 5.424622
UBA7 10.422949 10.193109 9.357451 9.985480
GAPDH 13.559894 13.739593 13.517791 12.047161
GAPD 13.619479 13.790955 13.670576 12.994784
STAT_1 10.175952 9.911392 9.424882 10.024869
STAT3 7.089633 6.898931 6.654907 5.894336
STAT4 8.843160 8.746647 8.389753 8.024894
我想为每一对可能的变量绘制散点图,为此我使用了以下R命令:
d<-read.table("Dataset.txt", header=T, sep="\t")
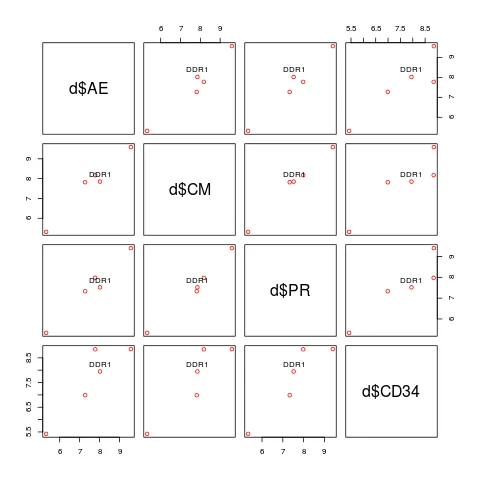
pairs(~d$AE+d$CM+d$PR+d$CD34,col="red")
现在,我希望离群值能够用基因名称标记出来。例如,DDR1在AE与CD34之间的差异为0.5。因此,在该图中应该用基因名称(在这种情况下为DDR1)标记它。
总的来说,对于任何一对显示出+-0.5差异的基因都应该作出标记。
数据: 我的数据的前100行的dput()结果。
structure(list(gene = structure(c(25L, 57L, 38L, 47L, 36L), .Label = c("ADAM32",
"AFG3L1", "AKD1", "ALG10", "ARMCX4", "ATP6V1E2", "BEST4", "C15orf40",
"C19orf26", "C4orf33", "C8orf45", "C8orf47", "C9orf30", "CATSPER1",
"CCDC11", "CCDC65", "CCL5", "CILP2", "CNOT7", "CORO6", "CRYZL1",
"CTCFL", "CYP2A6", "CYP2E1", "DDR1", "EPHB3", "ESRRA", "EYA3",
"FAM122C", "FAM18B2", "FAM71A", "FLJ30901", "GAPT", "GIMAP1",
"GSC", "GUCA1A", "HOXD4", "HSPA6", "KLK8", "LEAP2", "MAPK1",
"MFAP3", "MGC16385", "MSI2", "NCRNA00152", "NEXN", "PAX8", "PDE7A",
"PIGX", "PRR22", "PRSS33", "PTPN21", "PXK", "RAX2", "RBBP6",
"RDH10", "RFC2", "SCARB1", "SCIN", "SLC39A13", "SLC39A5", "SLC46A1",
"SP7", "SPATA17", "SRrp35", "THRA", "TIMD4", "TIRAP", "TMEM106A",
"TMEM196", "TRIOBP", "TTC39C", "TTLL12", "UBA7", "VPS18", "WFDC2",
"ZDHHC11", "ZNF333"), class = "factor"), AE = c(8.0207450402,
7.7784602732, 7.274639204, 9.5669027802, 5.3208241196), CM = c(7.8519007358,
8.1756591822, 7.8186806878, 9.5893790363, 5.3186130064), PR = c(7.5204580759,
7.9785595029, 7.3307638794, 9.4050027059, 5.3333625256), CD34 = c(7.9483258833,
8.8457084131, 6.9874872331, 8.8530693617, 5.4246223945), CMP = c(7.9362235824,
10.0085488406, 6.6883401632, 9.249816924, 5.4312885508), GMP = c(8.1370096058,
10.0990080444, 6.7144163183, 9.2157346882, 5.3700062534), Monocytes = c(8.1912723165,
8.7568163628, 9.2072172453, 9.2086040758, 5.3090689608)), .Names = c("gene",
"AE", "CM", "PR", "CD34", "CMP", "GMP", "Monocytes"), row.names = c(NA,
5L), class = "data.frame")
dput命令分享一下您的数据呢?(参考链接:https://dev59.com/eG025IYBdhLWcg3whGSx) - rbm