我正在尝试在R(通过Rstudio)中运行rjags来估计参数alpha和beta以及模型的超参数tau.nu:
y_i|x_i~pois(eta_i),
eta_i=exp(alpha + beta*x_i + nu_i),
nu_i~N(0,tau.nu)
这是我的代码:
#generating data
N = 1000
x = rnorm(N, mean=3,sd=1)
nu = rnorm(N,0,0.01)
eta = exp(1 + 2*x + nu)
y = rpois(N,eta)
data=data.frame(y=y,x=x)
###MCMC
library(rjags)
library(coda)
mod_string= "model {
for(i in 1:1000) {
y[i]~dpois(eta[i])
eta[i]=exp(alpha+beta*x[i]+nu[i])
nu[i]~dnorm(0,tau.nu)
}
alpha ~ dnorm(0,0.001)
beta ~ dnorm(0,0.001)
tau.nu ~ dgamma(0.01,0.01)
}"
params = c("alpha","beta","tau.nu")
inits = function() {
inits = list("alpha"=rnorm(1,0,100),"beta"=rnorm(1,0,80),"tau.nu"=rgamma(1,1,1))
}
mod = jags.model(textConnection(mod_string), data=data, inits=inits, n.chains =3)
update(mod,5000)
mod_sim = coda.samples(model=mod,
variable.names=params,
n.iter=2e4)
mod_csim = as.mcmc(do.call(rbind, mod_sim))
plot(mod_csim)
我得到了奇怪的输出,不知道哪里出错了。在这个模型中,MCMC是否不起作用?还是我的编码有问题?
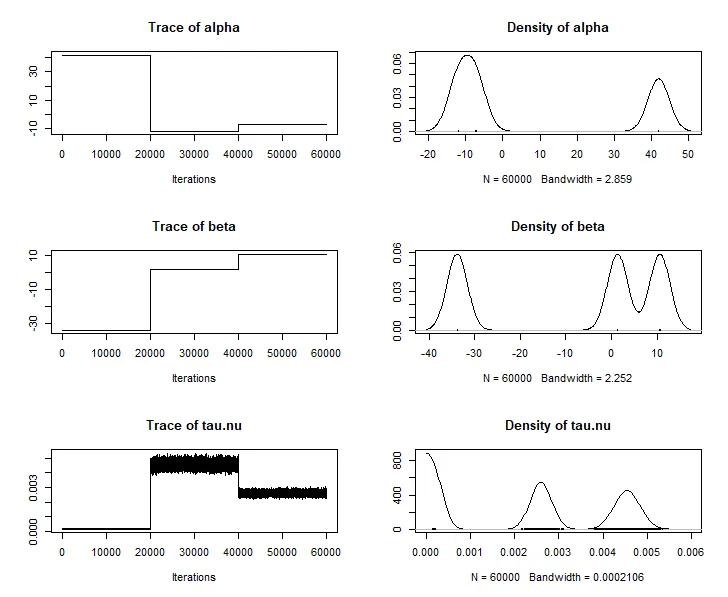
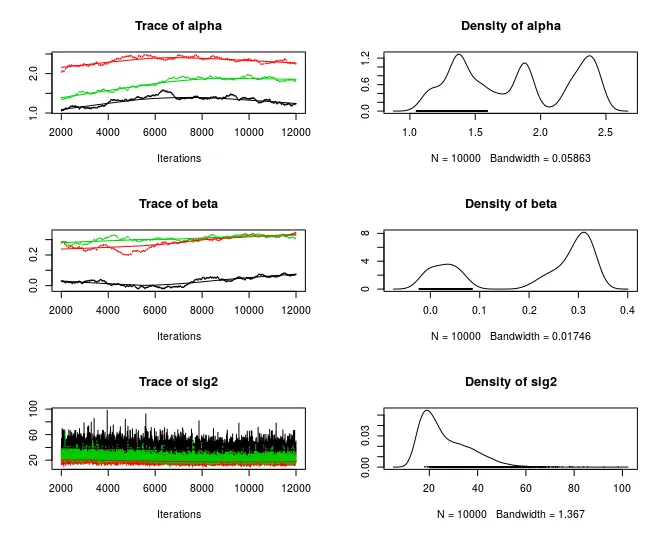
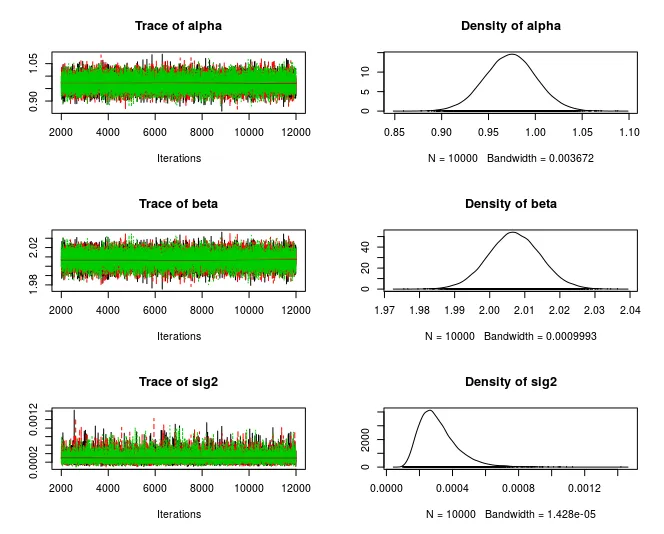
nu的先验项移出似然函数,增加了burnin和迭代次数——但这些更改没有任何影响。在加载glm模块(load.module("glm"))后,它按预期收敛了。(注意,我使用了N=100,否则会花费太长时间) - user20650glm模块,它收敛了,但我仍然有问题:我将迭代次数设置为5e4,为什么x轴的范围是0~1.5e5?而且tau.mu的估计值与真实值0.01相差甚远。 - Yukitau.nu是精度。要返回方差,请在先验下添加一行,sig2 = 1 / tau.nu,然后使用sig2替代tau.nu。 从你的示例中可以看出,图表的 x 轴迭代取决于jags.model中的n.adapt(1000)+coda.samples中的 5000 burnin + n.iter (2e4),因此我预计 x 轴从 6000 开始,并在plot(mod_sim)中到达 26000 - 这也是我认为你真正想要的。 由于您正在组合链,所以您会得到 3*5e4 = 1.5e5 -- 因此一个接一个连续出现。 - user20650bayesplot包,但当我尝试添加其他方法的输出时,它会出现问题。所以我尝试了R2jags包中的jags.hist函数,它报错“未找到jags.hist函数”,即使我运行了示例案例[1](https://rdrr.io/github/mbtyers/jagsplot/man/jags.hist.html)。当然,我已经安装了`R2jags`。 - Yuki