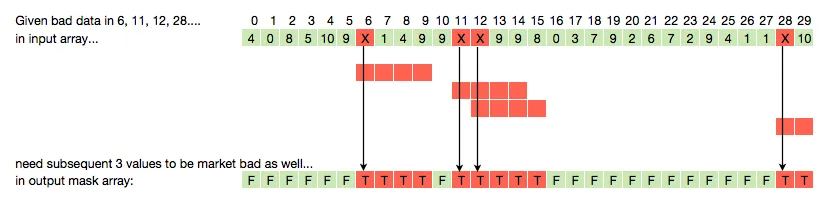
这里是基准测试结果。我包含了自己的代码("op"),它首先循环遍历坏索引并将1...n添加到它们中,然后取唯一值以查找掩码索引。您可以在下面的代码中看到它,以及其他所有响应。
无论如何,以下是结果。第一维是数组大小沿x(从10到10e7),第二维是窗口大小沿y(5、50、500、5000)。然后是每个方格中的编码器,带有对数10得分,因为我们正在讨论微秒到分钟。
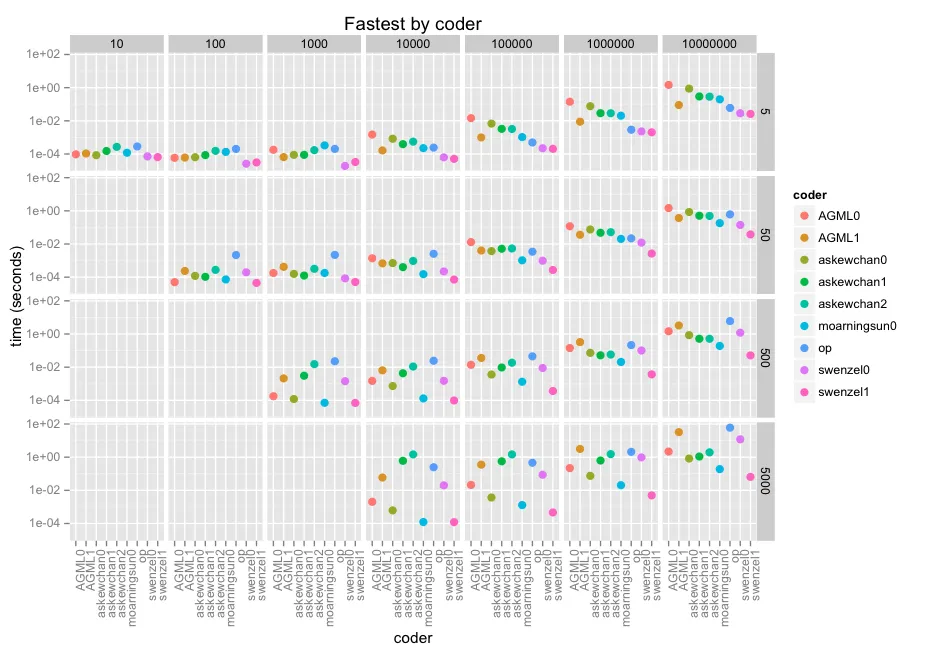
@swenzel似乎是获胜者,他的第二个答案取代了@moarningsun的第一个答案(moarningsun的第二个答案通过大量内存使用使机器崩溃,但这可能是因为它没有设计用于大的n或非稀疏a)。
图表不能完全反映这些贡献中最快的,因为(必要的)对数比例尺。它们比甚至是不错的循环解决方案快几十倍或几百倍。在最大的情况下,swenzel1比op快1000倍,而op已经利用了numpy。
请注意,我使用的是针对优化的英特尔MKL库编译的numpy版本,该库充分利用了自2012年以来存在的AVX指令。在某些矢量使用情况下,这将使i7/Xeon速度提高5倍。某些贡献可能受益更多。
这是运行到目前为止提交的所有答案(包括我的)的完整代码。函数allagree()确保结果是正确的,而timeall()将为您提供一个长格式的pandas数据帧,其中包含所有结果(以秒为单位)。
您可以相当容易地重新运行它以使用新代码或更改我的假设。请记住,我没有考虑其他因素,如内存使用。另外,我采用了R ggplot2作为图形,因为我不太熟悉seaborn/matplotlib,无法让它按我的意愿工作。
为了完整起见,所有结果都是一致的:
In [4]: allagree(n = 7, asize = 777)
Out[4]:
AGML0 AGML1 askewchan0 askewchan1 askewchan2 moarningsun0 \
AGML0 True True True True True True
AGML1 True True True True True True
askewchan0 True True True True True True
askewchan1 True True True True True True
askewchan2 True True True True True True
moarningsun0 True True True True True True
swenzel0 True True True True True True
swenzel1 True True True True True True
op True True True True True True
swenzel0 swenzel1 op
AGML0 True True True
AGML1 True True True
askewchan0 True True True
askewchan1 True True True
askewchan2 True True True
moarningsun0 True True True
swenzel0 True True True
swenzel1 True True True
op True True True
感谢所有提交的人!
在使用R中的pd.to_csv和read.csv导出timeall()输出后,用于制作图形的代码:
ww <- read.csv("ww.csv")
ggplot(ww, aes(x=coder, y=value, col = coder)) + geom_point(size = 3) + scale_y_continuous(trans="log10")+ facet_grid(nsize ~ asize) + theme(axis.text.x = element_text(angle = 90, hjust = 1)) + ggtitle("Fastest by coder") + ylab("time (seconds)")
测试用代码:
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
from timeit import Timer
from scipy import ndimage
from skimage import morphology
import itertools
import pdb
np.random.seed(8472)
def AGML0(a, n):
maskleft = np.where(np.isnan(a))[0]
maskright = maskleft + n
mask = np.zeros(len(a),dtype=bool)
for l,r in itertools.izip(maskleft,maskright):
mask[l:r] = True
return mask
def AGML1(a, n):
nn = n - 1
maskleft = np.where(np.isnan(a))[0]
ghost_mask = np.zeros(len(a)+nn,dtype=bool)
for i in range(0, nn+1):
thismask = maskleft + i
ghost_mask[thismask] = True
mask = ghost_mask[:len(ghost_mask)-nn]
return mask
def askewchan0(a, n):
m = np.isnan(a)
i = np.arange(1, len(m)+1)
ind = np.column_stack([i-n, i])
ind.clip(0, len(m)-1, out=ind)
return np.bitwise_or.reduceat(m, ind.ravel())[::2]
def askewchan1(a, n):
m = np.isnan(a)
s = np.full(n, True, bool)
return ndimage.binary_dilation(m, structure=s, origin=-(n//2))
def askewchan2(a, n):
m = np.isnan(a)
s = np.zeros(2*n - n%2, bool)
s[-n:] = True
return morphology.binary_dilation(m, selem=s)
def moarningsun0(a, n):
mask = np.isnan(a)
cs = np.cumsum(mask)
cs[n:] -= cs[:-n].copy()
return cs > 0
def moarningsun1(a, n):
mask = np.isnan(a)
idx = np.flatnonzero(mask)
expanded_idx = idx[:,None] + np.arange(1, n)
np.put(mask, expanded_idx, True, 'clip')
return mask
def swenzel0(a, n):
m = np.isnan(a)
k = m.copy()
for i in range(1, n):
k[i:] |= m[:-i]
return k
def swenzel1(a, n=4):
m = np.isnan(a)
k = m.copy()
lenM, lenK = 1, 1
while lenM+lenK < n:
m[lenM:] |= k[:-lenM]
m, k = k, m
lenM, lenK = lenK, lenM+lenK
k[n-lenM:] |= m[:-n+lenM]
return k
def op(a, n):
m = np.isnan(a)
for x in range(1, n):
m = np.logical_or(m, np.r_[False, m][:-1])
return m
funcs = [AGML0, AGML1, askewchan0, askewchan1, askewchan2, moarningsun0, swenzel0, swenzel1, op]
def allagree(fns = funcs, n = 10, asize = 100):
""" make sure result is the same from all functions """
fnames = [f.__name__ for f in fns]
a = np.random.rand(asize)
a[np.random.randint(0, asize, int(asize / 10))] = np.nan
results = dict([(f.__name__, f(a, n)) for f in fns])
isgood = [[np.array_equal(results[f1], results[f2]) for f1 in fnames] for f2 in fnames]
pdgood = pd.DataFrame(isgood, columns = fnames, index = fnames)
if not all([all(x) for x in isgood]):
print "not all results identical"
pdb.set_trace()
return pdgood
def timeone(f):
""" time one of the functions across the full range of a nd n """
print "Timing", f.__name__
Ns = np.array([10**x for x in range(0, 4)]) * 5
As = [np.random.rand(10 ** x) for x in range(1, 8)]
for i in range(len(As)):
As[i][np.random.randint(0, len(As[i]), len(As[i]) / 10)] = np.nan
results = np.array([[Timer(lambda: f(a, n)).timeit(number = 1) if n < len(a) \
else np.nan for n in Ns] for a in As])
pdresults = pd.DataFrame(results, index = [len(x) for x in As], columns = Ns)
return pdresults
def timeall(fns = funcs):
""" run timeone for all known funcs """
testd = dict([(x.__name__, timeone(x)) for x in fns])
testdf = pd.concat(testd.values(), axis = 0, keys = testd.keys())
testdf.index.names = ["coder", "asize"]
testdf.columns.names = ["nsize"]
testdf.reset_index(inplace = True)
testdf = pd.melt(testdf, id_vars = ["coder", "asize"])
return testdf