最快的选项不仅取决于DataFrame的长度(在这种情况下,约为13M行),还取决于组数。以下是性能图,比较了多种查找每个组中最大值的方法:
如果只有一些(大)组,则使用using_idxmax可能是最快的选择:
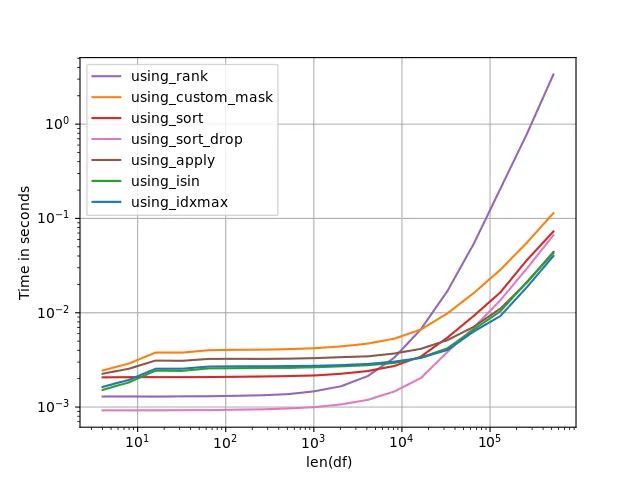
如果有许多(小)组且DataFrame不太大,则使用using_sort_drop可能是最快的选择:
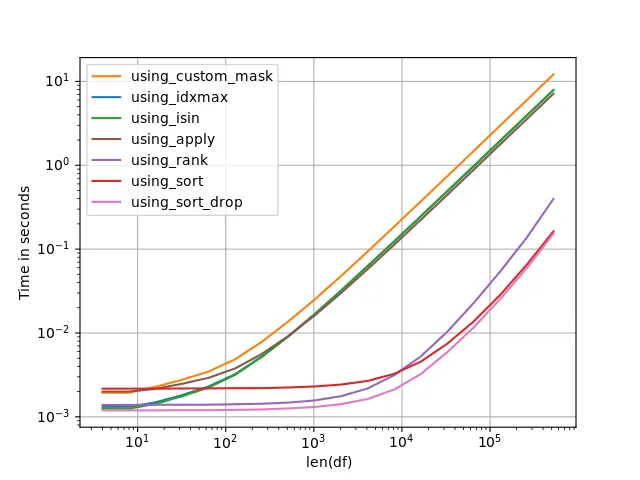
但请记住,尽管using_sort_drop、using_sort和using_rank开始看起来非常快,但随着N = len(df)的增加,它们相对于其他选项的速度很快就会消失。对于足够大的N,即使有许多组,using_idxmax也成为最快的选项。
使用
using_sort_drop、
using_sort和
using_rank对DataFrame(或DataFrame内的分组)进行排序。平均排序时间复杂度为
O(N * log(N)),而其他方法使用
O(N)操作。这就是为什么像
using_idxmax这样的方法在处理非常大的数据集时要比
using_sort_drop更快的原因。
请注意,基准测试结果可能会因多种原因而有所不同,包括机器规格、操作系统和软件版本。因此,在自己的机器上运行基准测试,并使用适合自己情况的测试数据非常重要。
根据上面的性能图表,如果您的DataFrame有许多(小)分组,则使用
using_sort_drop可能是值得考虑的选项,尤其是当它有1300万行时。否则,我会怀疑
using_idxmax是最快的选项——但再次强调,在自己的机器上检查基准测试非常重要。
以下是我用来制作perfplots的设置:
import numpy as np
import pandas as pd
import perfplot
def make_df(N):
df = pd.DataFrame(np.random.randint(N//10+1, size=(N, 2)), columns=['Id','delta'])
return df
def using_idxmax(df):
return df.loc[df.groupby("Id")['delta'].idxmax()]
def max_mask(s):
i = np.asarray(s).argmax()
result = [False]*len(s)
result[i] = True
return result
def using_custom_mask(df):
mask = df.groupby("Id")['delta'].transform(max_mask)
return df.loc[mask]
def using_isin(df):
idx = df.groupby("Id")['delta'].idxmax()
mask = df.index.isin(idx)
return df.loc[mask]
def using_sort(df):
df = df.sort_values(by=['delta'], ascending=False, kind='mergesort')
return df.groupby('Id', as_index=False).first()
def using_rank(df):
mask = (df.groupby('Id')['delta'].rank(method='first', ascending=False) == 1)
return df.loc[mask]
def using_sort_drop(df):
return df.sort_values(by=['delta'], ascending=False, kind='mergesort').drop_duplicates('Id')
def using_apply(df):
selected_idx = df.groupby("Id").apply(lambda df: df.delta.argmax())
return df.loc[selected_idx]
def check(df1, df2):
df1 = df1.sort_values(by=['Id','delta'], kind='mergesort').reset_index(drop=True)
df2 = df2.sort_values(by=['Id','delta'], kind='mergesort').reset_index(drop=True)
return df1.equals(df2)
perfplot.show(
setup=make_df,
kernels=[using_idxmax, using_custom_mask, using_isin, using_sort,
using_rank, using_apply, using_sort_drop],
n_range=[2**k for k in range(2, 20)],
logx=True,
logy=True,
xlabel='len(df)',
repeat=75,
equality_check=check)
另一种基准测试方法是使用 IPython %timeit:
In [55]: df = make_df(2**20)
In [56]: %timeit using_sort_drop(df)
1 loop, best of 3: 403 ms per loop
In [57]: %timeit using_rank(df)
1 loop, best of 3: 1.04 s per loop
In [58]: %timeit using_idxmax(df)
1 loop, best of 3: 15.8 s per loop

