重新生成您的示例:
clotting <- data.frame(
u = c(5,10,15,20,30,40,60,80,100),
lot1 = c(118,58,42,35,27,25,21,19,18),
lot2 = c(69,35,26,21,18,16,13,12,12))
glm2 <- glm(lot2 ~ log(u), data=clotting, family=Gamma)
profile.glm函数实际上是在MASS包中:
library(MASS)
prof<-profile(glm2)
为了弄清楚
profile.glm和
plot.profile在做什么,请参见
?profile.glm和
?plot.profile。然而,为了深入了解
profile对象,检查
MASS :::profile.glm和
MASS :::plot.profile的代码也可能很有用...基本上,这些告诉您的是
profile返回偏差与最小偏差之间差的
有符号平方根,由离散参数缩放。这样做的原因是使完全二次函数图像的轮廓显示为一条直线(通过眼睛检测偏离直线比偏离抛物线更容易)。
另一个有用的信息是如何存储此轮廓。基本上,它是一个数据帧列表(每个轮廓化参数一个),除了各个数据帧有点奇怪(包含一个向量组件和一个矩阵组件)。
> str(prof)
List of 2
$ (Intercept):'data.frame': 12 obs. of 3 variables:
..$ tau : num [1:12] -3.557 -2.836 -2.12 -1.409 -0.702 ...
..$ par.vals: num [1:12, 1:2] -0.0286 -0.0276 -0.0267 -0.0258 -0.0248 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : NULL
.. .. ..$ : chr [1:2] "(Intercept)" "log(u)"
..$ dev : num [1:12] 0.00622 0.00753 0.00883 0.01012 0.0114 ...
$ log(u) :'data.frame': 12 obs. of 2 variables:
..$ tau : num [1:12] -3.516 -2.811 -2.106 -1.403 -0.701 ...
..$ par.vals: num [1:12, 1:2] -0.0195 -0.0204 -0.0213 -0.0222 -0.023 ...
.. ..- attr(*, "dimnames")=List of 2
它还包含了
summary 和
original.fit 两个属性,你可以利用它们来恢复离散度和最小偏差:
disp <- attr(prof,"summary")$dispersion
mindev <- attr(prof,"original.fit")$deviance
现在对参数1进行反向转换:
dev1 <- prof[[1]]$tau^2
dev2 <- dev1*disp+mindev
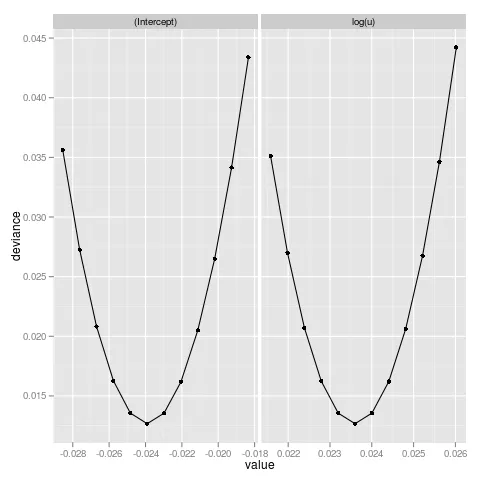
情节:
plot(prof[[1]][,1],dev2,type="b")
这是离差的图示。您可以乘以0.5得到负对数似然,或者乘以-0.5得到对数似然...
编辑:一些更普遍的函数可将配置文件转换为适用于lattice/ggplot绘图的有用格式...
tmpf <- function(x,n) {
data.frame(par=n,tau=x$tau,
deviance=x$tau^2*disp+mindev,
x$par.vals,check.names=FALSE)
}
pp <- do.call(rbind,mapply(tmpf,prof,names(prof),SIMPLIFY=FALSE))
library(reshape2)
pp2 <- melt(pp,id.var=1:3)
pp3 <- subset(pp2,par==variable,select=-variable)
现在使用lattice绘制它:
library(lattice)
xyplot(deviance~value|par,type="b",data=pp3,
scales=list(x=list(relation="free")))
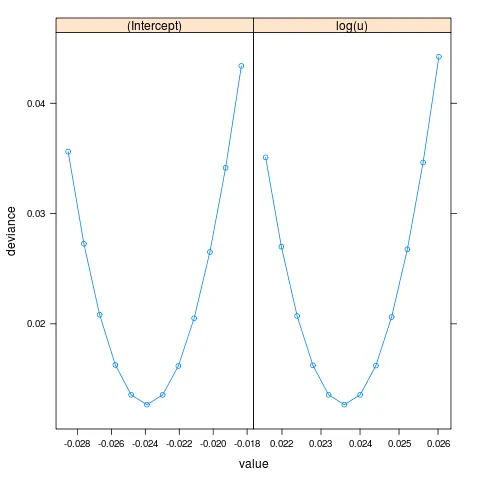
或者使用ggplot2:
library(ggplot2)
ggplot(pp3,aes(value,deviance))+geom_line()+geom_point()+
facet_wrap(~par,scale="free_x")