使用statsmodels的UnobservedComponents拟合本地级别模型后,我们正在尝试找到使用结果模拟新时间序列的方法。类似于:
import numpy as np
import statsmodels as sm
from statsmodels.tsa.statespace.structural import UnobservedComponents
np.random.seed(12345)
ar = np.r_[1, 0.9]
ma = np.array([1])
arma_process = sm.tsa.arima_process.ArmaProcess(ar, ma)
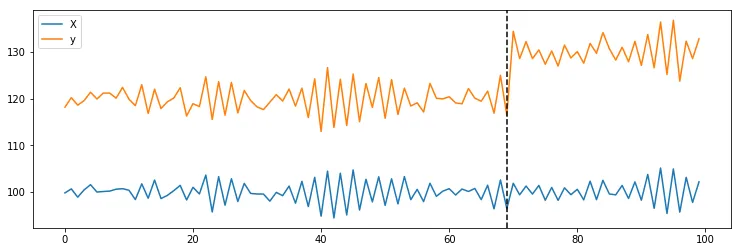
X = 100 + arma_process.generate_sample(nsample=100)
y = 1.2 * x + np.random.normal(size=100)
y[70:] += 10
plt.plot(X, label='X')
plt.plot(y, label='y')
plt.axvline(69, linestyle='--', color='k')
plt.legend();
ss = {}
ss["endog"] = y[:70]
ss["level"] = "llevel"
ss["exog"] = X[:70]
model = UnobservedComponents(**ss)
trained_model = model.fit()
是否可以使用trained_model来模拟给定外生变量X[70:]的新时间序列?就像我们有arma_process.generate_sample(nsample=100)一样,我们想知道是否可以做类似的事情:
trained_model.generate_random_series(nsample=100, exog=X[70:])
这样做的动机是为了计算时间序列与观察到的
y[70:] 一样极端的概率(用于确定响应大于预测值的p值)。
[编辑]
在阅读Josef和cfulton的评论后,我尝试实现以下内容:mod1 = UnobservedComponents(np.zeros(y_post), 'llevel', exog=X_post)
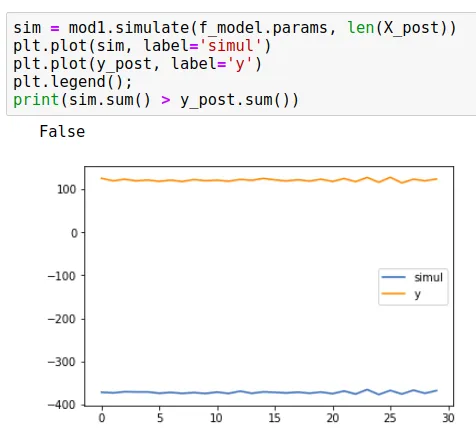
mod1.simulate(f_model.params, len(X_post))
但是这导致模拟似乎无法跟踪X_post作为exog的predicted_mean预测结果。以下是一个例子:
y_post徘徊在100左右时,模拟值为-400。这种方法总是导致p_value为50%。因此,当我尝试使用
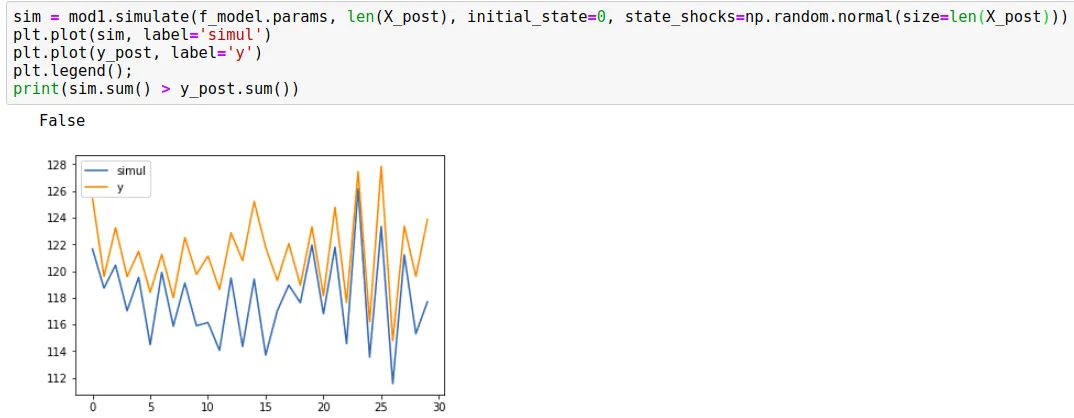
initial_state=0和随机冲击时,结果如下:
现在似乎模拟正在遵循预测的平均值及其95%的置信区间(正如cfulton在下面评论的那样,这实际上是一种错误的方法,因为它替换了训练模型的级别方差)。我尝试使用这种方法来观察我会得到什么p值。以下是我计算p值的方法:
samples = 1000
r = 0
y_post_sum = y_post.sum()
for _ in range(samples):
sim = mod1.simulate(f_model.params, len(X_post), initial_state=0, state_shocks=np.random.normal(size=len(X_post)))
r += sim.sum() >= y_post_sum
print(r / samples)
为了说明,这是由Google开发的Causal Impact模型。由于它已经在R中实现,我们一直在尝试使用
statsmodels作为核心来处理时间序列,用Python复制实现。我们已经有了一个相当酷的WIP 实现, 但我们仍然需要p值来知道我们是否存在影响不仅仅是由于随机性(模拟系列和计算总和超过
y_post.sum()的方法在Google的模型中也被实现)。在我的示例中,我使用了
y[70:] += 10。如果我只加了一个而不是十个,Google的p值计算返回0.001(y存在影响),而在Python的方法中,它返回0.247(没有影响)。只有当我将
y_post加上+5时,模型才会返回p_value为0.02,并且由于低于0.05,我们认为y_post受到影响。 我正在使用python3,statsmodels版本为0.9.0。在阅读cfulton的评论后,我决定完全调试代码以查看发生了什么。这是我发现的:当我们创建一个类型为UnobservedComponents的对象时,最终启动卡尔曼滤波器的表示形式。默认情况下,它接收参数initial_variance,其设置对象的同一属性为1e6。当我们运行
simulate方法时,使用相同的值创建initial_state_cov is created。initial_state_cov = (
np.eye(self.k_states, dtype=self.ssm.transition.dtype) *
self.ssm.initial_variance
)
最后,同样的值被用来查找 initial_state:
initial_state = np.random.multivariate_normal(
self._initial_state, self._initial_state_cov)
这将导致标准差为1e6的正态分布。 然后我尝试运行以下内容:
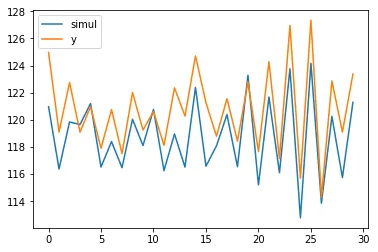
mod1 = UnobservedComponents(np.zeros(len(X_post)), level='llevel', exog=X_post, initial_variance=1)
sim = mod1.simulate(f_model.params, len(X_post))
plt.plot(sim, label='simul')
plt.plot(y_post, label='y')
plt.legend();
print(sim.sum() > y_post.sum())
这导致:
我测试了p值,最终发现在y_post变化1个单位时,模型现在能正确地识别添加的信号。
然而,当我使用R的Google包中相同的数据进行测试时,p值仍然不准确。也许需要进一步调整输入以提高其准确性。
simulate方法,但是理解为只适用于模型对象而不是训练好的模型。我也可以从训练好的模型中获取参数来构建一个新模型(有点间接,但我仍然希望它能够奏效)。 - Willian Fuksjupyter/datascience-notebook:latestDocker镜像中运行笔记本。我已经尝试重新启动所有内容,但结果仍然与预期相差很大(有时几乎达到-1000)。 - Willian Fuks