很遗憾,您要求ggplot2为每个facet定义单独的y,但在我所知道的范围内,它在语法上无法实现。
因此,针对您在评论线程中提到的“基本上只想要一个直方图”,我建议改用geom_histogram,或者如果您偏爱线条而不是条形图,则使用geom_freqpoly:
ggplot(iris, aes(Sepal.Width, ..count..)) +
geom_histogram(aes(colour=Species, fill=Species), binwidth=.2) +
geom_freqpoly(colour="black", binwidth=.2) +
facet_wrap(~Species)
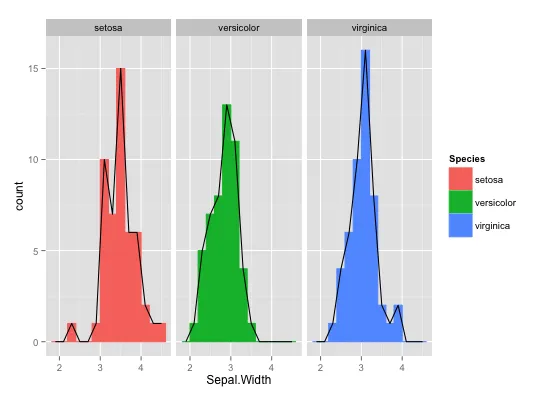
注意:在上面的示例中,geom_freqpoly与geom_histogram一样有效。我只是将两者都添加到一个图中以提高效率。
希望这可以帮助你。
编辑:好吧,我设法想出了一种快速而简单的方法来获得你想要的结果。它需要安装和加载plyr。提前道歉;从RAM使用方面来看,这可能不是最有效的方法,但它确实起作用。
首先,让我们打开iris(我使用RStudio,所以习惯于在窗口中查看所有对象):
d <- iris
现在,我们可以使用
ddply来计算每个唯一测量值所属的个体数量,这将成为您的x轴(这里我使用Sepal.Length而不是Sepal.Width,以便在绘制时能够看到更大的差异,从而获得更多范围)。
new <- ddply(d, c("Species", "Sepal.Length"), summarize, count=length(Sepal.Length))
请注意,
ddply会根据引用的变量自动对输出数据框进行排序。
然后我们可以将数据框分成每个独特的条件--在iris数据集中,每个三个物种(如果您处理的是大量数据,则不建议继续创建相同数据框的子集,因为可能会耗尽内存)。
set <- new[which(new$Species%in%"setosa"),]
ver <- new[which(new$Species%in%"versicolor"),]
vgn <- new[which(new$Species%in%"virginica"),]
使用ddply再次计算每个测量值下属于每个物种的个体比例,但是需要分别进行计算。
prop <- rbind(ddply(set, c("Species"), summarize, prop=set$count/sum(set$count)),
ddply(ver, c("Species"), summarize, prop=ver$count/sum(ver$count)),
ddply(vgn, c("Species"), summarize, prop=vgn$count/sum(vgn$count)))
然后,我们只需将所有需要的内容放入一个数据集中,并从我们的工作区中删除所有垃圾。
new$prop <- prop$prop
rm(list=ls()[which(!ls()%in%c("new", "d"))])
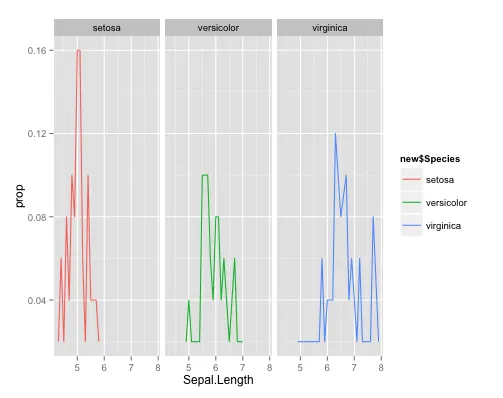
我们可以按照每个面的比例在y轴上制作图形。请注意,我现在使用geom_line,因为ddply已经自动对您的数据框进行了排序。
ggplot(new, aes(Sepal.Length, prop)) +
geom_line(aes(colour=new$Species)) +
facet_wrap(~Species)
sum(new$count[which(new$Species%in%"setosa")])
sum(new$count[which(new$Species%in%"versicolor")])
sum(new$count[which(new$Species%in%"versicolor")])
sum(new$prop[which(new$Species%in%"setosa")])
sum(new$prop[which(new$Species%in%"versicolor")])
sum(new$prop[which(new$Species%in%"versicolor")])


ggplot(iris[iris$Species == 'virginica',]) + geom_density(aes(x=Sepal.Width, colour=Species, y=..count../sum(..count..))) + facet_wrap(~Species)也没有给出百分比。所以我会编辑我的问题来澄清,谢谢。 - user248237