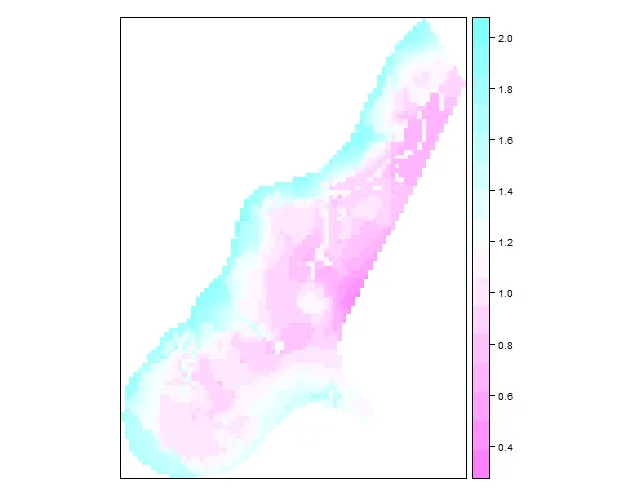
我使用gstat来预测二项式数据,但是预测值超过了1或者低于0。有人知道如何解决这个问题吗?谢谢。
data(meuse)
data(meuse.grid)
coordinates(meuse) <- ~x+y
coordinates(meuse.grid) <- ~x+y
gridded(meuse.grid) <- TRUE
#glm model
glm.lime <- glm(lime~dist+ffreq, meuse, family=binomial(link="logit"))
summary(glm.lime)
#variogram of residuals
var <- variogram(lime~dist+ffreq, data=meuse)
fit.var <- fit.variogram(var, vgm(nugget=0.9, "Sph", range=sqrt(diff(meuse@bbox\[1,\])^2 + diff(meuse@bbox\[2,\])^2)/4, psill=var(glm.lime$residuals)))
plot(var, fit.var, plot.nu=T)
#universal kriging
kri <- krige(lime~dist+ffreq, meuse, meuse.grid, fit.var)
spplot(kri[1])