有人能帮我理解为什么自动和手动克里金法得到的结果差异如此之大吗?我看到这两种算法使用了不同的变程模型,但这是否就是所有偏差的原因呢?我还不太确定的是,autokrige在没有数据的位置(例如网格的左下角和右下角)预测出的值明显比实际值高得多。这是否与将(对数转换的)克里金输出通过指数运算转换回来有关?此外,两种方法都预测出比数据低得多的值。似乎我在其中一个或两个算法中做错了什么,非常感谢任何帮助。
编辑:这里提供用于此处使用的数据的GitHub链接(我使用了此建议分享csv文件)。
编辑:这里提供用于此处使用的数据的GitHub链接(我使用了此建议分享csv文件)。
library(sp)
library(gstat)
library(automap)
# get data
df <- read.csv("data.csv")
---------------------------
> head(df)
long lat n
1 -76.7116 39.2992 1
2 -76.7113 39.3553 1
3 -76.7113 39.3607 1
4 -76.7112 39.3126 2
5 -76.7110 39.2973 1
6 -76.7110 39.3364 1
---------------------------
# generate regular grid
coordinates(df) <- ~ long + lat
grd <- spsample(df, type = "regular", n = 10000)
colnames(grd@coords) <- c("long", "lat") # rename coords as in data
gridded(grd) <- TRUE
# manual kriging
form <- as.formula("log(n) ~ long + lat") # universal kriging formula
v <- variogram(object = form, locations = df)
vmfit <- fit.variogram(v,
model = vgm(model = c("Sph", "Exp", "Mat", "Gau", "Ste")))
-----------------------------------
> vmfit
model psill range kappa
1 Ste 0.2886451 0.001786237 0.5
-----------------------------------
krg <- krige(formula = form, locations = df, newdata = grd, model = vmfit)
krg_df <- data.frame(krg@coords, # extract kriged data
pred = exp(krg@data$var1.pred))
# auto kriging
krg2 <- autoKrige(formula = form, input_data = df, new_data = grd)
krg2_df <- data.frame(krg2$krige_output@coords,
pred = exp(krg2$krige_output@data$var1.pred))
-----------------------------------
> krg2$var_model
model psill range
1 Nug 0.26473893 0.00000000
2 Sph 0.02574092 0.01657061
-----------------------------------
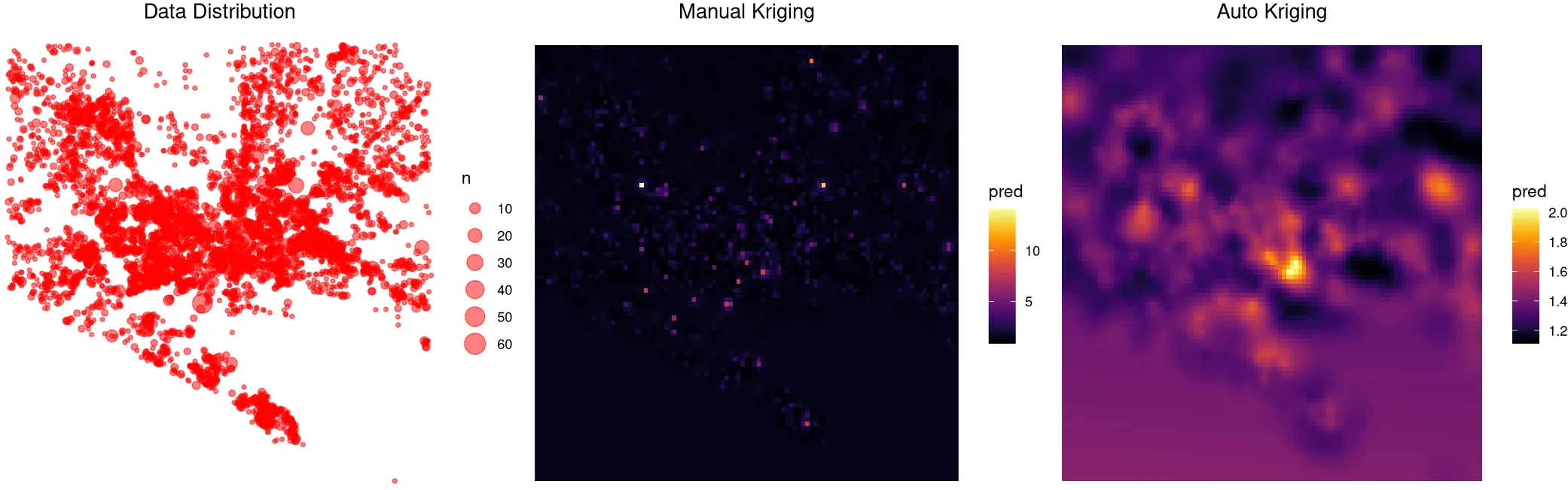
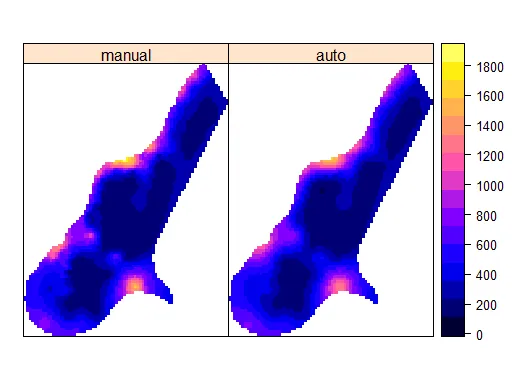
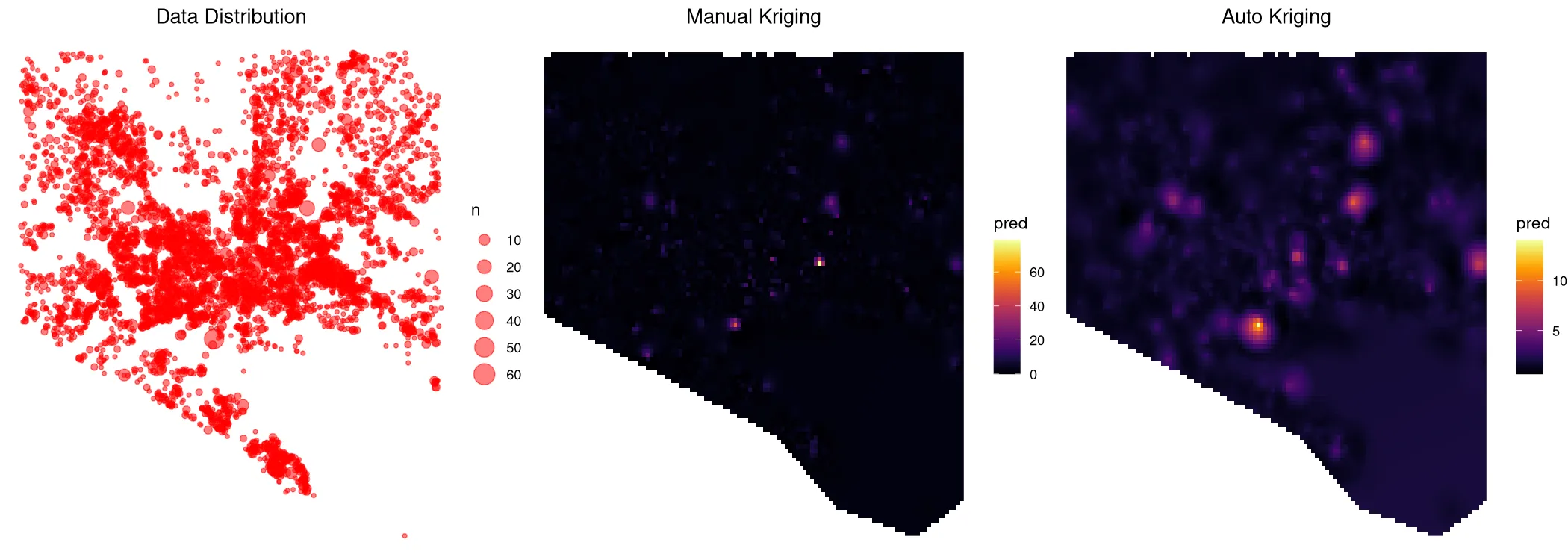
log(n) ~ long + lat(对数转换只是为了减少偏斜)。我首先尝试了meuse数据集,使用log(zinc) ~ x + y,得到了与您的非常相似的图形,两种方法之间没有明显的区别。它似乎可以在meuse数据集上工作,但不适用于我的数据。 - Manojit