我认为这里有两个问题。
现在我想要处理非常大的矩阵
那么请不要使用R代码来完成此工作。R将占用比您预期更多的内存。请尝试以下代码:
res.upper <- rnorm(4950)
res <- matrix(0, 100, 100)
tracemem(res)
res[upper.tri(res)] <- res.upper
rm(res.upper)
diag(res) <- 1
res[lower.tri(res)] <- t(res)[lower.tri(res)]
这是您将获得的内容:
> res.upper <- rnorm(4950) ## allocation of length 4950 vector
> res <- matrix(0, 100, 100) ## allocation of 100 * 100 matrix
> tracemem(res)
[1] "<0xc9e6c10>"
> res[upper.tri(res)] <- res.upper
tracemem[0xc9e6c10 -> 0xdb7bcf8]: ## allocation of 100 * 100 matrix
> rm(res.upper)
> diag(res) <- 1
tracemem[0xdb7bcf8 -> 0xdace438]: diag<- ## allocation of 100 * 100 matrix
> res[lower.tri(res)] <- t(res)[lower.tri(res)]
tracemem[0xdace438 -> 0xdb261d0]: ## allocation of 100 * 100 matrix
tracemem[0xdb261d0 -> 0xccc34d0]: ## allocation of 100 * 100 matrix
在R中,您需要使用
5 * (100 * 100) + 4950个双字来完成这些操作。而在C中,您只需要最多
4950 + 100 * 100个双字(实际上,只需要
100 * 100个双字!稍后会讨论)。在R中,直接覆盖对象而不进行额外的内存分配是困难的。
有没有办法可以直接将res.upper转换为对称矩阵,而无需先初始化矩阵res?
您确实需要为
res分配内存,因为这就是您最终得到的;但是,没有必要为
res.upper分配内存。您可以同时初始化上三角形,并填写下三角形。考虑以下模板:
#include <Rmath.h>
#include <R.h>
#include <Rinternals.h>
## N is matrix dimension, a length-1 integer vector in R
## this function returns the matrix you want
SEXP foo(SEXP N) {
int i, j, n = asInteger(N);
SEXP R_res = PROTECT(allocVector(REALSXP, n * n));
double *res = REAL(R_res);
double tmp;
getRNGstate();
for (i = 0; i < n; i++) {
res[i * n + i] = 1.0;
for (j = i + 1; j < n; j++) {
tmp = rnorm(0, 1);
res[j * n + i] = tmp;
res[i * n + j] = tmp;
}
}
putRNGstate();
UNPROTECT(1);
return R_res;
}
这段代码还没有被优化,因为在最内层循环中使用整数乘法 j * n + i 来寻址将导致性能损失。但我相信您可以将乘法移到内层循环之外,只留下加法。
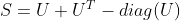
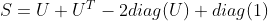
bigmemory包中的big.matrix吗?这可能是绕过内存限制的一种方法。 - konvassparseMatrix(i = sequence(1:99), j = rep(2:100, 1:99), x = res.upper, symmetric = TRUE, dims = c(100, 100)),避免了多次复制和使用(可能)更小的对象。 - alexis_laz