另一种选择是使用
to_records(),它比
itertuples和
iterrows都要快。
但对于您的情况,还有很多其他类型的改进空间。
这是我的最终优化版本。
def iterthrough():
ret = []
grouped = table2.groupby('letter', sort=False)
t2info = table2.to_records()
for index, letter, n1 in table1.to_records():
t2 = t2info[grouped.groups[letter].values]
maxrow = np.multiply(t2.number2, n1).argmax()
ret.append(t2[maxrow].tolist()[1:])
global table3
table3 = pd.DataFrame(ret, columns=('letter', 'number2'))
基准测试:
-- iterrows() --
100 loops, best of 3: 12.7 ms per loop
letter number2
0 a 0.5
1 b 0.1
2 c 5.0
3 d 4.0
-- itertuple() --
100 loops, best of 3: 12.3 ms per loop
-- to_records() --
100 loops, best of 3: 7.29 ms per loop
-- Use group by --
100 loops, best of 3: 4.07 ms per loop
letter number2
1 a 0.5
2 b 0.1
4 c 5.0
5 d 4.0
-- Avoid multiplication --
1000 loops, best of 3: 1.39 ms per loop
letter number2
0 a 0.5
1 b 0.1
2 c 5.0
3 d 4.0
完整代码:
import pandas as pd
import numpy as np
t1 = {'letter':['a','b','c','d'],
'number1':[50,-10,.5,3]}
t2 = {'letter':['a','a','b','b','c','d','c'],
'number2':[0.2,0.5,0.1,0.4,5,4,1]}
table1 = pd.DataFrame(t1)
table2 = pd.DataFrame(t2)
table3 = pd.DataFrame(np.nan, columns=['letter','number2'], index=table1.index)
print('\n-- iterrows() --')
def optimize(t2info, t1info):
calculation = []
for index, r in t2info.iterrows():
calculation.append(r['number2'] * t1info)
maxrow_in_t2 = calculation.index(max(calculation))
return t2info.loc[maxrow_in_t2]
def iterthrough():
for row_index, row in table1.iterrows():
t2info = table2[table2.letter == row['letter']].reset_index()
table3.iloc[row_index,:] = optimize(t2info, row['number1'])
%timeit iterthrough()
print(table3)
print('\n-- itertuple() --')
def optimize(t2info, n1):
calculation = []
for index, letter, n2 in t2info.itertuples():
calculation.append(n2 * n1)
maxrow = calculation.index(max(calculation))
return t2info.iloc[maxrow]
def iterthrough():
for row_index, letter, n1 in table1.itertuples():
t2info = table2[table2.letter == letter]
table3.iloc[row_index,:] = optimize(t2info, n1)
%timeit iterthrough()
print('\n-- to_records() --')
def optimize(t2info, n1):
calculation = []
for index, letter, n2 in t2info.to_records():
calculation.append(n2 * n1)
maxrow = calculation.index(max(calculation))
return t2info.iloc[maxrow]
def iterthrough():
for row_index, letter, n1 in table1.to_records():
t2info = table2[table2.letter == letter]
table3.iloc[row_index,:] = optimize(t2info, n1)
%timeit iterthrough()
print('\n-- Use group by --')
def iterthrough():
ret = []
grouped = table2.groupby('letter', sort=False)
for index, letter, n1 in table1.to_records():
t2 = table2.iloc[grouped.groups[letter]]
calculation = t2.number2 * n1
maxrow = calculation.argsort().iloc[-1]
ret.append(t2.iloc[maxrow])
global table3
table3 = pd.DataFrame(ret)
%timeit iterthrough()
print(table3)
print('\n-- Even Faster --')
def iterthrough():
ret = []
grouped = table2.groupby('letter', sort=False)
t2info = table2.to_records()
for index, letter, n1 in table1.to_records():
t2 = t2info[grouped.groups[letter].values]
maxrow = np.multiply(t2.number2, n1).argmax()
ret.append(t2[maxrow].tolist()[1:])
global table3
table3 = pd.DataFrame(ret, columns=('letter', 'number2'))
%timeit iterthrough()
print(table3)
最终版本比原始代码快了近10倍。策略如下:
- 使用
groupby 来避免重复比较数值。
- 使用
to_records 来访问原始的 numpy.records 对象。
- 在编译所有数据之前不要操作 DataFrame。
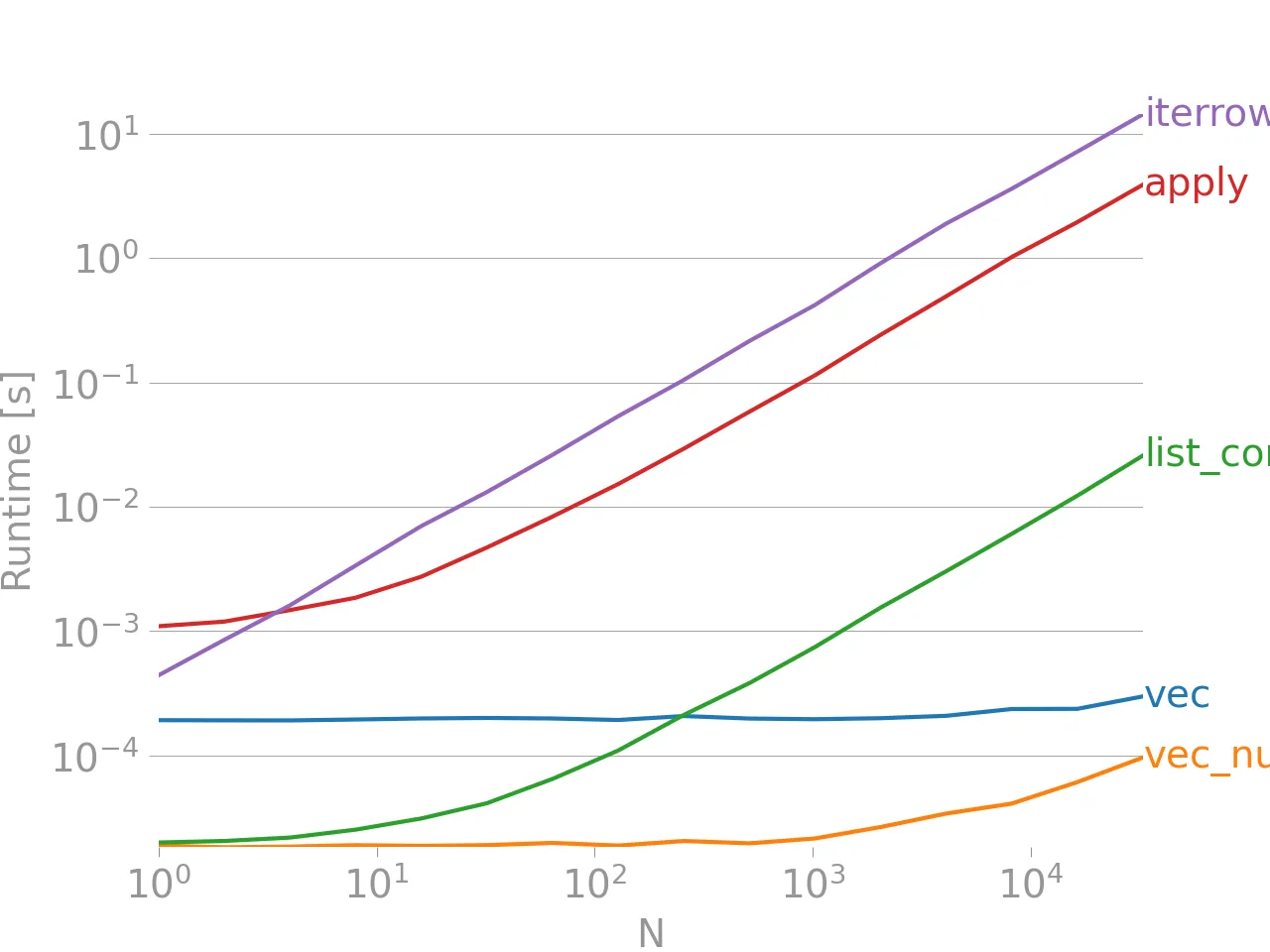
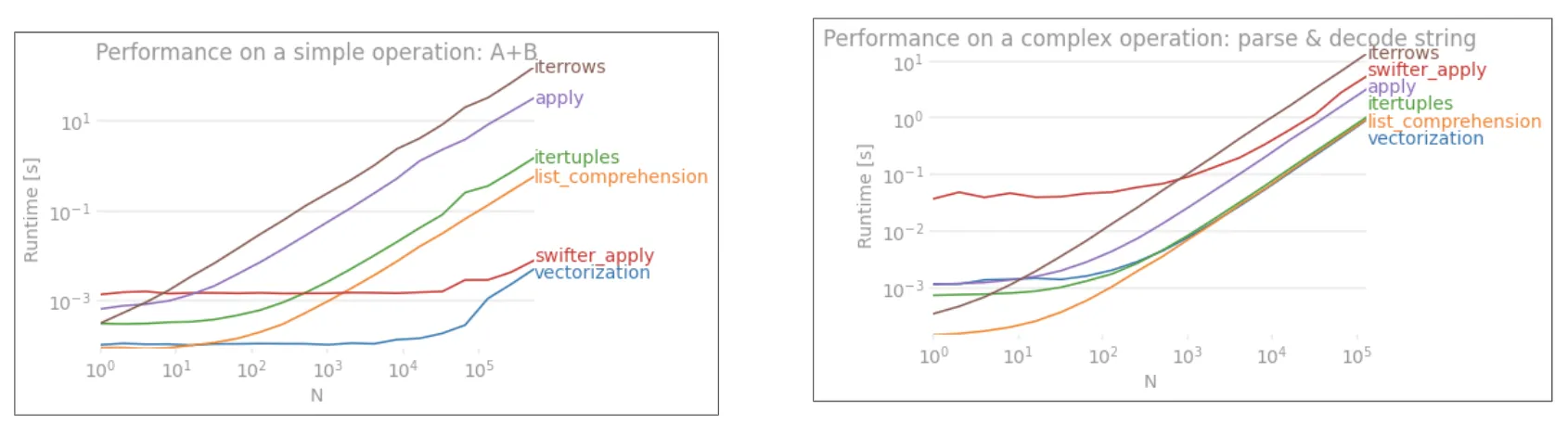
apply不支持向量化。iterrows更糟,因为它会把所有东西都装箱处理(这就是与“apply”相比的性能差异所在)。你应该只在非常少的情况下使用iterrows。在我看来,甚至从未使用。请展示一下你实际上是如何使用iterrows的。 - Jeff.iterrows()性能的讨论这里和我的答案。 - undefined