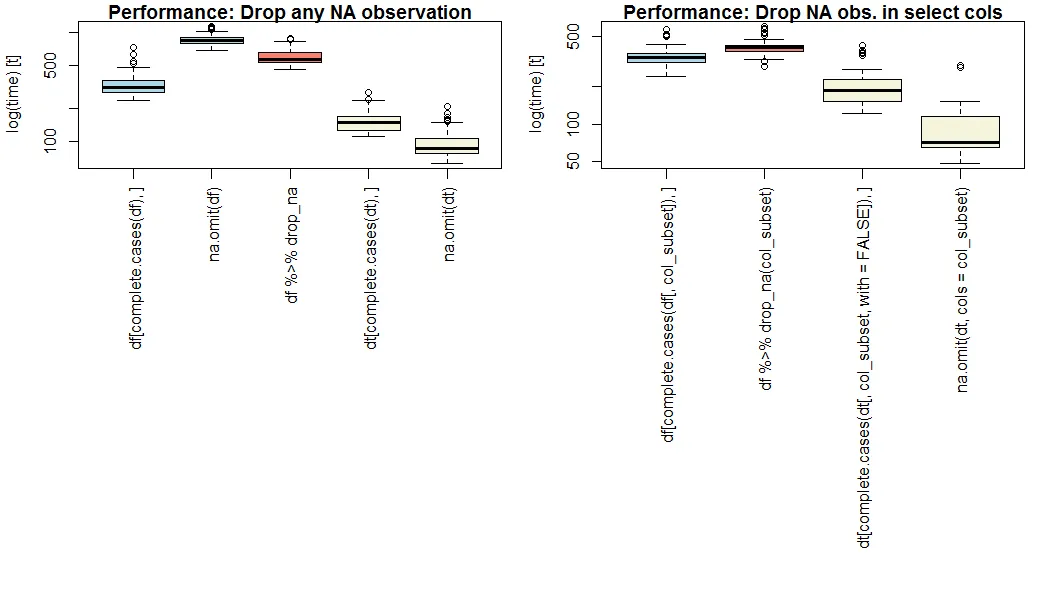
如果性能是优先考虑的因素,请使用 data.table 和 na.omit(),并选择可选参数 cols=。
na.omit.data.table 在我的基准测试中是最快的(请参见下文),无论是针对所有列还是选择列(OP问题第二部分)。
如果您不想使用 data.table,请使用 complete.cases()。
在一个原始的 data.frame 上,complete.cases 比 na.omit() 或者 dplyr::drop_na() 更快。注意,na.omit.data.frame 不支持 cols=。
基准测试结果
这里对于删除所有或选择性缺失观测的基本(蓝色)、dplyr(粉色)和 data.table(黄色)方法进行了比较,使用一个包含 20 个数值变量、独立 5% 的缺失可能性和 4 个变量的子集的 100 万观测量级数据集。
您的结果可能会根据数据集的长度、宽度和稀疏程度而有所不同。
请注意 y 轴上的对数刻度。
基准测试脚本
row_size <- 1e6L
col_size <- 20
p_missing <- 0.05
col_subset <- 18:21
R.version
library(data.table); packageVersion('data.table')
library(dplyr); packageVersion('dplyr')
library(tidyr); packageVersion('tidyr')
library(microbenchmark)
fakeData <- function(m, n, p){
set.seed(123)
m <- matrix(runif(m*n), nrow=m, ncol=n)
m[m<p] <- NA
return(m)
}
df <- cbind( data.frame(id = paste0('ID',seq(row_size)),
stringsAsFactors = FALSE),
data.frame(fakeData(row_size, col_size, p_missing) )
)
dt <- data.table(df)
par(las=3, mfcol=c(1,2), mar=c(22,4,1,1)+0.1)
boxplot(
microbenchmark(
df[complete.cases(df), ],
na.omit(df),
df %>% drop_na,
dt[complete.cases(dt), ],
na.omit(dt)
), xlab='',
main = 'Performance: Drop any NA observation',
col=c(rep('lightblue',2),'salmon',rep('beige',2))
)
boxplot(
microbenchmark(
df[complete.cases(df[,col_subset]), ],
df %>% drop_na(col_subset),
dt[complete.cases(dt[,col_subset,with=FALSE]), ],
na.omit(dt, cols=col_subset)
), xlab='',
main = 'Performance: Drop NA obs. in select cols',
col=c('lightblue','salmon',rep('beige',2))
)

final[complete.cases(final),]中,末尾逗号的意义是什么? - hertzsprungcomplete.cases(final)返回一个布尔值,其中包含没有NA的行,例如(TRUE, FALSE, TRUE)。逗号后面表示所有列。因此,在逗号之前,您可以对行进行过滤,但在逗号之后,您不需要进行过滤,并要求返回所有内容。 - Kaycomplete.cases语句来指定列。 - Sandyna.omit将删除包含至少一个NA的行(即,只要一列为NA,该行就会被删除)。对于complete.cases也是如此:如果您传递的一列或多列中有NA,则它将返回FALSE。 - robertspierre