问题:
我对我的Python程序进行了详细的分析,有一个函数使得整个程序变得缓慢。它大量使用了Python字典,所以我可能没有最好地使用它们。如果我无法让它运行得更快,我将不得不用C++重新编写它,那么有谁能帮助我优化Python代码呢?
我希望我已经给出了正确的解释,并且您可以理解我的代码!非常感谢您提供任何帮助。
我的代码:
这是有问题的函数,使用line_profiler和kernprof进行了分析。我正在运行Python 2.7
我特别困惑于像363、389和405行这样的地方,其中if语句与两个变量的比较似乎需要很长时间。
我考虑过使用NumPy(因为它可以处理稀疏矩阵),但我认为它不适用于以下原因:(1)我没有使用整数索引矩阵(我使用对象实例);(2)我没有在矩阵中存储简单的数据类型(我存储了一个浮点数和一个对象实例的元组)。但我愿意接受关于NumPy的说服。如果有人了解NumPy的稀疏矩阵性能与Python哈希表的比较,我会很感兴趣。
对不起,我没有提供一个简单的示例供您测试,因为这个函数涉及到一个更大的项目,而且我无法想出如何设置一个简单的示例来测试它,而不给您我的代码库的一半!
Timer unit: 3.33366e-10 s
File: routing_distances.py
Function: propagate_distances_node at line 328
Total time: 807.234 s
Line # Hits Time Per Hit % Time Line Contents
328 @profile
329 def propagate_distances_node(self, node_a, cutoff_distance=200):
330
331 # a makes sure its immediate neighbours are correctly in its distance table
332 # because its immediate neighbours may change as binds/folding change
333 737753 3733642341 5060.8 0.2 for (node_b, neighbour_distance_b_a) in self.neighbours[node_a].iteritems():
334 512120 2077788924 4057.2 0.1 use_neighbour_link = False
335
336 512120 2465798454 4814.9 0.1 if(node_b not in self.node_distances[node_a]): # a doesn't know distance to b
337 15857 66075687 4167.0 0.0 use_neighbour_link = True
338 else: # a does know distance to b
339 496263 2390534838 4817.1 0.1 (node_distance_b_a, next_node) = self.node_distances[node_a][node_b]
340 496263 2058112872 4147.2 0.1 if(node_distance_b_a > neighbour_distance_b_a): # neighbour distance is shorter
341 81 331794 4096.2 0.0 use_neighbour_link = True
342 496182 2665644192 5372.3 0.1 elif((None == next_node) and (float('+inf') == neighbour_distance_b_a)): # direct route that has just broken
343 75 313623 4181.6 0.0 use_neighbour_link = True
344
345 512120 1992514932 3890.7 0.1 if(use_neighbour_link):
346 16013 78149007 4880.3 0.0 self.node_distances[node_a][node_b] = (neighbour_distance_b_a, None)
347 16013 83489949 5213.9 0.0 self.nodes_changed.add(node_a)
348
349 ## Affinity distances update
350 16013 86020794 5371.9 0.0 if((node_a.type == Atom.BINDING_SITE) and (node_b.type == Atom.BINDING_SITE)):
351 164 3950487 24088.3 0.0 self.add_affinityDistance(node_a, node_b, self.chemistry.affinity(node_a.data, node_b.data))
352
353 # a sends its table to all its immediate neighbours
354 737753 3549685140 4811.5 0.1 for (node_b, neighbour_distance_b_a) in self.neighbours[node_a].iteritems():
355 512120 2129343210 4157.9 0.1 node_b_changed = False
356
357 # b integrates a's distance table with its own
358 512120 2203821081 4303.3 0.1 node_b_chemical = node_b.chemical
359 512120 2409257898 4704.5 0.1 node_b_distances = node_b_chemical.node_distances[node_b]
360
361 # For all b's routes (to c) that go to a first, update their distances
362 41756882 183992040153 4406.3 7.6 for node_c, (distance_b_c, node_after_b) in node_b_distances.iteritems(): # Think it's ok to modify items while iterating over them (just not insert/delete) (seems to work ok)
363 41244762 172425596985 4180.5 7.1 if(node_after_b == node_a):
364
365 16673654 64255631616 3853.7 2.7 try:
366 16673654 88781802534 5324.7 3.7 distance_b_a_c = neighbour_distance_b_a + self.node_distances[node_a][node_c][0]
367 187083 929898684 4970.5 0.0 except KeyError:
368 187083 1056787479 5648.8 0.0 distance_b_a_c = float('+inf')
369
370 16673654 69374705256 4160.7 2.9 if(distance_b_c != distance_b_a_c): # a's distance to c has changed
371 710083 3136751361 4417.4 0.1 node_b_distances[node_c] = (distance_b_a_c, node_a)
372 710083 2848845276 4012.0 0.1 node_b_changed = True
373
374 ## Affinity distances update
375 710083 3484577241 4907.3 0.1 if((node_b.type == Atom.BINDING_SITE) and (node_c.type == Atom.BINDING_SITE)):
376 99592 1591029009 15975.5 0.1 node_b_chemical.add_affinityDistance(node_b, node_c, self.chemistry.affinity(node_b.data, node_c.data))
377
378 # If distance got longer, then ask b's neighbours to update
379 ## TODO: document this!
380 16673654 70998570837 4258.1 2.9 if(distance_b_a_c > distance_b_c):
381 #for (node, neighbour_distance) in node_b_chemical.neighbours[node_b].iteritems():
382 1702852 7413182064 4353.4 0.3 for node in node_b_chemical.neighbours[node_b]:
383 1204903 5912053272 4906.7 0.2 node.chemical.nodes_changed.add(node)
384
385 # Look for routes from a to c that are quicker than ones b knows already
386 42076729 184216680432 4378.1 7.6 for node_c, (distance_a_c, node_after_a) in self.node_distances[node_a].iteritems():
387
388 41564609 171150289218 4117.7 7.1 node_b_update = False
389 41564609 172040284089 4139.1 7.1 if(node_c == node_b): # a-b path
390 512120 2040112548 3983.7 0.1 pass
391 41052489 169406668962 4126.6 7.0 elif(node_after_a == node_b): # a-b-a-b path
392 16251407 63918804600 3933.1 2.6 pass
393 24801082 101577038778 4095.7 4.2 elif(node_c in node_b_distances): # b can already get to c
394 24004846 103404357180 4307.6 4.3 (distance_b_c, node_after_b) = node_b_distances[node_c]
395 24004846 102717271836 4279.0 4.2 if(node_after_b != node_a): # b doesn't already go to a first
396 7518275 31858204500 4237.4 1.3 distance_b_a_c = neighbour_distance_b_a + distance_a_c
397 7518275 33470022717 4451.8 1.4 if(distance_b_a_c < distance_b_c): # quicker to go via a
398 225357 956440656 4244.1 0.0 node_b_update = True
399 else: # b can't already get to c
400 796236 3415455549 4289.5 0.1 distance_b_a_c = neighbour_distance_b_a + distance_a_c
401 796236 3412145520 4285.3 0.1 if(distance_b_a_c < cutoff_distance): # not too for to go
402 593352 2514800052 4238.3 0.1 node_b_update = True
403
404 ## Affinity distances update
405 41564609 164585250189 3959.7 6.8 if node_b_update:
406 818709 3933555120 4804.6 0.2 node_b_distances[node_c] = (distance_b_a_c, node_a)
407 818709 4151464335 5070.7 0.2 if((node_b.type == Atom.BINDING_SITE) and (node_c.type == Atom.BINDING_SITE)):
408 104293 1704446289 16342.9 0.1 node_b_chemical.add_affinityDistance(node_b, node_c, self.chemistry.affinity(node_b.data, node_c.data))
409 818709 3557529531 4345.3 0.1 node_b_changed = True
410
411 # If any of node b's rows have exceeded the cutoff distance, then remove them
412 42350234 197075504439 4653.5 8.1 for node_c, (distance_b_c, node_after_b) in node_b_distances.items(): # Can't use iteritems() here, as deleting from the dictionary
413 41838114 180297579789 4309.4 7.4 if(distance_b_c > cutoff_distance):
414 206296 894881754 4337.9 0.0 del node_b_distances[node_c]
415 206296 860508045 4171.2 0.0 node_b_changed = True
416
417 ## Affinity distances update
418 206296 4698692217 22776.5 0.2 node_b_chemical.del_affinityDistance(node_b, node_c)
419
420 # If we've modified node_b's distance table, tell its chemical to update accordingly
421 512120 2130466347 4160.1 0.1 if(node_b_changed):
422 217858 1201064454 5513.1 0.0 node_b_chemical.nodes_changed.add(node_b)
423
424 # Remove any neighbours that have infinite distance (have just unbound)
425 ## TODO: not sure what difference it makes to do this here rather than above (after updating self.node_distances for neighbours)
426 ## but doing it above seems to break the walker's movement
427 737753 3830386968 5192.0 0.2 for (node_b, neighbour_distance_b_a) in self.neighbours[node_a].items(): # Can't use iteritems() here, as deleting from the dictionary
428 512120 2249770068 4393.1 0.1 if(neighbour_distance_b_a > cutoff_distance):
429 150 747747 4985.0 0.0 del self.neighbours[node_a][node_b]
430
431 ## Affinity distances update
432 150 2148813 14325.4 0.0 self.del_affinityDistance(node_a, node_b)
我的代码解释:
这个函数维护了一个稀疏距离矩阵,表示(最短路径上的边权重之和)节点之间的网络距离在一个(非常大的)网络中。使用Floyd-Warshall算法处理完整表格会非常慢。(我先尝试了这个方法,但比当前版本慢了数个数量级。)因此,我的代码使用稀疏矩阵来表示完整距离矩阵的阈值版本(忽略距离大于200个单位的任何路径)。网络拓扑随时间变化,因此这个距离矩阵需要随时间更新。为了实现这一点,我使用了一个距离向量路由协议的简单实现:网络中的每个节点都知道到每个其他节点的距离和路径上的下一个节点。当发生拓扑变化时,与该变化相关联的节点相应地更新其距离表,并告诉它们的直接邻居。信息通过节点将它们的距离表发送给邻居而在网络中传播,邻居更新它们的距离表并将它们传播给它们的邻居。
有一个代表距离矩阵的对象:self.node_distances。这是一个将节点映射到路由表的字典。节点是我定义的一个对象。路由表是一个将节点映射到元组(距离,下一个节点)的字典。距离是从节点a到节点b的图距离,下一个节点是节点a和节点b之间路径上必须先到达的邻居节点。下一个节点为None表示节点a和节点b是图中的邻居。例如,距离矩阵的示例可能如下:
self.node_distances = { node_1 : { node_2 : (2.0, None),
node_3 : (5.7, node_2),
node_5 : (22.9, node_2) },
node_2 : { node_1 : (2.0, None),
node_3 : (3.7, None),
node_5 : (20.9, node_7)},
...etc...
由于拓扑变化,原本相距较远的两个节点(或根本未连接)可能会靠近。当发生这种情况时,此矩阵中会添加条目。由于阈值,两个节点可能会相距太远而不再重要。当发生这种情况时,此矩阵中的条目将被删除。
self.neighbours矩阵类似于self.node_distances,但包含有关网络中直接链接(边缘)的信息。self.neighbours在该函数外部不断进行修改,由化学反应引起网络拓扑变化。
实际上我遇到问题的函数:propagate_distances_node()执行距离向量路由协议的一步。给定一个节点node_a,该函数确保node_a的邻居在距离矩阵中正确地表示(拓扑变化)。然后,该函数将node_a的路由表发送到其在网络中的所有直接邻居。它将node_a的路由表与每个邻居自己的路由表集成。
在我的程序的其余部分中,将重复调用
propagate_distances_node()函数,直到距离矩阵收敛。维护一个节点集self.nodes_changed,其中包含自它们上次更新后已更改其路由表的节点。在算法的每次迭代中,会选择这些节点的随机子集,并对它们调用propagate_distances_node()。这意味着节点异步和随机地传播其路由表。当集合self.nodes_changed变为空时,此算法收敛于真实的距离矩阵。"亲和距离"部分(
add_affinityDistance和del_affinityDistance)是距离矩阵的一个(小)子矩阵的缓存,被程序的另一部分使用。The reason I'm doing this is that I'm simulating computational analogues of chemicals participating in reactions, as part of my PhD. A "chemical" is a graph of "atoms" (nodes in the graph). Two chemicals binding together is simulated as their two graphs being joined by new edges. A chemical reaction happens (by a complicated process that isn't relevant here), changing the topology of the graph. But what happens in the reaction depends on how far apart the different atoms are that make up the chemicals. So for each atom in the simulation, I want to know which other atoms it is close to. A sparse, thresholded distance matrix is the most efficient way to store this information. Since the topology of the network changes as the reaction happens, I need to update the matrix. A 距离向量路由协议 is the fastest way I could come up with of doing this. I don't need a more compliacted routing protocol, because things like routing loops don't happen in my particular application (because of how my chemicals are structured). The reason I'm doing it stochastically is so that I can interleve the chemical reaction processes with the distance spreading, and simulate a chemical gradually changing shape over time as the reaction happens (rather than changing shape instantly).
The `self` in this function is an object representing a chemical. The nodes in `self.node_distances.keys()` are the atoms that make up the chemical. The nodes in `self.node_distances[node_x].keys()` are nodes from the chemical and potentially nodes from any chemicals that the chemical is bound to (and reacting with).
Update:
我尝试将每个
node_x == node_y的实例替换为node_x is node_y(根据@Sven Marnach的评论),但是它减缓了速度!(我没有预料到这一点!)我的原始配置文件运行需要807.234秒,但是使用此修改后,时间增加到895.895秒。很抱歉,我对性能分析做错了!我正在逐行查看,这在我的代码中有太多差异(约90秒的差异都在噪声中)。正确进行性能分析时,is明显比==更快。使用CProfile,我的代码使用==需要34.394秒,但是使用is只需要33.535秒(我可以确认这已经超出了噪声范围)。
更新:
现有库我不确定是否有现有的库可以满足我的要求,因为我的需求很特殊:我需要计算加权、无向图中所有节点对之间的最短路径长度。我只关心低于阈值的路径长度。计算路径长度后,我会对网络拓扑进行微小的更改(添加或删除边),然后我想重新计算路径长度。与阈值相比,我的图形巨大(从给定节点开始,大部分图形距离阈值更远),所以拓扑变化不会影响大部分最短路径长度。这就是我使用路由算法的原因:因为它通过图形结构传播拓扑变化信息,所以当其传播超出阈值时,我可以停止传播。也就是说,我不需要每次都重新计算所有路径。我可以使用先前的路径信息(在拓扑变化之前)来加速计算。这就是为什么我认为我的算法将比任何最短路径算法的库实现更快的原因。 我从未见过路由算法在物理网络之外使用(但如果有人使用过,我会感兴趣)。
NetworkX被@Thomas K.建议使用。它有很多算法可以计算最短路径。它有一种算法可以计算带有截止点的所有对最短路径长度(这就是我想要的),但它只适用于无权图(我的是加权的)。不幸的是,它的加权图算法不允许使用截止点(这可能会使它们在我的图上运行缓慢)。而且它的算法似乎都不支持在非常相似的网络上使用预先计算的路径(即路由部分)。
igraph是我知道的另一个图形库,但是看了它的文档后,我没有找到关于最短路径的任何信息。但我可能错过了它 - 它的文档似乎不太全面。
NumPy也许是可能的,感谢@9000的评论。如果我给每个节点实例分配一个唯一的整数,我就可以将稀疏矩阵存储在NumPy数组中。然后,我可以使用整数而不是节点实例来索引NumPy数组。我还需要两个NumPy数组:一个用于距离,一个用于“next_node”引用。这可能比使用Python字典更快(我还不知道)。
还有其他有用的库吗?
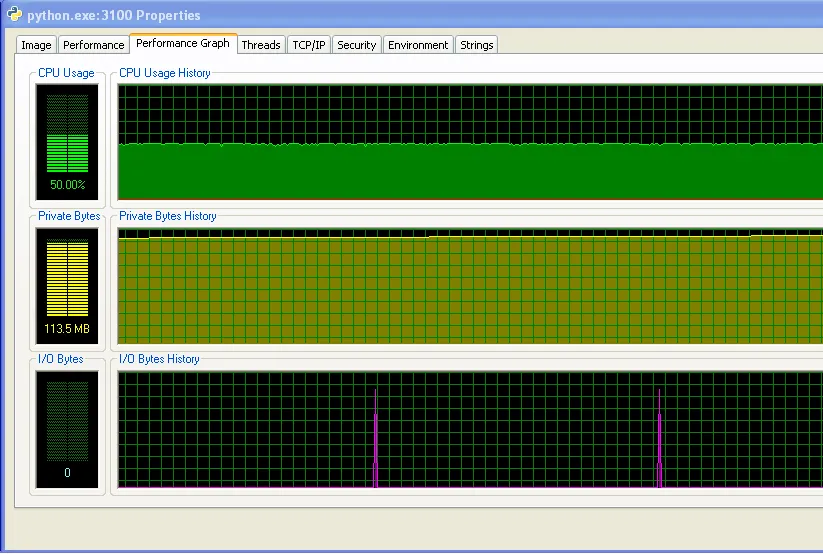
更新:内存使用情况
我正在运行Windows(XP),因此这里有一些有关内存使用情况的信息,来自Process Explorer。CPU使用率为50%,因为我有一台双核机器。
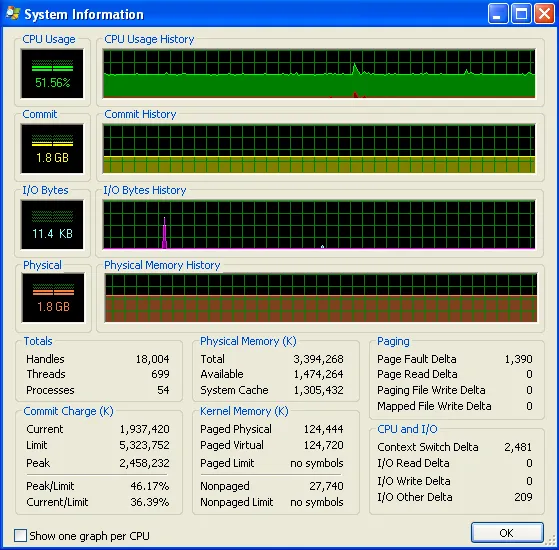
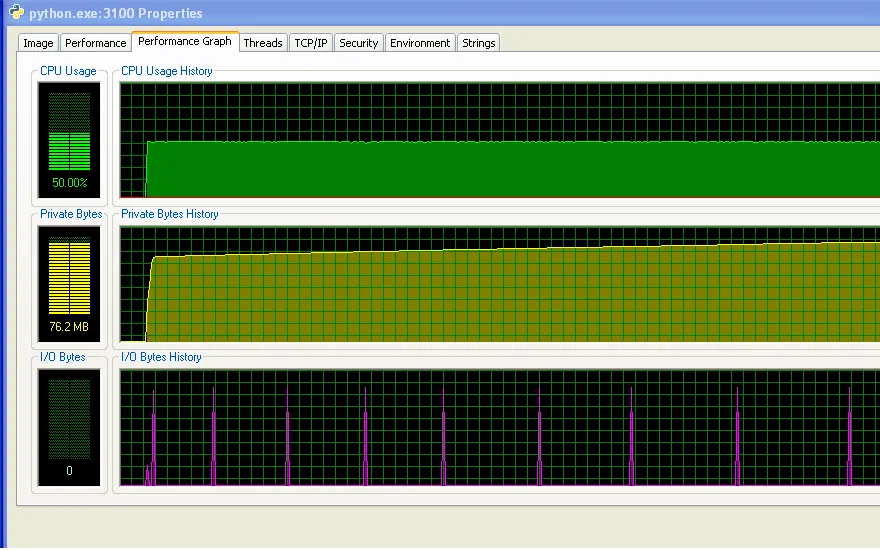
然而,我的程序随着时间的推移不断占用更多的RAM,这可能不是一个好现象(但总体上它没有占用太多的RAM,这就是为什么我直到现在才注意到增加)。
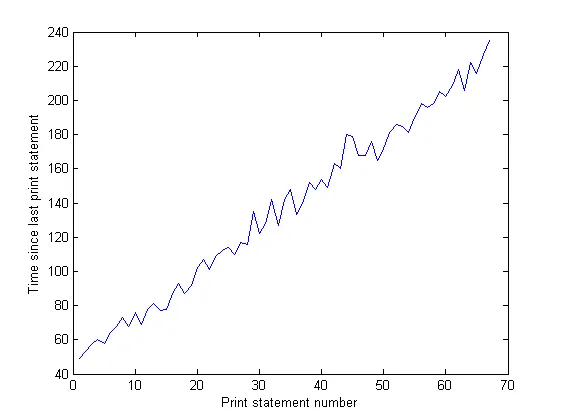
IO图上尖峰之间的距离随着时间的推移而增加。这是不好的——我的程序每100,000次迭代打印一次屏幕,这意味着随着时间的推移,每次迭代的执行时间都会变长……我通过运行程序并测量打印语句之间的时间(程序每10,000次迭代之间的时间)来确认这一点。这应该是恒定的,但是从图表中可以看出,它线性增加了……所以这里有些问题。(此图表的噪声是因为我的程序使用大量随机数,因此每次迭代的时间不同。)
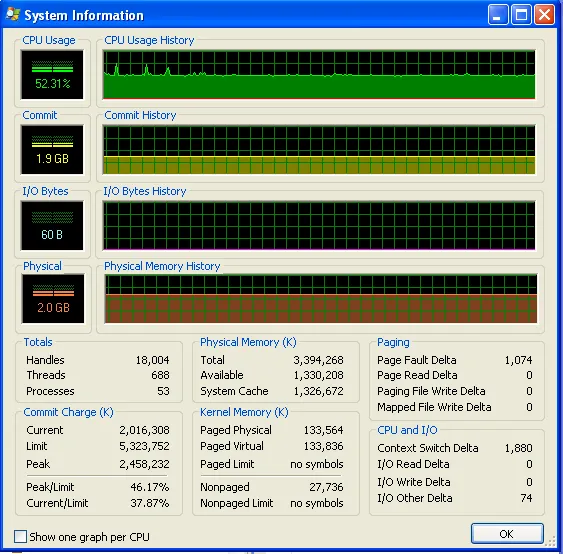
我的程序运行了很长时间后,内存使用情况如下(因此肯定不会用完RAM):
==实际上调用了被比较对象的方法__eq__()。如果您只想知道对象标识,可以使用is-- 这会更快。 - Sven Marnach__eq __()),并计算了像a == b,a is b和a is a的时间。对我而言,is比==速度快大约25%。我无法想象任何理由会导致自定义类的实例反过来操作。这两个操作符之间唯一的区别是==首先在实例字典中查找__eq __(),然后回退到与is执行相同的操作,而查找需要一些时间。 - Sven Marnach