我正在尝试重新创建这个图形(来自维基百科):
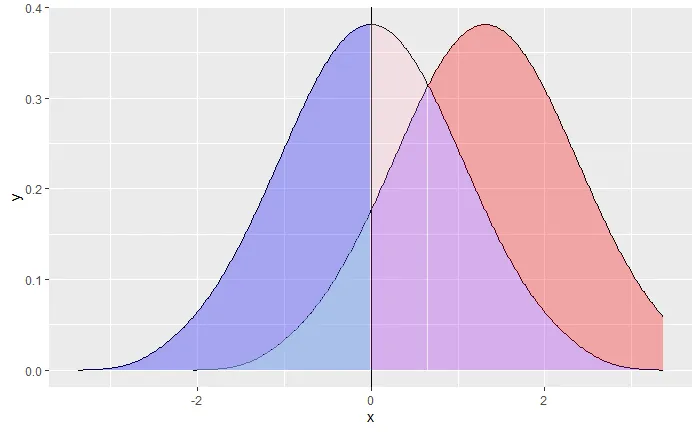
这是我目前的进展:
这让我来到这里: 我的使用
data <- distribution_normal(n = 100, mean = 0, sd = 1) %>%
density() %>%
as.data.frame() %>%
mutate(e = lag(y, 100))
ggplot(data) +
geom_line(aes(x=x, y=y)) +
geom_line(aes(x=x, y=e)) +
geom_vline(aes(xintercept=0)) +
geom_ribbon(data = subset(data, x<0), aes(x=x, ymin=0, ymax=y), fill = "blue", alpha = .3) +
geom_ribbon(data = subset(data, e<y & x<0), aes(x=x, ymin=0, ymax=e), fill = "light blue", alpha = .5) +
geom_ribbon(data = subset(data, y>e & x>0), aes(x=x, ymin=e, ymax=y), fill = "pink", alpha = .3) +
geom_ribbon(data = subset(data, e<y & x>0), aes(x=x, ymin=0, ymax=e), fill = "purple", alpha = .3) +
geom_ribbon(data = subset(data, y<=e & x>0), aes(x=x, ymin=0, ymax=y), fill = "purple", alpha = .3) +
geom_ribbon(data = subset(data, e>y & x>0), aes(x=x, ymin=y, ymax=e), fill = "red", alpha = .3)
这让我来到这里: 我的使用
lag的方法可以使第二条曲线的尾部被切掉。 有没有更好的方法可以让我展示完整的尾部? 我还需要解决下面紫色部分的不一致性问题,在两条曲线相交处和x=0处左侧。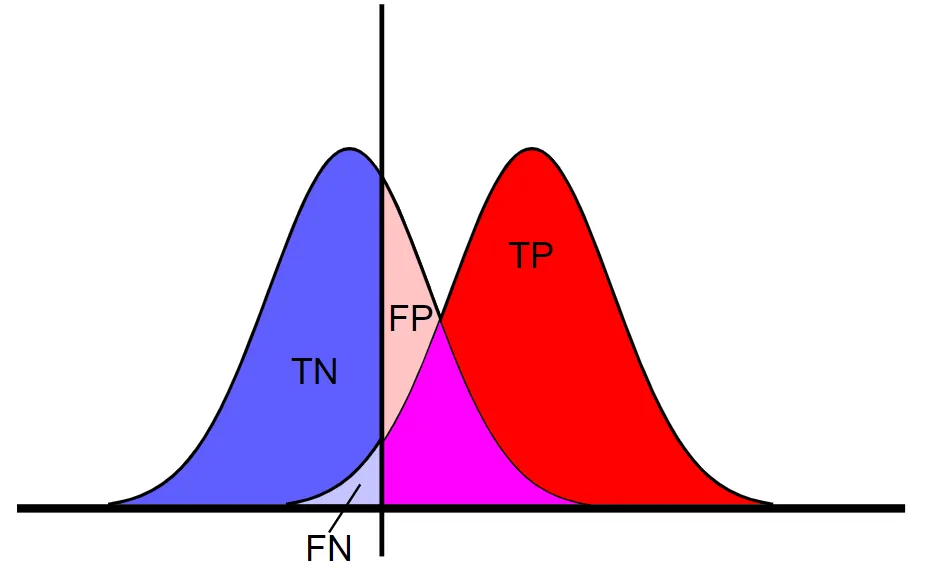
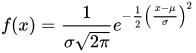
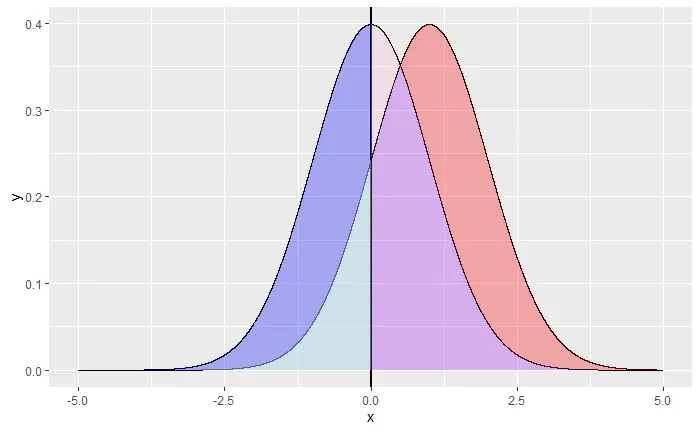
geom_ribbon中的跳过的区域。请看下面的回答。 - undefined