我想要标准化下面表示的概率密度函数Norm1。当我直接将该方程式导入到ax.contour时,它可以正常工作,但在调用该方程式外部时却不行。
当我尝试将Norm1传递给ax.contour时,我收到以下错误:
raise TypeError(f"Input z must be 2D, not {z.ndim}D")
TypeError: Input z must be 2D, not 3D
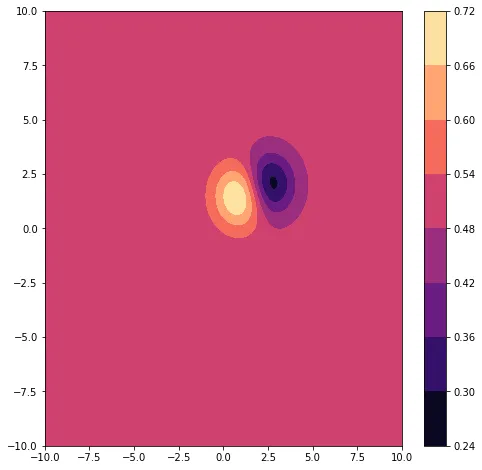
为了提供背景,我有两个不同的组,其中包含各种XY点。每个XY点都有一个标识符,标记为id。我为每个唯一点分配一个圆或半径。我想要从每个组中减去PDF。输出是一个从0到1归一化的PDF,其中>0.5由组A控制,<0.5由组B控制。
df = pd.DataFrame({'Int_1': [1.0, 2.0, 1.0, 3.0, 1.0, 2.0, 3.0, 2.0],
'Int_2': [1.0, 2.0, 2.0, 2.0, 1.0, 1.0, 1.0, 2.0],
'Item_X': [0.0, 0.0, 0.0, 0.0, 1.0, 1.0, 1.0, 1.0],
'Item_Y': [0.0, 0.0, 0.0, 0.0, 1.0, 1.0, 1.0, 1.0],
'Period': [1, 1, 1, 1, 2, 2, 2, 2],
'Group': ['A', 'B', 'A', 'B', 'A', 'B', 'A', 'B'],
'Item': ['Y', 'Y', 'A', 'B', 'A', 'B', 'A', 'B'],
'id': ['1', '2', '3', '4', '1', '2', '3', '4']})
Group_A = [df[df['Group'] == 'A'][['Int_1','Int_2']].to_numpy()]
Group_B = [df[df['Group'] == 'B'][['Int_1','Int_2']].to_numpy()]
Item = [df[['Item_X','Item_Y']].to_numpy()]
period = df['Period'].drop_duplicates().reset_index(drop = True)
def impact_func(member_no, location, time_index, group):
if group == 'A':
data = Group_A.copy()
elif group == 'B':
data = Group_B.copy()
else:
return
if np.all(np.isfinite(data[member_no][[time_index,time_index + 1],:])) & np.all(np.isfinite(Item[0][time_index,:])):
sxy = (data[member_no][time_index + 1,:] - data[member_no][time_index,:]) / (period[time_index + 1] - period[time_index])
mu = data[member_no][time_index,:] + sxy * 0.5
out = mvn.pdf(location,mu) / mvn.pdf(data[member_no][time_index,:],mu)
else:
out = np.zeros(location.shape[0])
return out
xx,yy = np.meshgrid(np.linspace(-10,10,200),np.linspace(-10,10,200))
Z_GA = np.zeros(40000)
Z_GB = np.zeros(40000)
for k in range(1):
Z_GA += impact_func(k,np.c_[xx.flatten(),yy.flatten()],0,'A')
Z_GB += impact_func(k,np.c_[xx.flatten(),yy.flatten()],0,'B')
fig, ax = plt.subplots(figsize=(8,8))
ax.set_xlim(-10,10)
ax.set_ylim(-10,10)
Z_GA = Z_GA.reshape((200,200))
Z_GB = Z_GB.reshape((200,200))
Norm1 = xx,yy, 1 / (1 + np.exp(Z_GB - Z_GA))
Norm2 = Z_GA / (Z_GA + Z_GB)
#cfs = ax.contourf(Norm1, cmap = 'magma')
cfs = ax.contourf(xx,yy, 1 / (1 + np.exp(Z_GB - Z_GA)), cmap = 'magma')
fig.colorbar(cfs, ax = ax)