我希望能够执行适合于将任意曲线函数拟合到数据的操作,并允许我对参数设置任意限制。例如,我想要拟合函数:
f(x) = a1(x-a2)^a3\cdot\exp(-\a4*x^a5)
并且说:
a2在以下范围内:(-1, 1)a3和a5是正数
有一个不错的scipy curve_fit函数,但它不允许指定参数范围。还有一个很好的http://code.google.com/p/pyminuit/库可以进行通用极小化,并且允许设置参数范围,但在我的情况下它没有收敛。
我希望能够执行适合于将任意曲线函数拟合到数据的操作,并允许我对参数设置任意限制。例如,我想要拟合函数:
f(x) = a1(x-a2)^a3\cdot\exp(-\a4*x^a5)
并且说:
a2 在以下范围内:(-1, 1)a3 和 a5 是正数有一个不错的scipy curve_fit函数,但它不允许指定参数范围。还有一个很好的http://code.google.com/p/pyminuit/库可以进行通用极小化,并且允许设置参数范围,但在我的情况下它没有收敛。
y=a*t**alpha+b
并且在alpha的限制条件下
0<alpha<2
当其他参数a和b保持自由时,我们应该按照以下方式使用curve_fit的bounds选项:
import numpy as np
from scipy.optimize import curve_fit
def func(t, a,alpha,b):
return a*t**alpha+b
param_bounds=([-np.inf,0,-np.inf],[np.inf,2,np.inf])
popt, pcov = curve_fit(func, xdata, ydata,bounds=param_bounds)
源代码在这里。
如Rob Falck所述,您可以使用例如scipy.minimize中的scipy非线性优化程序来最小化任意误差函数,例如均方误差。
请注意,您提供的函数不一定具有实值 - 也许这就是您在pyminuit中的最小化未收敛的原因。您必须更加明确地处理此问题,参见示例2。
以下示例都使用支持有界参数区域的L-BFGS-B最小化方法。我将此答案分为两部分:
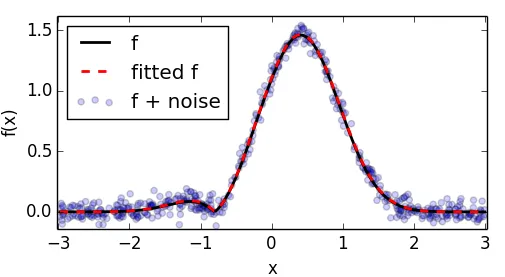
下面的示例显示了对您的函数稍作修改后进行的优化。
import numpy as np
import pylab as pl
from scipy.optimize import minimize
points = 500
xlim = 3.
def f(x,*p):
a1,a2,a3,a4,a5 = p
return a1*np.abs(x-a2)**a3 * np.exp(-a4 * np.abs(x)**a5)
# generate noisy data with known coefficients
p0 = [1.4,-.8,1.1,1.2,2.2]
x = (np.random.rand(points) * 2. - 1.) * xlim
x.sort()
y = f(x,*p0)
y_noise = y + np.random.randn(points) * .05
# mean squared error wrt. noisy data as a function of the parameters
err = lambda p: np.mean((f(x,*p)-y_noise)**2)
# bounded optimization using scipy.minimize
p_init = [1.,-1.,.5,.5,2.]
p_opt = minimize(
err, # minimize wrt to the noisy data
p_init,
bounds=[(None,None),(-1,1),(None,None),(0,None),(None,None)], # set the bounds
method="L-BFGS-B" # this method supports bounds
).x
# plot everything
pl.scatter(x, y_noise, alpha=.2, label="f + noise")
pl.plot(x, y, c='#000000', lw=2., label="f")
pl.plot(x, f(x,*p_opt) ,'--', c='r', lw=2., label="fitted f")
pl.xlabel("x")
pl.ylabel("f(x)")
pl.legend(loc="best")
pl.xlim([-xlim*1.01,xlim*1.01])
pl.show()
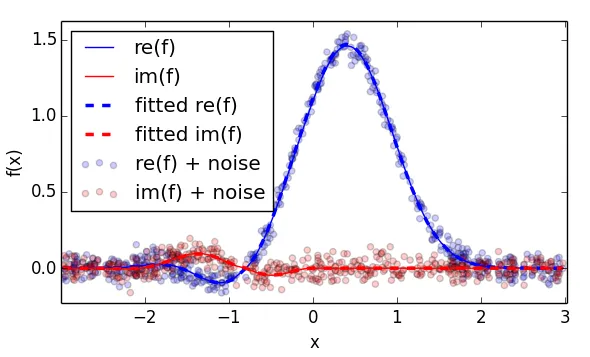
将上述最小化问题扩展到复数域可以通过显式转换为复数并调整误差函数来完成:
首先,您需要将值x显式转换为复数以确保f返回复杂值并且能够计算负数的分数幂。其次,我们在实部和虚部上计算某些误差函数 - 一个直接的候选是平方复数绝对值的平均值。
import numpy as np
import pylab as pl
from scipy.optimize import minimize
points = 500
xlim = 3.
def f(x,*p):
a1,a2,a3,a4,a5 = p
x = x.astype(complex) # cast x explicitly to complex, to ensure complex valued f
return a1*(x-a2)**a3 * np.exp(-a4 * x**a5)
# generate noisy data with known coefficients
p0 = [1.4,-.8,1.1,1.2,2.2]
x = (np.random.rand(points) * 2. - 1.) * xlim
x.sort()
y = f(x,*p0)
y_noise = y + np.random.randn(points) * .05 + np.random.randn(points) * 1j*.05
# error function chosen as mean of squared absolutes
err = lambda p: np.mean(np.abs(f(x,*p)-y_noise)**2)
# bounded optimization using scipy.minimize
p_init = [1.,-1.,.5,.5,2.]
p_opt = minimize(
err, # minimize wrt to the noisy data
p_init,
bounds=[(None,None),(-1,1),(None,None),(0,None),(None,None)], # set the bounds
method="L-BFGS-B" # this method supports bounds
).x
# plot everything
pl.scatter(x, np.real(y_noise), c='b',alpha=.2, label="re(f) + noise")
pl.scatter(x, np.imag(y_noise), c='r',alpha=.2, label="im(f) + noise")
pl.plot(x, np.real(y), c='b', lw=1., label="re(f)")
pl.plot(x, np.imag(y), c='r', lw=1., label="im(f)")
pl.plot(x, np.real(f(x,*p_opt)) ,'--', c='b', lw=2.5, label="fitted re(f)")
pl.plot(x, np.imag(f(x,*p_opt)) ,'--', c='r', lw=2.5, label="fitted im(f)")
pl.xlabel("x")
pl.ylabel("f(x)")
pl.legend(loc="best")
pl.xlim([-xlim*1.01,xlim*1.01])
pl.show()
看起来最小化器可能对初始值有些敏感 - 因此我将我的第一个猜测(p_init)放得离最优解不太远。如果你必须为此而奋斗,你可以使用相同的最小化过程加上全局优化循环,例如盆地跳跃或蛮力搜索。
xdata = np.array([177.,180.,183.,187.,189.,190.,196.,197.,201.,202.,203.,204.,206.,218.,225.,231.,234.,
252.,262.,266.,267.,268.,277.,286.,303.])
ydata = np.array([0.81,0.74,0.78,0.75,0.77,0.81,0.73,0.76,0.71,0.74,0.81,0.71,0.74,0.71,
0.72,0.69,0.75,0.59,0.61,0.63,0.64,0.63,0.35,0.27,0.26])
你想将模型拟合到这样的数据中:
model = n1 + (n2 * x + n3) * 1./ (1. + np.exp(n4 * (n5 - x)))
在约束条件下
0.2 < n1 < 0.8
-0.3 < n2 < 0
lmfit(版本0.8.3),您将获得以下输出:n1: 0.26564921 +/- 0.024765 (9.32%) (init= 0.2)
n2: -0.00195398 +/- 0.000311 (15.93%) (init=-0.005)
n3: 0.87261892 +/- 0.068601 (7.86%) (init= 1.0766)
n4: -1.43507072 +/- 1.223086 (85.23%) (init=-0.36379)
n5: 277.684530 +/- 3.768676 (1.36%) (init= 274)
正如您所看到的,拟合非常好地重现了数据,并且参数在请求的范围内。
以下是完整的代码,其中包含一些额外的注释以重现绘图:
from lmfit import minimize, Parameters, Parameter, report_fit
import numpy as np
xdata = np.array([177.,180.,183.,187.,189.,190.,196.,197.,201.,202.,203.,204.,206.,218.,225.,231.,234.,
252.,262.,266.,267.,268.,277.,286.,303.])
ydata = np.array([0.81,0.74,0.78,0.75,0.77,0.81,0.73,0.76,0.71,0.74,0.81,0.71,0.74,0.71,
0.72,0.69,0.75,0.59,0.61,0.63,0.64,0.63,0.35,0.27,0.26])
def fit_fc(params, x, data):
n1 = params['n1'].value
n2 = params['n2'].value
n3 = params['n3'].value
n4 = params['n4'].value
n5 = params['n5'].value
model = n1 + (n2 * x + n3) * 1./ (1. + np.exp(n4 * (n5 - x)))
return model - data #that's what you want to minimize
# create a set of Parameters
# 'value' is the initial condition
# 'min' and 'max' define your boundaries
params = Parameters()
params.add('n1', value= 0.2, min=0.2, max=0.8)
params.add('n2', value= -0.005, min=-0.3, max=10**(-10))
params.add('n3', value= 1.0766, min=-1000., max=1000.)
params.add('n4', value= -0.36379, min=-1000., max=1000.)
params.add('n5', value= 274.0, min=0., max=1000.)
# do fit, here with leastsq model
result = minimize(fit_fc, params, args=(xdata, ydata))
# write error report
report_fit(params)
xplot = np.linspace(min(xdata), max(xdata), 1000)
yplot = result.values['n1'] + (result.values['n2'] * xplot + result.values['n3']) * \
1./ (1. + np.exp(result.values['n4'] * (result.values['n5'] - xplot)))
#plot results
try:
import pylab
pylab.plot(xdata, ydata, 'k+')
pylab.plot(xplot, yplot, 'r')
pylab.show()
except:
pass
编辑:
如果您使用的是0.9.x版本,则需要相应地调整代码;请查看这里,了解从0.8.3到0.9.x所做的更改。
解决方法:使用变量转换,例如a2=tanh(a2')、a3=exp(a3')或a5=a5'^2。
您是否考虑将其视为优化问题,并使用scipy中的非线性优化例程通过变化函数系数来最小化最小二乘误差?optimize中的许多例程允许对自变量进行边界约束。