我有一些数据,想要根据Gap统计量来评估最佳聚类数。
我阅读了关于Gap统计量在R中的页面,其中给出了以下示例:
gs.pam.RU <- clusGap(ruspini, FUN = pam1, K.max = 8, B = 500)
gs.pam.RU
当我调用
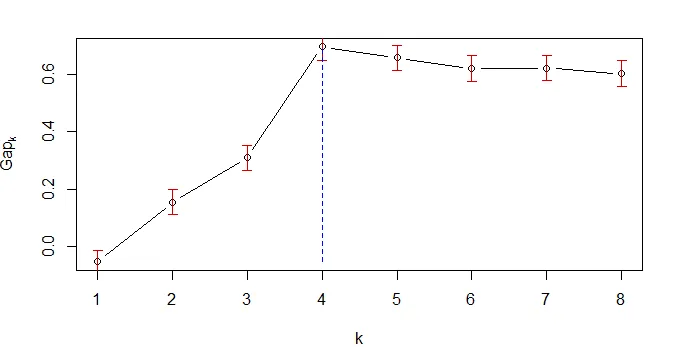
gs.pam.RU.Tab时,我得到:Clustering Gap statistic ["clusGap"].
B=500 simulated reference sets, k = 1..8
--> Number of clusters (method 'firstSEmax', SE.factor=1): 4
logW E.logW gap SE.sim
[1,] 7.187997 7.135307 -0.05268985 0.03729363
[2,] 6.628498 6.782815 0.15431689 0.04060489
[3,] 6.261660 6.569910 0.30825062 0.04296625
[4,] 5.692736 6.384584 0.69184777 0.04346588
[5,] 5.580999 6.238587 0.65758835 0.04245465
[6,] 5.500583 6.119701 0.61911779 0.04336084
[7,] 5.394195 6.016255 0.62205988 0.04243363
[8,] 5.320052 5.921086 0.60103416 0.04233645
我想要获取聚类数量,但与pamk函数不同的是,我无法找到使用clusGap函数获取此数字的方法。
然后我尝试使用maxSE函数,但我不知道参数f和SE.f代表什么,也不知道如何从数据矩阵中获取它们。
是否有简单的方法来获取最佳聚类数?