我是R语言的新手,我从数据框(allTog)中创建了一个图形对象,如下所示:
library(igraph)
df.g <- graph.data.frame(d = allTog, directed = TRUE)
plot(df.g, vertex.label = V(df.g)$name)
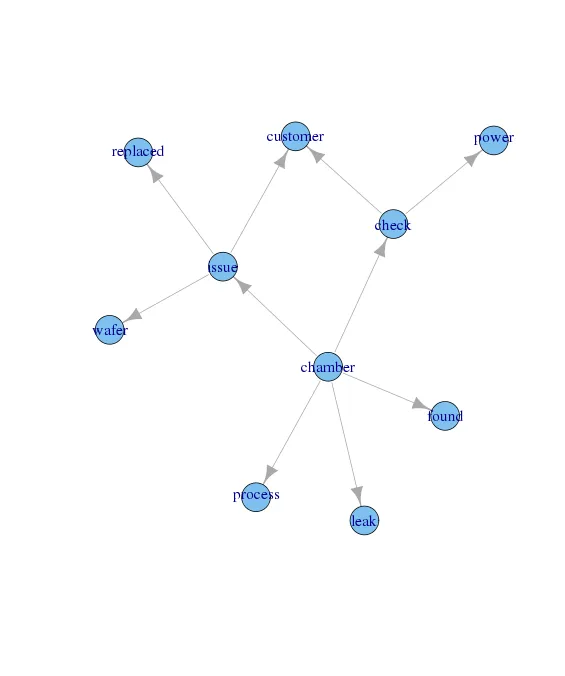
allTog数据框的定义如下:
allTog <- data.frame(
source = c("chamber", "chamber", "chamber", "chamber", "chamber",
"check", "check", "issue", "issue", "issue"),
target = c("check", "issue", "leak", "process", "found", "power",
"customer", "customer", "wafer", "replaced")
)
这里不关心“row.names”和“values”列。
我该如何从每个根节点(在此示例中为“chamber”)遍历到每个叶节点并获取路径,即所有节点(顶点)名称?我正在寻找一种通用解决方案,因为我的根节点可能会随着代码的每次运行而改变。例如,在下一次运行中,根节点可以是“issue”。
我想要的输出是:
chamber->check->power
chamber->issue->replaced
chamber->process
chamber->issue->customer
So and so forth....