我正在尝试使用nlme和lsoda来拟合一阶常微分方程模型。基本思路是:首先定义函数以生成微分方程的解决方案:
library(deSolve)
ODE1 <- function(time, x, parms) {with(as.list(c(parms, x)), {
import <- excfunc(time)
dS <- import*k/tau - (S-yo)/tau
res <- c(dS)
list(res)})}
solution_ODE1 = function(tau1,k1,yo1,excitation,time){
excfunc <- approxfun(time, excitation, rule = 2)
parms <- c(tau = tau1, k = k1, yo = yo1, excfunc = excfunc)
xstart = c(S = yo1)
out <- lsoda(xstart, time, ODE1, parms)
return(out[,2])
}
我会生成两个ID的数据,遵循以下公式:
time <- 0:49
excitation <- c(rep(0,10),rep(1,10),rep(0,10),rep(1,10),rep(0,10))
simu_data <- data.frame(signal = c(solution_ODE1(3,2,0.1,excitation,time)+rnorm(length(time),0,0.1),
solution_ODE1(3.2,1.5,0.3,excitation,time)+rnorm(length(time),0,0.1)),
time = rep(time,2),
excitation = rep(excitation,2),
ID = rep(c("A","B"),each = length(time)))
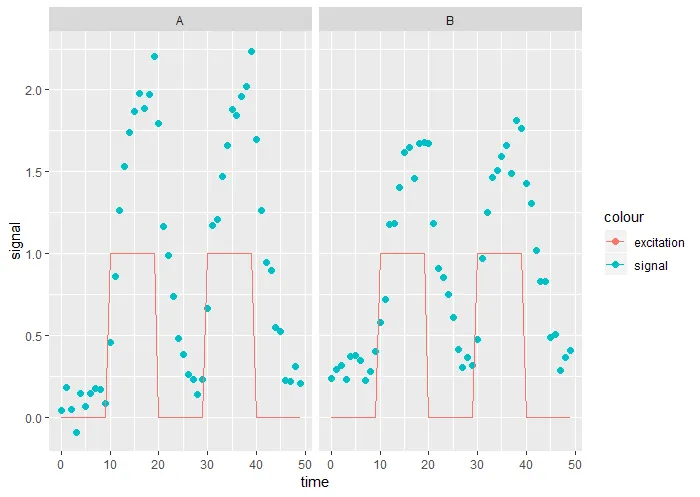
这是它的外观:
library(ggplot2)
ggplot(simu_data)+
geom_point(aes(time,signal,color = "signal"),size = 2)+
geom_line(aes(time,excitation,color = "excitation"))+
facet_wrap(~ID)
我正在尝试使用nlme进行拟合:
fit1 <- nlme(signal ~ solution_ODE1(damping,gain,eq,excitation,time),
data = simu_data,
fixed = damping + gain + eq ~1,
random = damping ~ 1 ,
groups = ~ ID,
start = c(damping = 5, gain = 1,eq = 0))
我得到了这个错误,而我没有得到:
在评估(substitute(expr),data,enclos = parent.frame())时出错:对象'k'未找到
traceback显示该错误来自ODE1模型,在生成值时有效。
16. eval(substitute(expr), data, enclos = parent.frame())
15. eval(substitute(expr), data, enclos = parent.frame())
14. with.default(as.list(c(parms, x)), {
import <- excfunc(time)
dS <- import * k/tau - (S - yo)/tau
res <- c(dS) ...
13. with(as.list(c(parms, x)), {
import <- excfunc(time)
dS <- import * k/tau - (S - yo)/tau
res <- c(dS) ...
12. func(time, state, parms, ...)
11. Func2(times[1], y)
10. eval(Func2(times[1], y), rho)
9. checkFunc(Func2, times, y, rho)
8. lsoda(xstart, time, ODE1, parms)
7. solution_ODE1(damping, gain, eq, excitation, time)
6. eval(model, data.frame(data, pars))
5. eval(model, data.frame(data, pars))
4. eval(modelExpression[[2]], envir = nlEnv)
3. eval(modelExpression[[2]], envir = nlEnv)
2. nlme.formula(signal ~ solution_ODE1(damping, gain, eq, excitation,
time), data = simu_data, fixed = damping + gain + eq ~ 1,
random = damping ~ 1, groups = ~ID, start = c(damping = 5,
gain = 1, eq = 0))
1. nlme(signal ~ solution_ODE1(damping, gain, eq, excitation, time),
data = simu_data, fixed = damping + gain + eq ~ 1, random = damping ~
1, groups = ~ID, start = c(damping = 5, gain = 1, eq = 0))
有没有人有想法,我应该如何继续?
编辑
我尝试根据mikeck的建议进行修改:
ODE1 <- function(time, x, parms) {
import <- parms$excfunc(time)
dS <- import*parms$k/parms$tau - (x["S"]-parms$yo)/parms$tau
res <- c(dS)
list(res)}
生成数据没有问题。但使用
nlme 现在出现以下错误:
并伴随着以下回溯信息:Error in checkFunc(Func2, times, y, rho) : The number of derivatives returned by func() (0) must equal the length of the initial conditions vector (100)
> traceback()
10: stop(paste("The number of derivatives returned by func() (",
length(tmp[[1]]), ") must equal the length of the initial conditions vector (",
length(y), ")", sep = ""))
9: checkFunc(Func2, times, y, rho)
8: lsoda(xstart, time, ODE1, parms) at #5
7: solution_ODE1(damping, gain, eq, excitation, time)
6: eval(model, data.frame(data, pars))
5: eval(model, data.frame(data, pars))
4: eval(modelExpression[[2]], envir = nlEnv)
3: eval(modelExpression[[2]], envir = nlEnv)
2: nlme.formula(signal ~ solution_ODE1(damping, gain, eq, excitation,
time), data = simu_data, fixed = damping + gain + eq ~ 1,
random = damping ~ 1, groups = ~ID, start = c(damping = 5,
gain = 1, eq = 0))
1: nlme(signal ~ solution_ODE1(damping, gain, eq, excitation, time),
data = simu_data, fixed = damping + gain + eq ~ 1, random = damping ~
1, groups = ~ID, start = c(damping = 5, gain = 1, eq = 0))
nlmeODE包吗? - Ben Bolkerxstart <- yo1(然后在ODE1中直接引用x),但我仍然收到“非法输入”消息... - Ben BolkerODE1(),不使用with(),而是使用parms$k等吗?错误信息看起来可能是某种作用域问题引起的。 - mikecknlme在内部做什么,但它似乎给函数提供了初始条件的向量,从而导致错误。 - denis