我有一个可用的glm模型。由于我想添加(岭)正则化,所以我想切换到glmnet。但出现了某些问题,我不能让glmnet工作。它似乎总是预测第一个类别,而不是第二个类别,这导致了低准确度和kappa=0。
以下是一些重现问题的代码。我做错了什么?
它生成的测试数据如下:
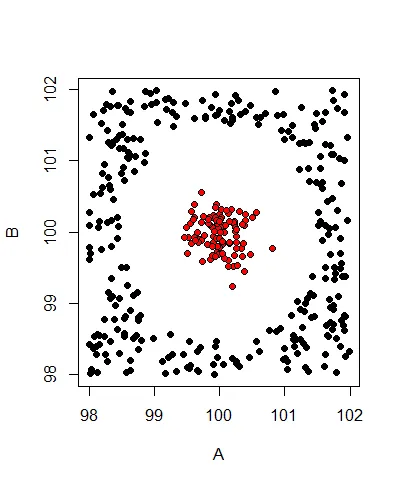
由于数据无法线性分离,因此添加了两个多项式项A^2和B^2.
一个glm模型可以正确预测数据(准确率为1,kappa为1)。下面是它的预测边界:
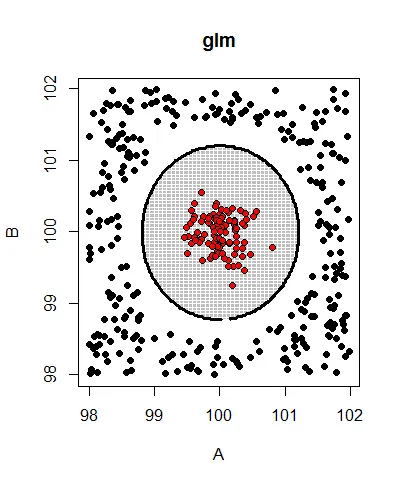
而glmnet模型始终具有kappa = 0,无论尝试什么lambda值:
lambda Accuracy Kappa Accuracy SD Kappa SD
0 0.746 0 0.0295 0
1e-04 0.746 0 0.0295 0
0.01 0.746 0 0.0295 0
0.1 0.746 0 0.0295 0
1 0.746 0 0.0295 0
10 0.746 0 0.0295 0
重现问题的代码:
library(caret)
# generate test data
set.seed(42)
n <- 500; m <- 100
data <- data.frame(A=runif(n, 98, 102), B=runif(n, 98, 102), Type="foo")
data <- subset(data, sqrt((A-100)^2 + (B-100)^2) > 1.5)
data <- rbind(data, data.frame(A=rnorm(m, 100, 0.25), B=rnorm(m, 100, 0.25), Type="bar"))
# add a few polynomial features to match ellipses
polymap <- function(data) cbind(data, A2=data$A^2, B2=data$B^2)
data <- polymap(data)
plot(x=data$A, y=data$B, pch=21, bg=data$Type, xlab="A", ylab="B")
# train a binomial glm model
model.glm <- train(Type ~ ., data=data, method="glm", family="binomial",
preProcess=c("center", "scale"))
# train a binomial glmnet model with ridge regularization (alpha = 0)
model.glmnet <- train(Type ~ ., data=data, method="glmnet", family="binomial",
preProcess=c("center", "scale"),
tuneGrid=expand.grid(alpha=0, lambda=c(0, 0.0001, 0.01, 0.1, 1, 10)))
print(model.glm) # <- Accuracy = 1, Kappa = 1 - good!
print(model.glmnet) # <- Accuracy = low, Kappa = 0 - bad!
直接调用glmnet(不使用caret)会导致相同的问题:
x <- as.matrix(subset(data, select=-c(Type)))
y <- data$Type
model.glmnet2 <- cv.glmnet(x=x, y=y, family="binomial", type.measure="class")
preds <- predict(model.glmnet2, x, type="class", s="lambda.min")
# all predictions are class 1...
编辑:按比例缩放的数据绘制的图形和glm找到的决策边界:
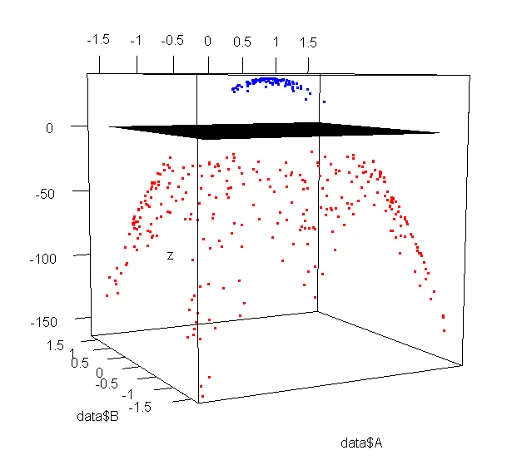
模型:-37 + 6317*A + 6059*B - 6316*A2 - 6059*B2