我正在进行聚类任务,有一个距离矩阵。我希望将这个距离矩阵可视化为2D图形。请告诉我是否有在线或在R或Python等编程语言中完成此操作的方法。
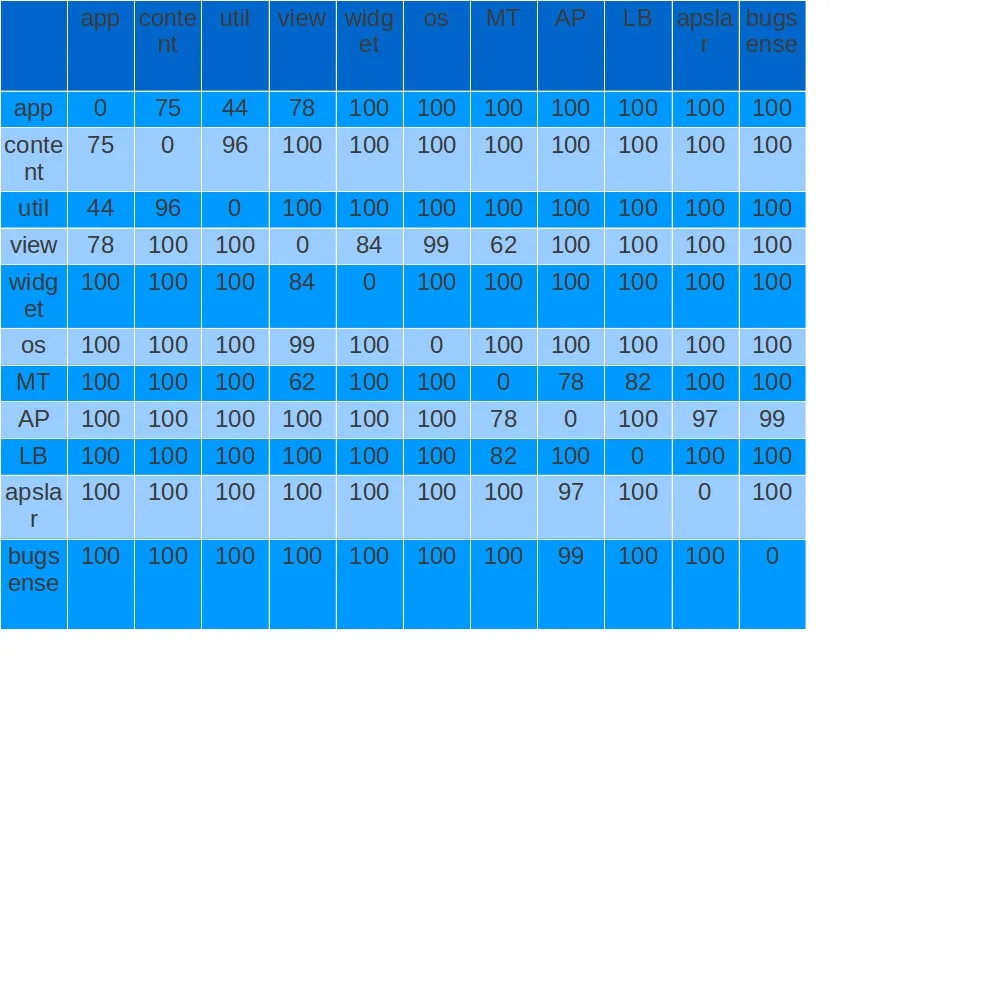
我的距离矩阵如下所示:
 我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图:
我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图: 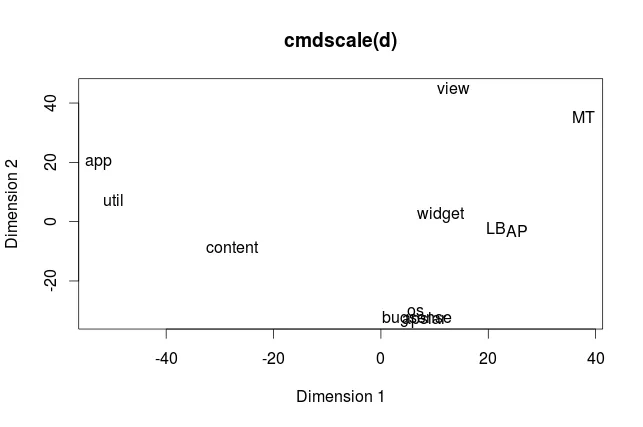 但是我想要的是一个具有节点和加权边的图形。
但是我想要的是一个具有节点和加权边的图形。
 我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图:
我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图:  但是我想要的是一个具有节点和加权边的图形。
但是我想要的是一个具有节点和加权边的图形。 我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图:
我使用了经典多维缩放功能(在R中),并获得了一个看起来像这样的2D图:  但是我想要的是一个具有节点和加权边的图形。
但是我想要的是一个具有节点和加权边的图形。我假定您想要一个二维图,其中节点位置之间的距离与您的表所提供的距离相同。
在Python中,您可以使用networkx进行此类应用程序。一般来说,有许多方法可以实现这一点,记住,它们都只是近似值(因为通常不可能根据它们的成对距离创建点的二维表示),它们是某种应力最小化(或能量最小化)的近似值,试图找到与所提供的“相似”距离具有“合理”的表示。
例如,您可以考虑一个四个点的示例(使用正确的离散度量):
p1 p2 p3 p4
---------------
p1 0 1 1 1
p2 1 0 1 1
p3 1 1 0 1
p4 1 1 1 0
通常情况下,绘制实际的“图形”是多余的,因为你已经完全连接了它(每一对节点都是相连的),所以只绘制点应该就足够了。
import networkx as nx
import numpy as np
import string
dt = [('len', float)]
A = np.array([(0, 0.3, 0.4, 0.7),
(0.3, 0, 0.9, 0.2),
(0.4, 0.9, 0, 0.1),
(0.7, 0.2, 0.1, 0)
])*10
A = A.view(dt)
G = nx.from_numpy_matrix(A)
G = nx.relabel_nodes(G, dict(zip(range(len(G.nodes())),string.ascii_uppercase)))
G = nx.to_agraph(G)
G.node_attr.update(color="red", style="filled")
G.edge_attr.update(color="blue", width="2.0")
G.draw('distances.png', format='png', prog='neato')
在R中,你可以尝试多维缩放
# Classical MDS
# N rows (objects) x p columns (variables)
# each row identified by a unique row name
d <- dist(mydata) # euclidean distances between the rows
fit <- cmdscale(d,eig=TRUE, k=2) # k is the number of dim
fit # view results
# plot solution
x <- fit$points[,1]
y <- fit$points[,2]
plot(x, y, xlab="Coordinate 1", ylab="Coordinate 2",
main="Metric MDS", type="n")
text(x, y, labels = row.names(mydata), cex=.7)
你只是想绘制带有标签的边缘图
同样,networkx 可以提供帮助:
import networkx as nx
# Create a graph
G = nx.Graph()
# distances
D = [ [0, 1], [1, 0] ]
labels = {}
for n in range(len(D)):
for m in range(len(D)-(n+1)):
G.add_edge(n,n+m+1)
labels[ (n,n+m+1) ] = str(D[n][n+m+1])
pos=nx.spring_layout(G)
nx.draw(G, pos)
nx.draw_networkx_edge_labels(G,pos,edge_labels=labels,font_size=30)
import pylab as plt
plt.show()
http://vida.io/documents/SyT7DREdQmGSpsBkK
另一种可视化的方法是使用节点之间相同的距离,但不同的线条粗细。在这种情况下,您需要根据数值计算描边宽度:.style("stroke-width", function(d) { return Math.sqrt(d.value / 50); });