我使用了'raster'包来制作具有二项响应变量概率的地图。问题是,当我绘制结果时,我的预测栅格值范围从-15到5,而不是0-1。我使用的代码与Hijmans、Elith. 2016年的《R中的物种分布建模》(第34页)相同。他们得到的概率范围是从0到1,而我却一直得到奇怪的值。我做错了什么?以下是我的前50行数据的可重现示例。
这是我的理解:
install.packages("lme4")
install.packages("raster")
install.packages("rgdal")
library("lme4")
library("raster")
library("rgdal")
# my data
data = structure(list(colorSymbol = c(1L, 1L, 1L, 0L, 0L, 1L, 0L, 0L,
1L, 1L, 0L, 0L, 1L, 1L, 1L, 1L, 0L, 0L, 1L, 1L, 0L, 0L, 0L, 0L,
0L, 0L, 0L, 1L, 0L, 1L, 0L, 1L, 0L, 1L, 1L, 0L, 0L, 0L, 0L, 0L,
0L, 1L, 1L, 0L, 0L, 0L, 0L, 0L, 0L, 1L), bio_2 = c(75L, 168L,
57L, 127L, 120L, 100L, 97L, 97L, 97L, 97L, 94L, 102L, 102L, 89L,
89L, 102L, 96L, 97L, 92L, 100L, 97L, 97L, 97L, 96L, 97L, 95L,
97L, 105L, 96L, 92L, 96L, 97L, 97L, 88L, 95L, 95L, 95L, 99L,
96L, 97L, 97L, 100L, 97L, 96L, 94L, 94L, 94L, 94L, 98L, 94L),
bio_3 = c(24L, 36L, 32L, 57L, 31L, 31L, 32L, 32L, 32L, 32L,
31L, 33L, 31L, 32L, 32L, 33L, 32L, 31L, 32L, 33L, 32L, 32L,
32L, 32L, 32L, 32L, 32L, 34L, 31L, 32L, 32L, 32L, 32L, 32L,
32L, 32L, 32L, 31L, 32L, 32L, 32L, 34L, 31L, 32L, 31L, 31L,
31L, 31L, 32L, 32L)), .Names = c("colorSymbol", "bio_2",
"bio_3"), row.names = c(NA, 50L), class = "data.frame")
# model
bio2 = data$bio_2
bio3 = data$bio_3
colorSymbol = data$colorSymbol
model = glm(colorSymbol ~ bio2 + bio3, family = binomial)
# predictors
w = getData('worldclim', var='bio', res=10)
rasstack <- stack(w$bio2, w$bio3)
p <- raster::predict(rasstack, model)
plot(p)
这是我的理解:
我得到的结果如下: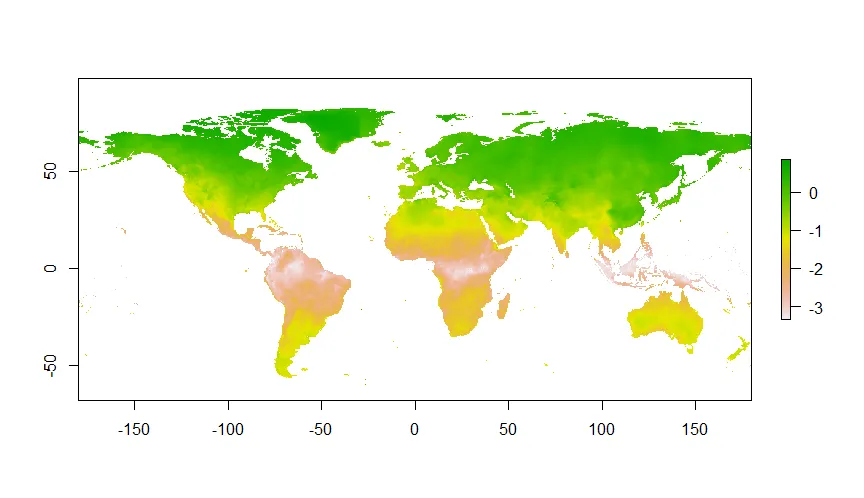
我进行了彻底的在线搜索,但无法确定问题出在哪里。