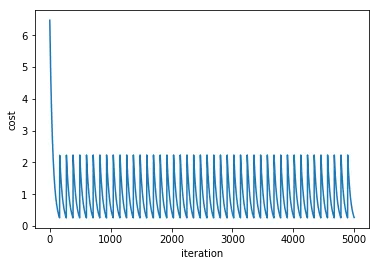
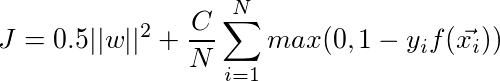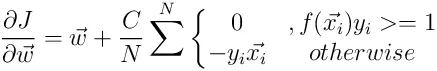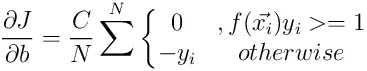我正在学习支持向量机,并尝试编写一个简单的Python实现(我知道sklearn包,只是为了更好地理解概念),用于进行简单的线性分类。这是我参考的主要材料。
我正在尝试从原始问题开始解决SVM,通过最小化以下内容:
以下是我的代码:
所以这只是一个玩具示例,只有4个线性可分的训练样本,我删除了偏置项b,期望结果w为[0.5,0.5](skimage结果),而我的实现将倾向于给出大于0.5的值(例如[1.4650,1.4650]),无论是使用梯度下降还是scipy.optimize。只有当C>1时,才会发生这种情况,当C==1时,它会给我[0.5,0.5]。此外,当C>1时,scipy.optimize会失败(我尝试了几种不同的方法,例如Newton-CG、BFGS),尽管最终结果接近于梯度下降结果。
我有点困惑为什么w向量停止缩小。我认为当所有数据都被正确分类时,松弛惩罚将停止对总成本函数的贡献,因此它只会通过减少w的大小来优化J。我算错了吗?
我知道这可能是一个新手问题,我在粘贴一些脏代码,这已经困扰我几天了,我周围没有人可以提供帮助,所以任何支持都将不胜感激!
更新:
感谢所有的帮助。我正在更新代码以处理稍微复杂一些的样本。这次我包括了偏置项,并使用以下内容进行更新:
我正在尝试从原始问题开始解决SVM,通过最小化以下内容:
J对w的导数是(根据上面的参考文献):
所以这里使用了“铰链”损失函数,C是惩罚参数。如果我理解正确,设置较大的C将会强制SVM具有更严格的间隔。以下是我的代码:
import numpy
from scipy import optimize
class SVM2C(object):
def __init__(self,xdata,ydata,c=200.,learning_rate=0.01,
n_iter=5000,method='GD'):
self.values=numpy.unique(ydata)
self.xdata=xdata
self.ydata=numpy.where(ydata==self.values[-1],1,-1)
self.c=c
self.lr=learning_rate
self.n_iter=n_iter
self.method=method
self.m=len(xdata)
self.theta=numpy.random.random(xdata.shape[1])-0.5
def costFunc(self,theta,x,y):
zs=numpy.dot(x,theta)
j=numpy.maximum(0.,1.-y*zs).mean()*self.c+0.5*numpy.sum(theta**2)
return j
def jac(self,theta,x,y):
'''Derivative of cost function'''
zs=numpy.dot(x,theta)
ee=numpy.where(y*zs>=1.,0.,-y)[:,None]
# multiply rows by ee
dj=(ee*x).mean(axis=0)*self.c+theta
return dj
def train(self):
#----------Optimize using scipy.optimize----------
if self.method=='optimize':
opt=optimize.minimize(self.costFunc,self.theta,args=(self.xdata,self.ydata),\
jac=self.jac,method='BFGS')
self.theta=opt.x
#---------Optimize using Gradient descent---------
elif self.method=='GD':
costs=[]
lr=self.lr
for ii in range(self.n_iter):
dj=self.jac(self.theta,self.xdata,self.ydata)
self.theta=self.theta-lr*dj
cii=self.costFunc(self.theta,self.xdata,self.ydata)
costs.append(cii)
self.costs=numpy.array(costs)
return self
def predict(self,xdata):
yhats=[]
for ii in range(len(xdata)):
xii=xdata[ii]
yhatii=xii.dot(self.theta)
yhatii=1 if yhatii>=0 else 0
yhats.append(yhatii)
yhats=numpy.array(yhats)
return yhats
#-------------Main---------------------------------
if __name__=='__main__':
xdata = numpy.array([[-1, -1], [-2, -1], [1, 1], [2, 1]])
ydata = numpy.array([1, 1, 2, 2])
mysvm=SVM2C(xdata,ydata,method='GD')
mysvm.train()
from sklearn import svm
clf=svm.SVC(C=50,kernel='linear')
clf.fit(xdata,ydata)
print mysvm.theta
print clf.coef_
#-------------------Plot------------------------
import matplotlib.pyplot as plt
figure=plt.figure(figsize=(12,10),dpi=100)
ax=figure.add_subplot(111)
cmap=plt.cm.jet
nclasses=numpy.unique(ydata).tolist()
colors=[cmap(float(ii)/len(nclasses)) for ii in nclasses]
#----------------Plot training data----------------
for ii in range(len(ydata)):
xii=xdata[ii][0]
yii=xdata[ii][1]
colorii=colors[nclasses.index(ydata[ii])]
ax.plot(xii,yii,color=colorii,marker='o')
plt.show(block=False)
所以这只是一个玩具示例,只有4个线性可分的训练样本,我删除了偏置项b,期望结果w为[0.5,0.5](skimage结果),而我的实现将倾向于给出大于0.5的值(例如[1.4650,1.4650]),无论是使用梯度下降还是scipy.optimize。只有当C>1时,才会发生这种情况,当C==1时,它会给我[0.5,0.5]。此外,当C>1时,scipy.optimize会失败(我尝试了几种不同的方法,例如Newton-CG、BFGS),尽管最终结果接近于梯度下降结果。
我有点困惑为什么w向量停止缩小。我认为当所有数据都被正确分类时,松弛惩罚将停止对总成本函数的贡献,因此它只会通过减少w的大小来优化J。我算错了吗?
我知道这可能是一个新手问题,我在粘贴一些脏代码,这已经困扰我几天了,我周围没有人可以提供帮助,所以任何支持都将不胜感激!
更新:
感谢所有的帮助。我正在更新代码以处理稍微复杂一些的样本。这次我包括了偏置项,并使用以下内容进行更新:
根据我收到的反馈,我尝试了scipy.optimize的Nelder-Mead方法,并尝试了两种自适应梯度下降方法。以下是代码:
import numpy
from scipy import optimize
class SVM2C(object):
def __init__(self,xdata,ydata,c=9000,learning_rate=0.001,
n_iter=600,method='GD'):
self.values=numpy.unique(ydata)
# Add 1 dimension for bias
self.xdata=numpy.hstack([xdata,numpy.ones([xdata.shape[0],1])])
self.ydata=numpy.where(ydata==self.values[-1],1,-1)
self.c=c
self.lr=learning_rate
self.n_iter=n_iter
self.method=method
self.m=len(xdata)
self.theta=numpy.random.random(self.xdata.shape[1])-0.5
def costFunc(self,theta,x,y):
zs=numpy.dot(x,theta)
j=numpy.maximum(0.,1.-y*zs).mean()*self.c+0.5*numpy.sum(theta[:-1]**2)
return j
def jac(self,theta,x,y):
'''Derivative of cost function'''
zs=numpy.dot(x,theta)
ee=numpy.where(y*zs>=1.,0.,-y)[:,None]
dj=numpy.zeros(self.theta.shape)
dj[:-1]=(ee*x[:,:-1]).mean(axis=0)*self.c+theta[:-1] # weights
dj[-1]=(ee*self.c).mean(axis=0) # bias
return dj
def train(self):
#----------Optimize using scipy.optimize----------
if self.method=='optimize':
opt=optimize.minimize(self.costFunc,self.theta,args=(self.xdata,self.ydata),\
jac=self.jac,method='Nelder-Mead')
self.theta=opt.x
#---------Optimize using Gradient descent---------
elif self.method=='GD':
costs=[]
lr=self.lr
# ADAM parameteres
beta1=0.9
beta2=0.999
epsilon=1e-8
mt_1=0
vt_1=0
for ii in range(self.n_iter):
t=ii+1
dj=self.jac(self.theta,self.xdata,self.ydata)
'''
mt=beta1*mt_1+(1-beta1)*dj
vt=beta2*vt_1+(1-beta2)*dj**2
mt=mt/(1-beta1**t)
vt=vt/(1-beta2**t)
self.theta=self.theta-lr*mt/(numpy.sqrt(vt)+epsilon)
mt_1=mt
vt_1=vt
cii=self.costFunc(self.theta,self.xdata,self.ydata)
'''
old_theta=self.theta
self.theta=self.theta-lr*dj
if ii>0 and cii>costs[-1]:
lr=lr*0.9
self.theta=old_theta
costs.append(cii)
self.costs=numpy.array(costs)
self.b=self.theta[-1]
self.theta=self.theta[:-1]
return self
def predict(self,xdata):
yhats=[]
for ii in range(len(xdata)):
xii=xdata[ii]
yhatii=numpy.sign(xii.dot(self.theta)+self.b)
yhatii=xii.dot(self.theta)+self.b
yhatii=self.values[-1] if yhatii>=0 else self.values[0]
yhats.append(yhatii)
yhats=numpy.array(yhats)
return yhats
#-------------Main---------------------------------
if __name__=='__main__':
#------------------Sample case 1------------------
#xdata = numpy.array([[-1, -1], [-2, -1], [1, 1], [2, 1]])
#ydata = numpy.array([1, 1, 2, 2])
#------------------Sample case 2------------------
from sklearn import datasets
iris=datasets.load_iris()
xdata=iris.data[20:,:2]
ydata=numpy.where(iris.target[20:]>0,1,0)
#----------------------Train----------------------
mysvm=SVM2C(xdata,ydata,method='GD')
mysvm.train()
ntest=20
xtest=2*(numpy.random.random([ntest,2])-0.5)+xdata.mean(axis=0)
from sklearn import svm
clf=svm.SVC(C=50,kernel='linear')
clf.fit(xdata,ydata)
yhats=mysvm.predict(xtest)
yhats2=clf.predict(xtest)
print 'mysvm weights:', mysvm.theta, 'intercept:', mysvm.b
print 'sklearn weights:', clf.coef_, 'intercept:', clf.intercept_
print 'mysvm predict:',yhats
print 'sklearn predict:',yhats2
#-------------------Plot------------------------
import matplotlib.pyplot as plt
figure=plt.figure(figsize=(12,10),dpi=100)
ax=figure.add_subplot(111)
cmap=plt.cm.jet
nclasses=numpy.unique(ydata).tolist()
colors=[cmap(float(ii)/len(nclasses)) for ii in nclasses]
#----------------Plot training data----------------
for ii in range(len(ydata)):
xii=xdata[ii][0]
yii=xdata[ii][1]
colorii=colors[nclasses.index(ydata[ii])]
ax.plot(xii,yii,color=colorii,marker='o',markersize=15)
#------------------Plot test data------------------
for ii in range(ntest):
colorii=colors[nclasses.index(yhats2[ii])]
ax.plot(xtest[ii][0],xtest[ii][1],color=colorii,marker='^',markersize=5)
#--------------------Plot line--------------------
x1=xdata[:,0].min()
x2=xdata[:,0].max()
y1=(-clf.intercept_-clf.coef_[0][0]*x1)/clf.coef_[0][1]
y2=(-clf.intercept_-clf.coef_[0][0]*x2)/clf.coef_[0][1]
y3=(-mysvm.b-mysvm.theta[0]*x1)/mysvm.theta[1]
y4=(-mysvm.b-mysvm.theta[0]*x2)/mysvm.theta[1]
ax.plot([x1,x2],[y1,y2],'-k',label='sklearn line')
ax.plot([x1,x2],[y3,y4],':k',label='mysvm line')
ax.legend(loc=0)
plt.show(block=False)
我遇到的新问题:
- 它不稳定,取决于初始随机参数的设置,结果可能会相差很大。即使我已经将
C设置得非常大,它仍然有约一半的时间会在训练集中误分类1个样本。这在scipy.optimize和GD中都会发生。 - ADAM方法倾向于给出
vt的inf值,因为对于较大的C,vt增长得非常快。我是不是在计算梯度时出了问题?
提前无限感谢!