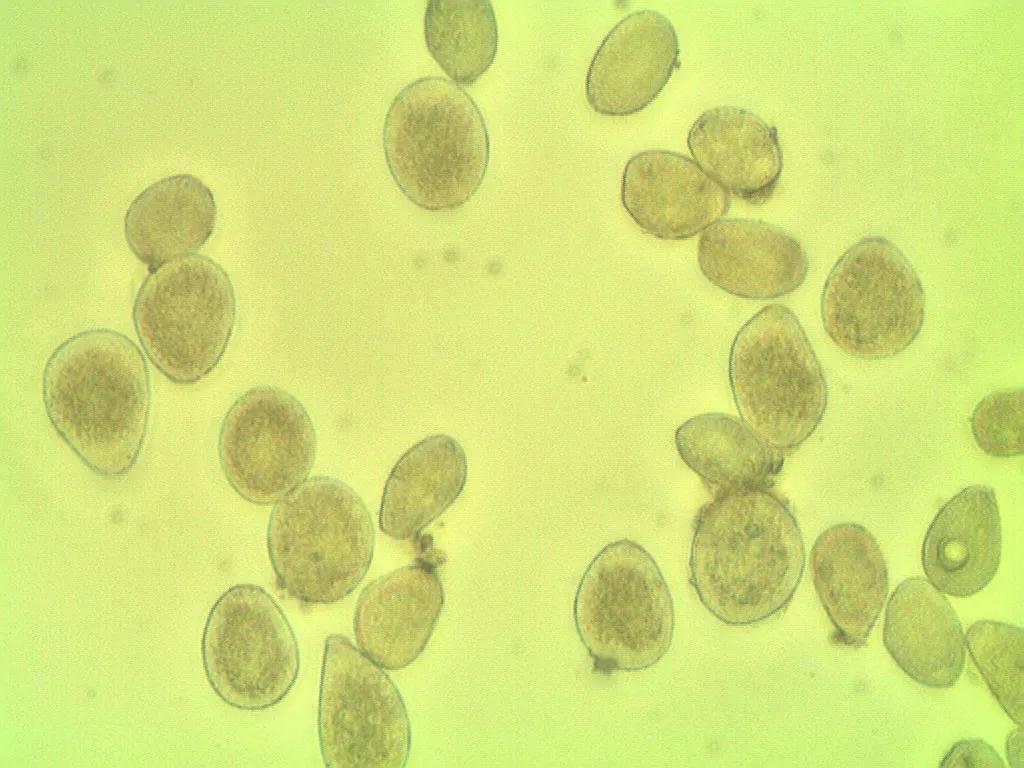
首先,我们需要一些预备代码,下面将会用到它:
import numpy as np
import cv2
from matplotlib import pyplot as plt
from skimage.morphology import extrema
from skimage.morphology import watershed as skwater
def ShowImage(title,img,ctype):
if ctype=='bgr':
b,g,r = cv2.split(img)
rgb_img = cv2.merge([r,g,b])
plt.imshow(rgb_img)
elif ctype=='hsv':
rgb = cv2.cvtColor(img,cv2.COLOR_HSV2RGB)
plt.imshow(rgb)
elif ctype=='gray':
plt.imshow(img,cmap='gray')
elif ctype=='rgb':
plt.imshow(img)
else:
raise Exception("Unknown colour type")
plt.title(title)
plt.show()
供参考,这是您的原始图像:
img = cv2.imread('cells.jpg')
ShowImage('Original',img,'bgr')

大津法是一种分割颜色的方法。该方法假设图像像素的强度可以绘制成双峰直方图,并找到该直方图的最佳分隔符。我在下面应用了这种方法。
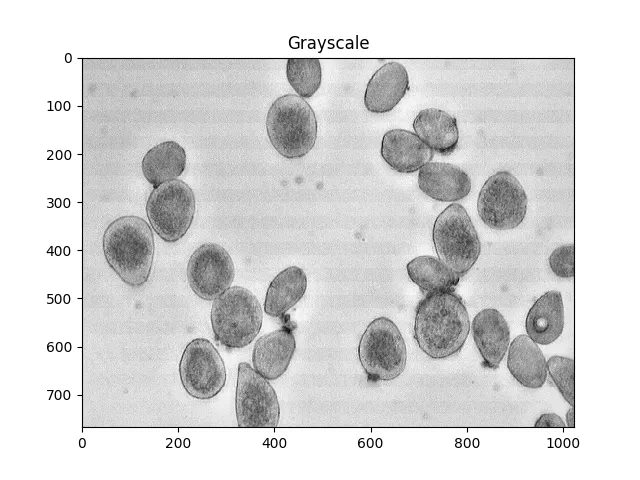
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, thresh = cv2.threshold(gray,0,255,cv2.THRESH_BINARY_INV+cv2.THRESH_OTSU)
ShowImage('Grayscale',gray,'gray')
ShowImage('Applying Otsu',thresh,'gray')

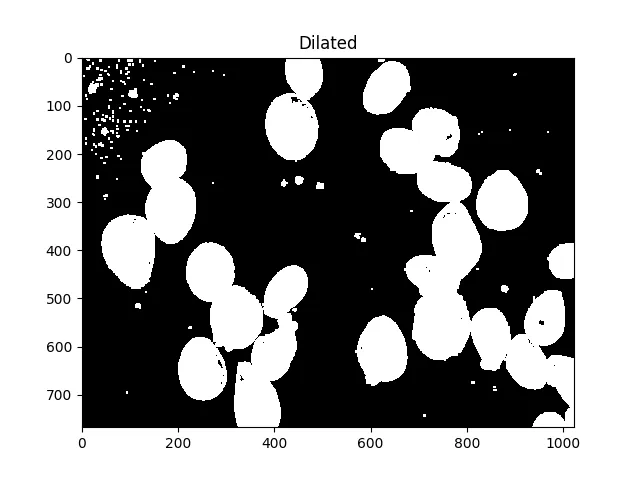
所有这些小斑点都很烦人,我们可以通过膨胀来消除它们:
#Adjust iterations until desired result is achieved
kernel = np.ones((3,3),np.uint8)
dilated = cv2.dilate(thresh, kernel, iterations=5)
ShowImage('Dilated',dilated,'gray')
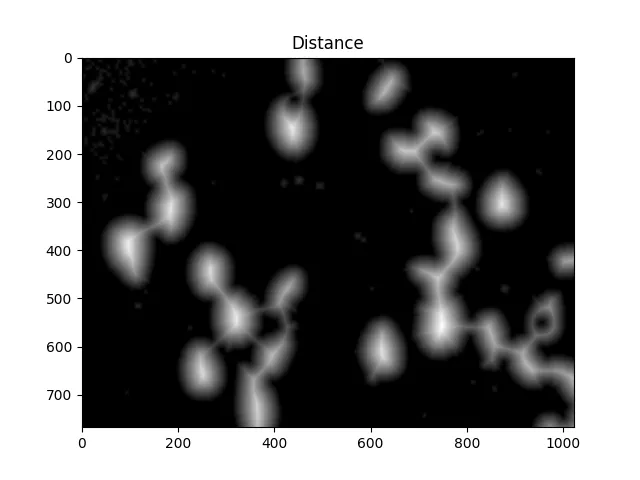
现在我们需要确定分水岭的峰值并给它们单独的标签。这样做的目的是生成一组像素,使每个单元格内都有一个像素,并且没有两个单元格的标识符像素相接触。
为了实现这一点,我们执行距离变换,然后过滤掉距离细胞中心太远的距离。
dist = cv2.distanceTransform(dilated,cv2.DIST_L2,5)
ShowImage('Distance',dist,'gray')
#Adjust this parameter until desired separation occurs
fraction_foreground = 0.6
ret, sure_fg = cv2.threshold(dist,fraction_foreground*dist.max(),255,0)
ShowImage('Surely Foreground',sure_fg,'gray')
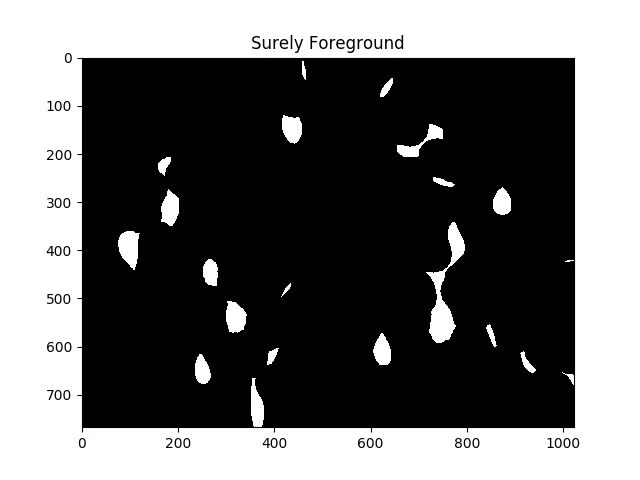
以上图像中的每个白色区域在算法看来都是一个单独的单元。
现在,我们通过减去最大值来识别未知区域,即将由分水岭算法标记的区域:
unknown = cv2.subtract(dilated,sure_fg.astype(np.uint8))
ShowImage('Unknown',unknown,'gray')
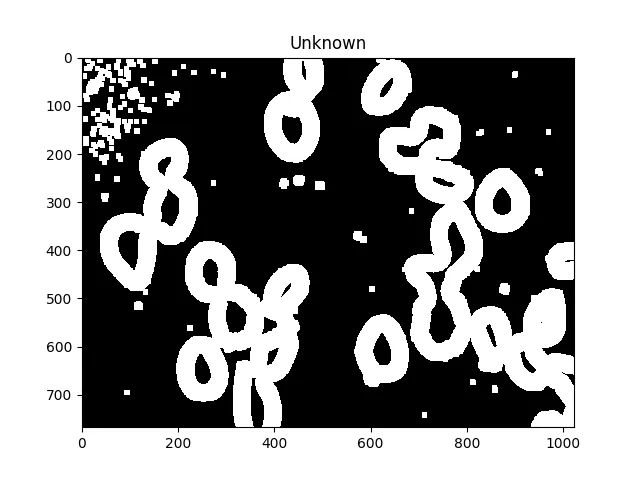
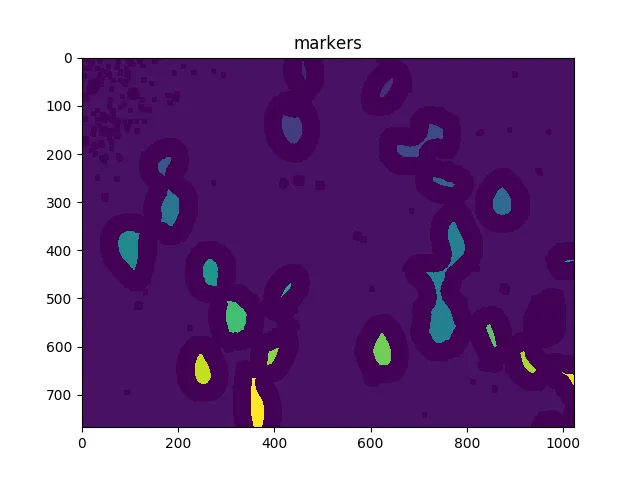
未知区域应该在每个单元格周围形成完整的甜甜圈。
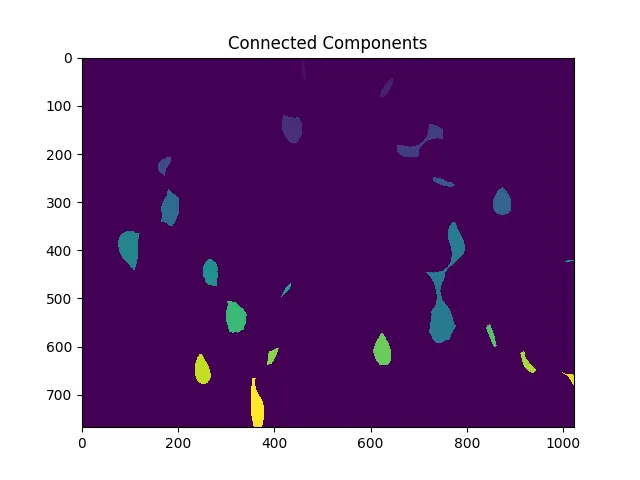
接下来,我们为距离变换产生的每个不同区域分配唯一标签,然后标记未知区域,最后执行分水岭变换:
ret, markers = cv2.connectedComponents(sure_fg.astype(np.uint8))
ShowImage('Connected Components',markers,'rgb')
markers = markers+1
markers[unknown==np.max(unknown)] = 0
ShowImage('markers',markers,'rgb')
dist = cv2.distanceTransform(dilated,cv2.DIST_L2,5)
markers = skwater(-dist,markers,watershed_line=True)
ShowImage('Watershed',markers,'rgb')
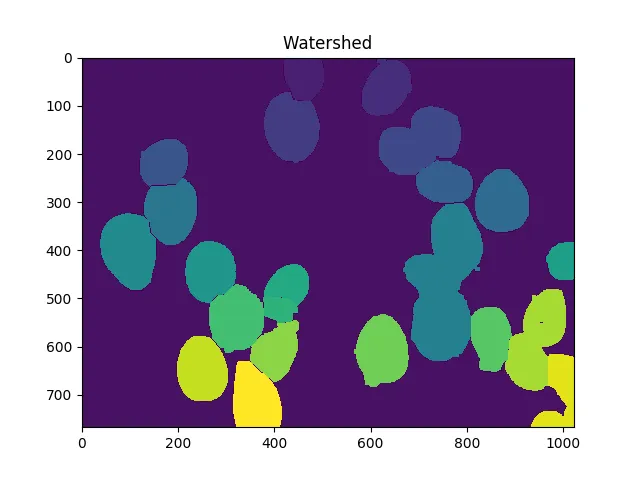
现在单元格的总数是唯一标记数量减1(忽略背景):
len(set(markers.flatten()))-1
在这种情况下,我们得到了23。
通过调整距离阈值、膨胀程度,可能使用h-maxima(局部阈值最大值)等方法,可以使其更加准确或不准确。但要注意过度拟合;也就是说,不要假设为单个图像进行调整将在任何地方都给出最佳结果。
估计不确定性
您还可以通过算法略微改变参数来了解计数中的不确定性。这可能看起来像这样:
import numpy as np
import cv2
import itertools
from matplotlib import pyplot as plt
from skimage.morphology import extrema
from skimage.morphology import watershed as skwater
def CountCells(dilation=5, fg_frac=0.6):
img = cv2.imread('cells.jpg')
gray = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
ret, thresh = cv2.threshold(gray,0,255,cv2.THRESH_BINARY_INV+cv2.THRESH_OTSU)
kernel = np.ones((3,3),np.uint8)
dilated = cv2.dilate(thresh, kernel, iterations=dilation)
dist = cv2.distanceTransform(dilated,cv2.DIST_L2,5)
fraction_foreground = fg_frac
ret, sure_fg = cv2.threshold(dist,fraction_foreground*dist.max(),255,0)
unknown = cv2.subtract(dilated,sure_fg.astype(np.uint8))
ret, markers = cv2.connectedComponents(sure_fg.astype(np.uint8))
markers = markers+1
markers[unknown==np.max(unknown)] = 0
markers = skwater(-dist,markers,watershed_line=True)
return len(set(markers.flatten()))-1
dilations = [4,5,6]
fracs = [0.5, 0.6, 0.7, 0.8]
for params in itertools.product(dilations,fracs):
print("Dilation={0}, FG frac={1}, Count={2}".format(*params,CountCells(*params)))
给出结果:
Dilation=4, FG frac=0.5, Count=22
Dilation=4, FG frac=0.6, Count=23
Dilation=4, FG frac=0.7, Count=17
Dilation=4, FG frac=0.8, Count=12
Dilation=5, FG frac=0.5, Count=21
Dilation=5, FG frac=0.6, Count=23
Dilation=5, FG frac=0.7, Count=20
Dilation=5, FG frac=0.8, Count=13
Dilation=6, FG frac=0.5, Count=20
Dilation=6, FG frac=0.6, Count=23
Dilation=6, FG frac=0.7, Count=24
Dilation=6, FG frac=0.8, Count=14
取计数值的中位数是将不确定性合并为一个数字的一种方法。
请记住,StackOverflow的许可要求您给予适当的 归属。在学术工作中,可以通过引用来完成此操作。