我正在尝试使用Python实现Population Monte Carlo算法,该算法在这篇论文中有描述(参见第78页图3),用于一个只有一个参数的简单模型(请参见函数
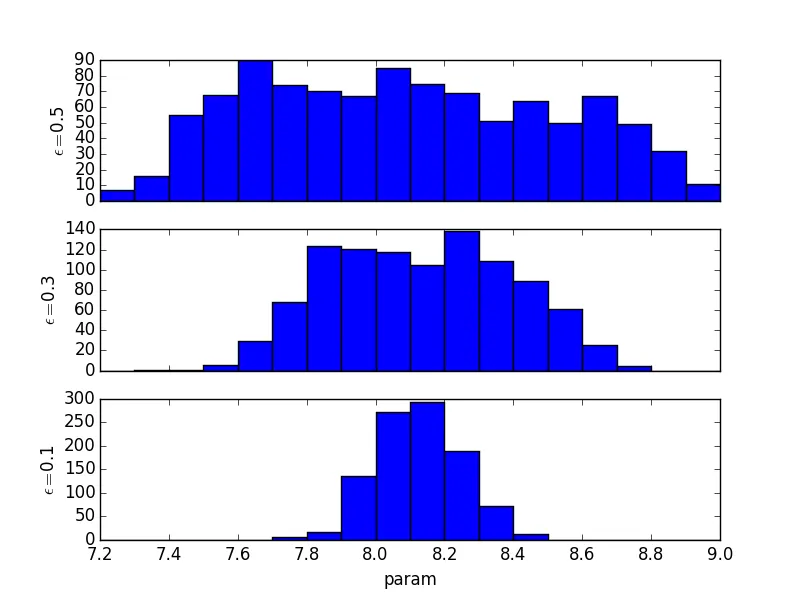
为了检查算法是否有效,我首先使用模型的唯一参数param = 8生成观察数据。因此,由ABC算法产生的后验应该集中在8周围。但事实并非如此,我想知道原因。
我将非常感谢任何帮助或评论。
model())。不幸的是,算法无法正常工作,我无法弄清楚问题所在。请查看下面的代码实现。实际函数名为abc()。所有其他函数都可以视为辅助函数,并且似乎工作正常。为了检查算法是否有效,我首先使用模型的唯一参数param = 8生成观察数据。因此,由ABC算法产生的后验应该集中在8周围。但事实并非如此,我想知道原因。
我将非常感谢任何帮助或评论。
# imports
from math import exp
from math import log
from math import sqrt
import numpy as np
import random
from scipy.stats import norm
# globals
N = 300 # sample size
N_PARTICLE = 300 # number of particles
ITERS = 5 # number of decreasing thresholds
M = 10 # number of words to remember
MEAN = 7 # prior mean of parameter
SD = 2 # prior sd of parameter
def model(param):
recall_prob_all = 1/(1 + np.exp(M - param))
recall_prob_one_item = np.exp(np.log(recall_prob_all) / float(M))
return sum([1 if random.random() < recall_prob_one_item else 0 for item in range(M)])
## example
print "Output of model function: \n" + str(model(10)) + "\n"
# generate data from model
def generate(param):
out = np.empty(N)
for i in range(N):
out[i] = model(param)
return out
## example
print "Output of generate function: \n" + str(generate(10)) + "\n"
# distance function (sum of squared error)
def distance(obsData,simData):
out = 0.0
for i in range(len(obsData)):
out += (obsData[i] - simData[i]) * (obsData[i] - simData[i])
return out
## example
print "Output of distance function: \n" + str(distance([1,2,3],[4,5,6])) + "\n"
# sample new particles based on weights
def sample(particles, weights):
return np.random.choice(particles, 1, p=weights)
## example
print "Output of sample function: \n" + str(sample([1,2,3],[0.1,0.1,0.8])) + "\n"
# perturbance function
def perturb(variance):
return np.random.normal(0,sqrt(variance),1)[0]
## example
print "Output of perturb function: \n" + str(perturb(1)) + "\n"
# compute new weight
def computeWeight(prevWeights,prevParticles,prevVariance,currentParticle):
denom = 0.0
proposal = norm(currentParticle, sqrt(prevVariance))
prior = norm(MEAN,SD)
for i in range(len(prevParticles)):
denom += prevWeights[i] * proposal.pdf(prevParticles[i])
return prior.pdf(currentParticle)/denom
## example
prevWeights = [0.2,0.3,0.5]
prevParticles = [1,2,3]
prevVariance = 1
currentParticle = 2.5
print "Output of computeWeight function: \n" + str(computeWeight(prevWeights,prevParticles,prevVariance,currentParticle)) + "\n"
# normalize weights
def normalize(weights):
return weights/np.sum(weights)
## example
print "Output of normalize function: \n" + str(normalize([3.,5.,9.])) + "\n"
# sampling from prior distribution
def rprior():
return np.random.normal(MEAN,SD,1)[0]
## example
print "Output of rprior function: \n" + str(rprior()) + "\n"
# ABC using Population Monte Carlo sampling
def abc(obsData,eps):
draw = 0
Distance = 1e9
variance = np.empty(ITERS)
simData = np.empty(N)
particles = np.empty([ITERS,N_PARTICLE])
weights = np.empty([ITERS,N_PARTICLE])
for t in range(ITERS):
if t == 0:
for i in range(N_PARTICLE):
while(Distance > eps[t]):
draw = rprior()
simData = generate(draw)
Distance = distance(obsData,simData)
Distance = 1e9
particles[t][i] = draw
weights[t][i] = 1./N_PARTICLE
variance[t] = 2 * np.var(particles[t])
continue
for i in range(N_PARTICLE):
while(Distance > eps[t]):
draw = sample(particles[t-1],weights[t-1])
draw += perturb(variance[t-1])
simData = generate(draw)
Distance = distance(obsData,simData)
Distance = 1e9
particles[t][i] = draw
weights[t][i] = computeWeight(weights[t-1],particles[t-1],variance[t-1],particles[t][i])
weights[t] = normalize(weights[t])
variance[t] = 2 * np.var(particles[t])
return particles[ITERS-1]
true_param = 9
obsData = generate(true_param)
eps = [15000,10000,8000,6000,3000]
posterior = abc(obsData,eps)
#print posterior