我正在尝试使用e1071的svm绘制2维超平面(直线),以分离一个3类问题。我使用了默认方法(因此没有涉及公式),如下所示:
library('e1071')
## S3 method for class 'default':
machine <- svm(x, y, kernel="linear")
我无法使用
plot.svm方法绘制它:plot(machine, x)
Error in plot.svm(machine, x) : missing formula.
但我没有使用formula方法,而是使用了default方法。如果我将'~'或'~.'作为公式参数传递,它会抱怨矩阵x不是data.frame。
- 在使用default方法的同时,有没有一种方法可以绘制2D问题的拟合分隔符?
- 我该如何实现这个目标?
提前致谢。
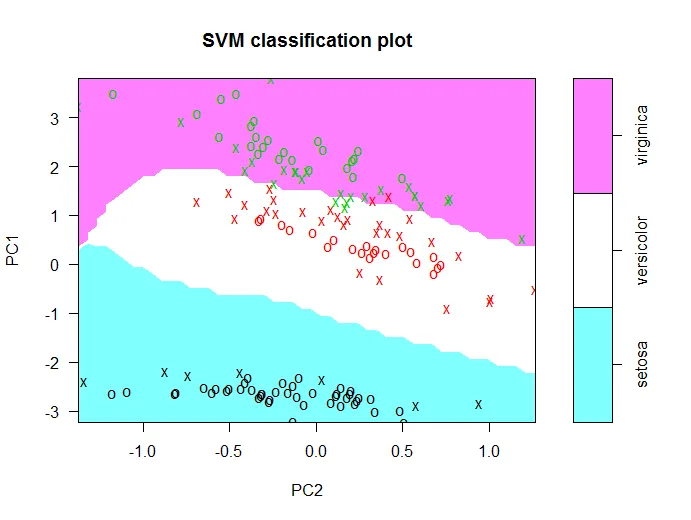
x <- prcomp(iris[,1:4])$x[,1:2]和y <- iris[,5]- undefined