目标:
可视化特定生物种群在有限时间内的大小。
假设:
- 该生物的寿命为
age_limit天 - 只有年龄为
day_lay_egg天的雌性才能产卵,每个雌性最多可以产下max_lay_egg次。每个繁殖期最多只能产下egg_no个蛋,并有50%的概率产生公共后代。 - 初始种群由2个雌性和1个雄性组成,共3个生物。
代码片段:
当前,以下代码应该产生预期输出。
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
def get_breeding(d,**kwargs):
if d['lay_egg'] <= kwargs['max_lay_egg'] and d['dborn'] > kwargs['day_lay_egg'] and d['s'] == 1:
nums = np.random.choice([0, 1], size=kwargs['egg_no'], p=[.5, .5]).tolist()
npol=[dict(s=x,d=d['d'], lay_egg=0, dborn=0) for x in nums]
d['lay_egg'] = d['lay_egg'] + 1
return d,npol
return d,None
def to_loop_initial_population(**kwargs):
npol=kwargs['ipol']
nday = 0
total_population_per_day = []
while nday < kwargs['nday_limit']:
# print(f'Executing day {nday}')
k = []
for dpol in npol:
dpol['d'] += 1
dpol['dborn'] += 1
dpol,h = get_breeding(dpol,**kwargs)
if h is None and dpol['dborn'] <= kwargs['age_limit']:
# If beyond the age limit, ignore the parent and update only the decedent
k.append(dpol)
elif isinstance(h, list) and dpol['dborn'] <= kwargs['age_limit']:
# If below age limit, append the parent and its offspring
h.extend([dpol])
k.extend(h)
total_population_per_day.append(dict(nsize=len(k), day=nday))
nday += 1
npol = k
return total_population_per_day
## Some spec and store all setting in a dict
numsex=[1,1,0] # 0: Male, 1: Female
# s: sex, d: day, lay_egg: Number of time the female lay an egg, dborn: The organism age
ipol=[dict(s=x,d=0, lay_egg=0, dborn=0) for x in numsex] # The initial population
age_limit = 45 # Age limit for the species
egg_no=3 # Number of eggs
day_lay_egg = 30 # Matured age for egg laying
nday_limit=360
max_lay_egg=2
para=dict(nday_limit=nday_limit,ipol=ipol,age_limit=age_limit,
egg_no=egg_no,day_lay_egg=day_lay_egg,max_lay_egg=max_lay_egg)
dpopulation = to_loop_initial_population(**para)
### make some plot
df = pd.DataFrame(dpopulation)
sns.lineplot(x="day", y="nsize", data=df)
plt.xticks(rotation=15)
plt.title('Day vs population')
plt.show()
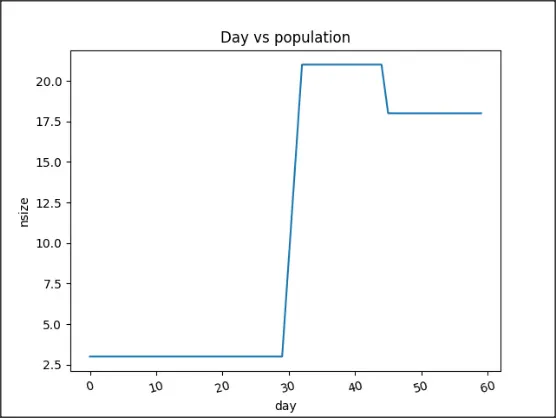
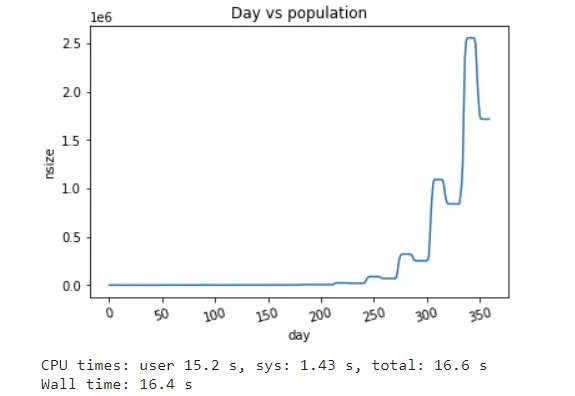
输出:
问题/疑问:
随着nday_limit的增加,执行时间呈指数增长。我需要提高代码效率。如何缩短运行时间?
其他想法:
我想尝试使用joblib,但令我惊讶的是,执行时间更差了。
def djob(dpol,k,**kwargs):
dpol['d'] = dpol['d'] + 1
dpol['dborn'] = dpol['dborn'] + 1
dpol,h = get_breeding(dpol,**kwargs)
if h is None and dpol['dborn'] <= kwargs['age_limit']:
# If beyond the age limit, ignore the that particular subject
k.append(dpol)
elif isinstance(h, list) and dpol['dborn'] <= kwargs['age_limit']:
# If below age limit, append the parent and its offspring
h.extend([dpol])
k.extend(h)
return k
def to_loop_initial_population(**kwargs):
npol=kwargs['ipol']
nday = 0
total_population_per_day = []
while nday < kwargs['nday_limit']:
k = []
njob=1 if len(npol)<=50 else 4
if njob==1:
print(f'Executing day {nday} with single cpu')
for dpols in npol:
k=djob(dpols,k,**kwargs)
else:
print(f'Executing day {nday} with single parallel')
k=Parallel(n_jobs=-1)(delayed(djob)(dpols,k,**kwargs) for dpols in npol)
k = list(itertools.chain(*k))
ll=1
total_population_per_day.append(dict(nsize=len(k), day=nday))
nday += 1
npol = k
return total_population_per_day
对于
nday_limit=365
age_limit?什么是age_limit?没有资源限制(如果有的话,我可以告诉你,人口要么呈指数增长,要么就会灭绝)。 - Bobto_loop_initial_population(**para)时,必须将para的所有值提取出来,并仅作为关键字值传递,以便再次重组成字典,因为您正在将单个参数声明为**kwargs。我认为,如果您将此函数声明为to_loop_initial_population(kwargs)或更好地声明为to_loop_initial_population(para)并使用to_loop_initial_population(para)调用它会更有效率。也就是说,显式地传递一个dict实例。 - Booboo