我是机器学习的新手,正在使用Keras中的LSTMs执行多变量时间序列预测。我有一个包含4个输入变量(温度、降水量、露点和风速)和1个输出变量(污染)的月度时间序列数据集。利用这些数据,我构建了一个预测问题,即在给定先前几个月的天气条件和污染情况的情况下,预测下一个月的污染情况。以下是我的代码:
X = df[['Temperature', 'Precipitation', 'Dew', 'Wind_speed' ,'Pollution (t_1)']].values
y = df['Pollution (t)'].values
y = y.reshape(-1,1)
from sklearn.preprocessing import MinMaxScaler
scaler = MinMaxScaler(feature_range=(0, 1))
scaled = scaler.fit_transform(X)
#dataset has 359 samples in total
train_X, train_y = X[:278], y[:278]
test_X, test_y = X[278:], y[278:]
# reshape input to be 3D [samples, timesteps, features]
train_X = train_X.reshape((train_X.shape[0], 1, train_X.shape[1]))
test_X = test_X.reshape((test_X.shape[0], 1, test_X.shape[1]))
print(train_X.shape, train_y.shape, test_X.shape, test_y.shape)
model = Sequential()
model.add(LSTM(100, input_shape=(train_X.shape[1], train_X.shape[2])))
model.add(Dropout(0.2))
# model.add(LSTM(70))
# model.add(Dropout(0.3))
model.add(Dense(1))
model.compile(loss='mean_squared_error', optimizer='adam')
history = model.fit(train_X, train_y, epochs=700, batch_size=70, validation_data=(test_X, test_y), verbose=2, shuffle=False)
# summarize history for loss
plt.plot(history.history['loss'])
plt.plot(history.history['val_loss'])
plt.title('model loss')
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['train', 'test'], loc='upper right')
plt.show()
为了进行预测,我使用以下代码。
from sklearn.metrics import mean_squared_error,r2_score
yhat = model.predict(test_X)
mse = mean_squared_error(test_y, yhat)
rmse = np.sqrt(mse)
r2 = r2_score(test_y, yhat)
print("test set performance")
print("--------------------")
print("MSE:",mse)
print("RMSE:",rmse)
print("R^2: ",r2)
fig, ax = plt.subplots(figsize=(10,5))
ax.plot(range(len(test_y)), test_y, '-b',label='Actual')
ax.plot(range(len(yhat)), yhat, 'r', label='Predicted')
plt.legend()
plt.show()
运行此代码时,我遇到了以下问题:
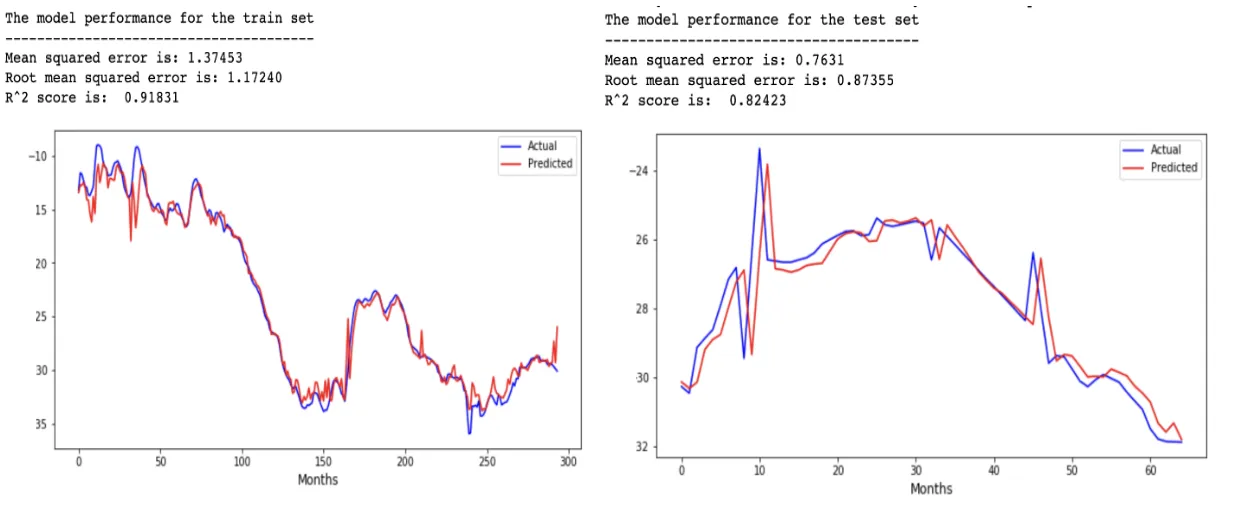
- 由于一些原因,我的测试集产生了滞后的结果,这种情况在我的训练数据中并不存在,如下图所示。我不明白为什么会出现这种滞后的结果(这是否与将“pollution(t_1)”包括在我的输入中有关?)
图形结果:
- 通过将“污染物(t_1)”作为输入的一部分添加进去,它现在似乎支配着预测,因为删除其他变量似乎对我的结果(r-squared和rmse)没有影响,这很奇怪,因为所有这些变量都有助于污染物预测。
我在我的代码中做错了什么导致出现这些问题吗?我是Python新手,非常感谢您对上述两个问题的帮助。
train_X = train_X.reshape((train_X.shape[0], 1, train_X.shape[1]))替换为train_X = train_X.reshape(-1, 4, train_X.shape[1])吗?因为这个操作会导致以下错误:ValueError: cannot reshape array of size 1390 into shape (4,5)。 - Deniseprint(train_X.shape, train_y.shape, test_X.shape, test_y.shape),我会得到:(278, 1, 5) (278, 1) (81, 1, 5) (81, 1)。 - Denise