我正在使用nls()函数将逻辑回归模型(自启动;SSlogis)拟合到多个鸟类种群的数据上。我的目标是仅使用每个数据集的一部分来拟合期望函数,并在图表上显示关于期望的方差度量。然后,我想拟合和绘制观察到的函数(对于每个种群使用整个数据集),以确定观察到的动态是否落在期望值的方差之内。以下是我目前编写的代码以实现此目的:
CE.mod = nls(CE.observed ~ SSlogis(t.CattleEgret, Asym, xmid, scal))
with(collapse.data, plot(CE.time, CE.obs))
CE.extrap = predict(CE.mod, data.frame(t.CattleEgret = CE.time))
lines(CE.time, CE.extrap)
CE.se.fit = sqrt(apply(attr(CE.extrap, "gradient"), 1, function(x)
sum(vcov(CE.mod)*outer(x,x))))
matplot(CE.time, CE.extrap+outer(CE.se.fit, qnorm(c(0.5, 0.025, 0.975))),
type = "l", lty = c(1,1,1), ylab = "Abundance (# per party hour)",
xlab = "Time (year)", main = "Cattle Egret Collapse Analysis",
pch = 15, font.lab = 2, font.axis = 2, cex = 4, cex.lab = 1.5,
cex.axis = 2, cex.main = 2, frame.plot = FALSE, lwd = 4, 10)
with(collapse.data, matpoints(CE.time, CE.obs, pch = 15, cex = 3))
lines(CE.time, predict(nls(CE.obs ~ SSlogis(log(CE.time),
Asym, xmid, scal))), lty = 3, lwd = 4)
来源于“collapse.data”文件:
t.CattleEgret = c(1:20)
CE.time = c(1:45)
CE.obs = c(0.3061324, 0.0000100, 0.2361211, 0.5058240, 2.0685032, 2.1944544,
4.2689494, 4.9508297, 3.1334720, 3.6570752, 5.6753381, 10.9133183,
5.4518257, 20.4166979, 15.9741054, 19.0970426, 13.7559959, 14.1358153,
15.9986416, 29.6762828, 10.3760667, 8.4284488, 6.1060359, 3.7099982,
3.3584060, 2.5981386, 2.5697082, 2.8091952, 5.5487979, 1.6505442,
2.2696972, 2.1835692, 3.6747876, 4.8307886, 3.5019731, 2.8397137,
1.8605288, 11.1848738, 2.6268683, 4.1215127, 2.3996210, 2.6569938,
2.1987387, 3.0267252, 2.4420927)
CE.observed = c(0.3061324, 0.0000100, 0.2361211, 0.5058240, 2.0685032, 2.1944544,
4.2689494, 4.9508297, 3.1334720, 3.6570752, 5.6753381, 10.9133183,
5.4518257, 20.4166979, 15.9741054, 19.0970426, 13.7559959, 14.1358153,
15.9986416, 29.6762828)
那段代码运行良好,会生成如下的图像:
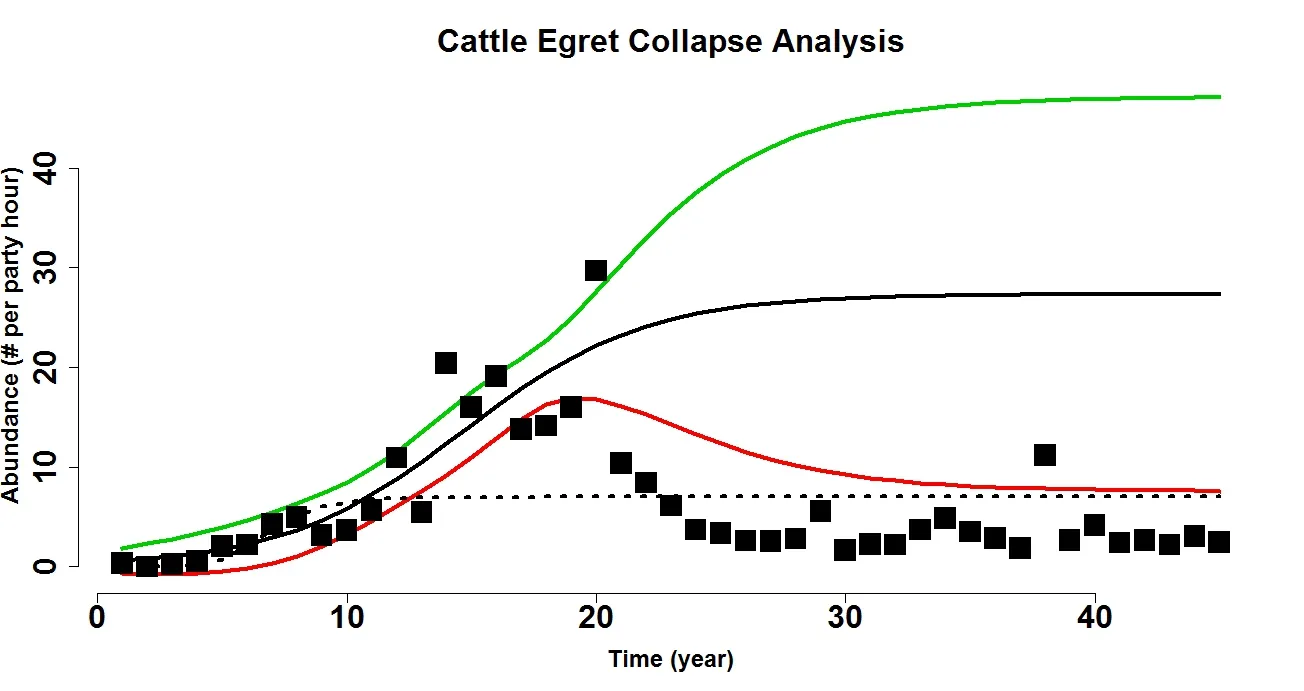
但是,如果我在代码的最后一行删除“log()”,以便写成这样:
lines(CE.time, predict(nls(CE.obs ~ SSlogis(CE.time,
Asym, xmid, scal))), lty = 3, lwd = 4),
该行不会绘制,并显示以下错误:
Error in nls(y ~ 1/(1 + exp((xmid - x)/scal)), data = xy, start = list(xmid =
aux[1L], : step factor 0.000488281 reduced below 'minFactor' of 0.000976562
即使我玩弄nls.controls并改变“minFactor”值,我也无法更改。对于某些人群,在定义mod(##.mod部分)的初始行后,我也会收到此错误消息。
此外,对于一些人群,在报告以下内容的代码最后一行后,我会收到错误消息:
Error in qr.solve(QR.B, cc) : singular matrix 'a' in solve
我想不出自然对数转换数据的合理理由,所以我认为我只是任意地改变了数据(在这种情况下任意记录了它),以便让predict()和SSlogis()函数正常运行,但我不知道原因。我在任何论坛中都找不到适当的答案来解决这个问题。非常感谢任何帮助。
*更新:我已经尝试实现Roland(下面)推荐的nlsLM函数。这确实清理了具有混淆log()使用的代码部分。
lines(CE.time, predict(nlsLM(CE.obs ~ Asym/(1 + exp((xmid - CE.time)/scal)), start
= list(Asym = max(CE.obs), xmid = popsizetime[1], scal = 1), control =
nls.lm.control(maxiter = 1000))
然而,对于其他人群,在初始模型规范中我遇到了与上述相同的错误信息:
ChMa.mod = nls(ChMa.observed ~ SSlogis(t.ChestnutMannikin, Asym, xmid, scal))
Error in nls(y ~ 1/(1 + exp((xmid - x)/scal)), data = xy, start = list(xmid =
aux[1L], : step factor 0.000488281 reduced below 'minFactor' of 0.000976562
切换至:
ChMa.mod = nlsLM(ChMa.observed ~ Asym/(1 + exp((xmid - t.ChestnutMannikin)/
scal)), start = list(Asym = max(ChMa.obs), xmid = popsizetime[2],
scal = 1), control = nls.lm.control(maxiter = 1000))
在哪里
ChMa.observed = c(4.02785074, 0.33847154, 0.99029776, 2.86516540, 0.59588068,
0.01334333, 2.07693362, 0.62485994, 3.48979515, 3.67785202, 20.84180181)
t.ChestnutMannikin = c(1:11)
popsizetime[2] = 11
虽然这个开关避免了错误消息,但nlsLM评估函数却不评估梯度。没有梯度的评估,我无法使用se.fit代码,因此无法获得绘图方差的估计。
minpack.lm包中的nlsLM。我试了一下,看起来似乎可以运行,但是我没有时间检查结果是否正确,所以我不会发布答案。 - Rolandnls和nlsLM之间;nls可以使用不同的初始条件正常工作。 - Ben Bolker