一位不熟悉ggplot2的lattice图形用户需要帮助:如何请求在直方图中每个facet上按变量进行分割的语法?
library(ggplot2)
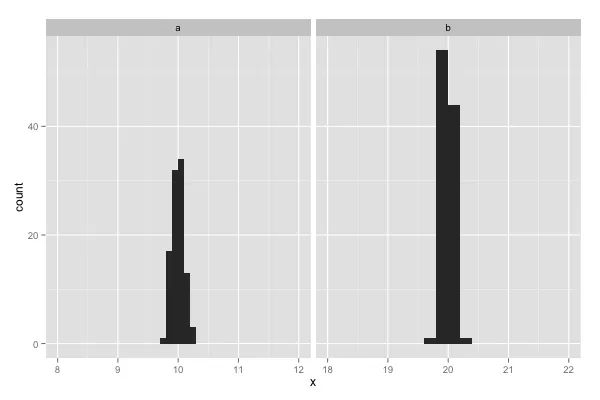
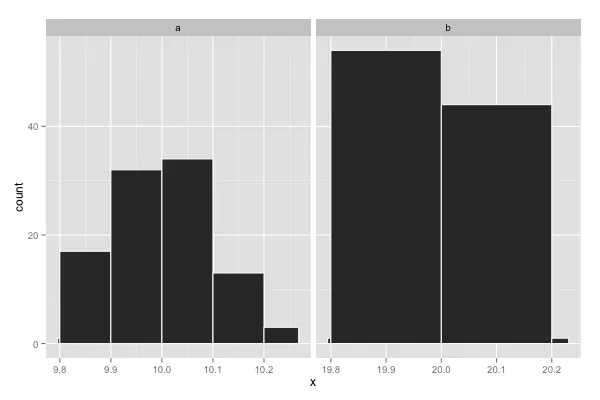
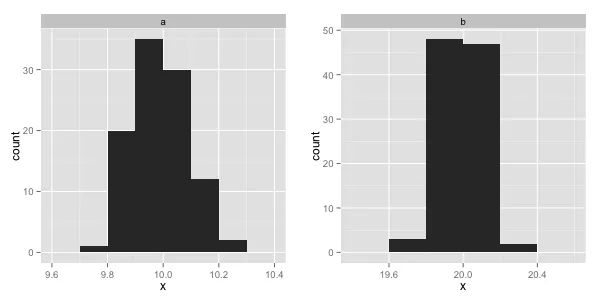
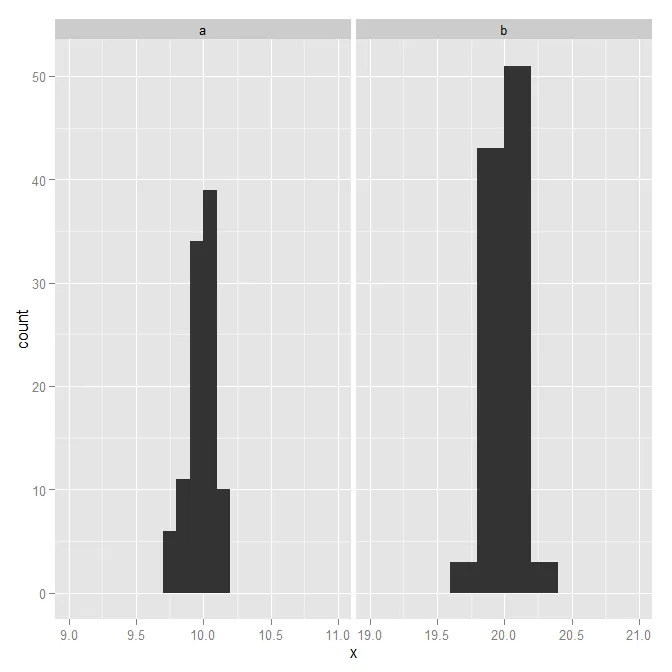
d = data.frame(x=c(rnorm(100,10,0.1),rnorm(100,20,0.1)),par=rep(letters[1:2],each=100))
# Note: breaks have different length by par
breaks = list(a=seq(9,11,by=0.1),b=seq(19,21,by=0.2))
ggplot(d, aes(x=x) ) +
geom_histogram() + ### Here the ~breaks should be added
facet_wrap(~ par, scales="free")
如jucor所指出的,这里提供了更多解决方案。
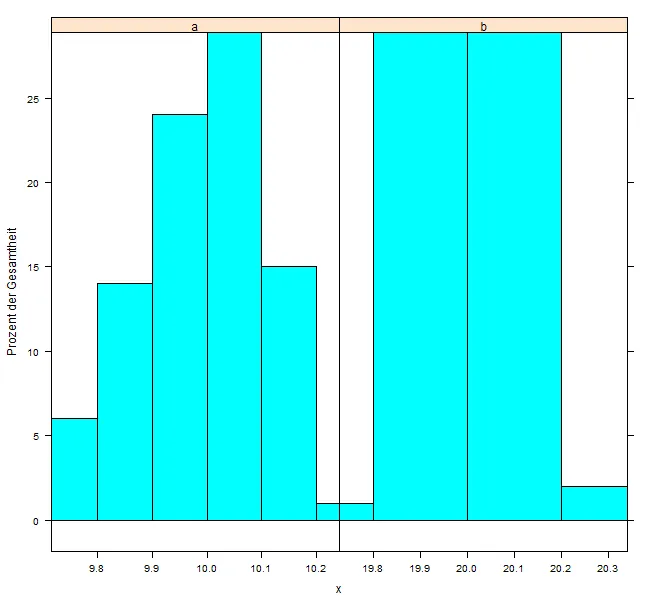
应特殊要求,并展示我为何不是一个伟大的ggplot粉丝,这是lattice版本。
library(lattice)
d = data.frame(x=c(rnorm(100,10,0.1),rnorm(100,20,0.1)),par=rep(letters[1:2],each=100))
# Note: breaks have different length by par
myBreaks = list(a=seq(8,12,by=0.1),b=seq(18,22,by=0.2))
histogram(~x|par,data=d,
panel = function(x,breaks,...){
# I don't know of a generic way to get the
# grouping variable with histogram, so
# this is not very generic
par = levels(d$par)[which.packet()]
breaks = myBreaks[[par]]
panel.histogram(x,breaks=breaks,...)
},
breaks=NULL, # important to force per-panel compute
scales=list(x=list(relation="free")))
hist结合ddply或data.table等方式,并制作条形图。 - Roland