我需要将重叠的片段聚合成一个范围涵盖所有连接的片段。
请注意,简单的foverlaps无法检测非重叠但相互连接的片段之间的连接,参见示例以了解更清楚的情况。如果我的绘图中的片段遭受雨淋,我正在寻找干燥地面的伸展区域。
到目前为止,我通过迭代算法来解决这个问题,但我想知道是否有更优雅和直接的方法来解决这个问题。我确定不是第一个面对这个问题的人。
我考虑过非等滚动连接,但未能实现。
请注意,简单的foverlaps无法检测非重叠但相互连接的片段之间的连接,参见示例以了解更清楚的情况。如果我的绘图中的片段遭受雨淋,我正在寻找干燥地面的伸展区域。
到目前为止,我通过迭代算法来解决这个问题,但我想知道是否有更优雅和直接的方法来解决这个问题。我确定不是第一个面对这个问题的人。
我考虑过非等滚动连接,但未能实现。
library(data.table)
(x <- data.table(start = c(41,43,43,47,47,48,51,52,54,55,57,59),
end = c(42,44,45,53,48,50,52,55,57,56,58,60)))
# start end
# 1: 41 42
# 2: 43 44
# 3: 43 45
# 4: 47 53
# 5: 47 48
# 6: 48 50
# 7: 51 52
# 8: 52 55
# 9: 54 57
# 10: 55 56
# 11: 57 58
# 12: 59 60
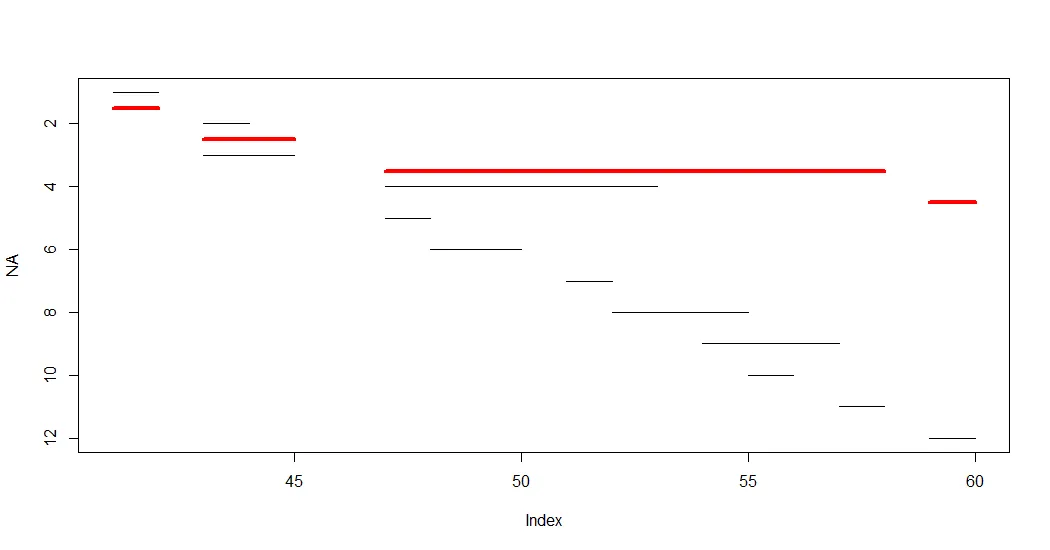
setorder(x, start)[, i := .I] # i is just a helper for plotting segments
plot(NA, xlim = range(x[,.(start,end)]), ylim = rev(range(x$i)))
do.call(segments, list(x$start, x$i, x$end, x$i))
x$grp <- c(1,3,3,2,2,2,2,2,2,2,2,4) # the grouping I am looking for
do.call(segments, list(x$start, x$i, x$end, x$i, col = x$grp))
(y <- x[, .(start = min(start), end = max(end)), k=grp])
# grp start end
# 1: 1 41 42
# 2: 2 47 58
# 3: 3 43 45
# 4: 4 59 60
do.call(segments, list(y$start, 12.2, y$end, 12.2, col = 1:4, lwd = 3))
编辑:
太棒了,谢谢cummax和cumsum的帮助,Uwe的答案比David的评论略好。
end[.N]可能会得到错误的结果,请尝试下面的示例数据x。 在所有情况下,max(end)都是正确的,而且速度更快。x <- data.table(start = c(11866, 12696, 13813, 14011, 14041), end = c(13140, 14045, 14051, 14039, 14045))min(start)和start[1L]相同(因为x按start排序),但后者更快。- 即时生成grp的速度要快得多,不幸的是我需要分配grp。
cumsum(cummax(shift(end, fill = 0)) < start)比cumsum(c(0, start[-1L] > cummax(head(end, -1L))))快得多。- 我没有测试过GenomicRanges包的解决方案。
x[, .(start[1L], end[.N]), by = .(grp = cumsum(c(0, start[-1L] > cummax(head(end, -1L)))))]可以工作。基本上是一个数据表版本的我的解决方案在这里。 - David Arenburg