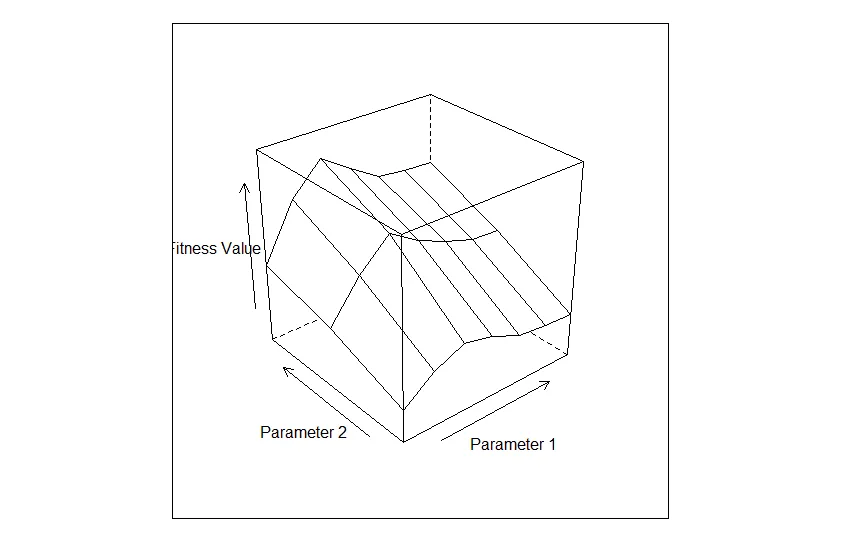
 我正在尝试使用3D表面图来分析结果的稳定性,作为输入变量的函数。z是我的结果,x和y是参数值。使用lattice的wireframe可以轻松制作一个表面图,但是拥有plotly的交互和可共享的能力会更好。volcano示例没有展示如何将x、y、z坐标点组合成表面图。有人知道如何简洁地做到这一点吗?以下是lattice wireframe示例和数据:
我正在尝试使用3D表面图来分析结果的稳定性,作为输入变量的函数。z是我的结果,x和y是参数值。使用lattice的wireframe可以轻松制作一个表面图,但是拥有plotly的交互和可共享的能力会更好。volcano示例没有展示如何将x、y、z坐标点组合成表面图。有人知道如何简洁地做到这一点吗?以下是lattice wireframe示例和数据:
library(lattice)
z <- c(0.035, 0.043, 0.036, 0.037, 0.016, 0.013, 0.025, 0.037, 0.039, 0.043,
0.049, 0.029, 0.025, 0.02, 0.032, 0.045, 0.049, 0.043, 0.027, 0.031,
0.028,0.039, 0.038, 0.037, 0.045, 0.046, 0.04, 0.041, 0.037, 0.048, 0.048,
0.032, 0.03, 0.032, 0.029, 0.043, 0.056, 0.057, 0.046, 0.034, 0.035,
0.026,0.036, 0.048, 0.038, 0.032, 0.034, 0.027, 0.024, 0.041, 0.05, 0.056,
0.038, 0.027, 0.034, 0.027, 0.042, 0.054, 0.054, 0.039, 0.036, 0.029,
0.027,0.04, 0.052, 0.058, 0.051, 0.028, 0.025, 0.035, 0.037, 0.053, 0.059,
0.049, 0.044, 0.035, 0.021, 0.044, 0.059, 0.054, 0.052, 0.041, 0.032,
0.036,0.039, 0.051, 0.056, 0.038, 0.04, 0.022, 0.015, 0.043, 0.054, 0.052,
0.05, 0.031, 0.03, 0.03, 0.031, 0.044, 0.051, 0.047, 0.04, 0.038,
0.037,0.06, 0.071, 0.07, 0.066, 0.038, 0.031, 0.045, 0.064, 0.065, 0.074,
0.08, 0.052, 0.048, 0.041, 0.053, 0.072, 0.079, 0.071, 0.043, 0.059, 0.05,
0.061, 0.061, 0.055, 0.069, 0.07, 0.057, 0.057, 0.055, 0.074, 0.071, 0.051,
0.048, 0.05, 0.053, 0.069, 0.086, 0.085, 0.07, 0.047, 0.05, 0.047,0.055,
0.073, 0.065, 0.056, 0.054, 0.048, 0.043, 0.063, 0.078, 0.087, 0.068, 0.047,
0.052, 0.048, 0.065, 0.085, 0.084, 0.062, 0.049, 0.05, 0.05, 0.068, 0.084,
0.095, 0.089, 0.052, 0.052, 0.062, 0.062, 0.086, 0.09, 0.078, 0.062, 0.058,
0.038, 0.068, 0.095, 0.09, 0.082, 0.069, 0.057, 0.058,0.064, 0.082, 0.087,
0.055, 0.053, 0.032, 0.033, 0.067, 0.087, 0.087, 0.079, 0.058, 0.053, 0.057,
0.047, 0.069, 0.085, 0.075, 0.068, 0.063, 0.062,0.076, 0.089, 0.095, 0.086,
0.056, 0.053, 0.063, 0.082, 0.08, 0.099, 0.101, 0.067, 0.065, 0.058, 0.066,
0.086, 0.103, 0.088, 0.051, 0.072, 0.06, 0.08, 0.078, 0.067, 0.084, 0.086,
0.067, 0.066, 0.068, 0.087, 0.087, 0.071, 0.063, 0.065, 0.067, 0.087, 0.104,
0.108, 0.086, 0.057, 0.063, 0.056,0.064, 0.087, 0.084, 0.07, 0.063, 0.061,
0.06, 0.076, 0.095, 0.11, 0.088, 0.057, 0.062, 0.067, 0.079, 0.097, 0.105,
0.079, 0.055, 0.064, 0.066,0.087, 0.104, 0.12, 0.113, 0.063, 0.07, 0.075,
0.073, 0.104, 0.111, 0.091, 0.072, 0.07, 0.05, 0.083, 0.114, 0.11, 0.098,
0.083, 0.077, 0.07, 0.078, 0.098, 0.105, 0.069, 0.062, 0.045, 0.046, 0.083,
0.106, 0.106, 0.093, 0.077, 0.075, 0.073, 0.057, 0.088, 0.105, 0.094, 0.084,
0.082, 0.081)
x <- c(1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7,
10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10,13, 16, 19, 1,
4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16,
19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10,
13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4,
7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19,
1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13,
16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7,
10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1,
4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16,
19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10,
13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19,1, 4,
7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19,
1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10,13, 16, 19, 1, 4, 7, 10, 13,
16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7,
10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19, 1,
4, 7, 10, 13, 16, 19, 1, 4, 7, 10, 13, 16, 19)
y <- c(0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1,0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1, 0.1,
0.1, 0.1, 0.1, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.2,0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2, 0.2,
0.2, 0.2, 0.2, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3,0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3, 0.3,
0.3, 0.3, 0.3, 0.3)
wireframe(z ~ x * y, xlab="Parameter 1", ylab="Parameter 2", zlab="Fitness Value")
使用plotly,我想做类似的事情,但它总是说z需要是一个矩阵。而我不明白这如何映射到x和y。
library(plotly)
p <- plot_ly(x = x, y = y, z = z) %>% add_surface()
p
最好提供一个 data.table 的示例,因为我的数据实际上是在 data.table 中,其中 z 是一列,x 和 y 是另外两列。此外,还有许多其他参数列将用于绘图。
df.ex <- data.table(Output=z,Input1=x,Input2=y,Input3=rnorm(315),Input4 =
abs(rnorm(315)+.5))
wireframe(df.ex$Output ~ df.ex$Input1 * df.ex$Input2)
