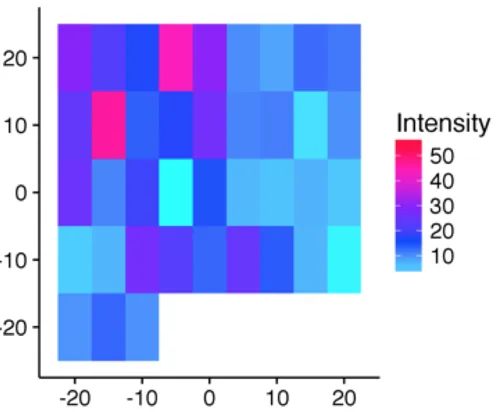
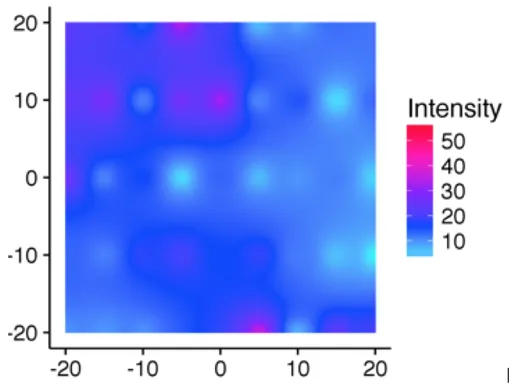
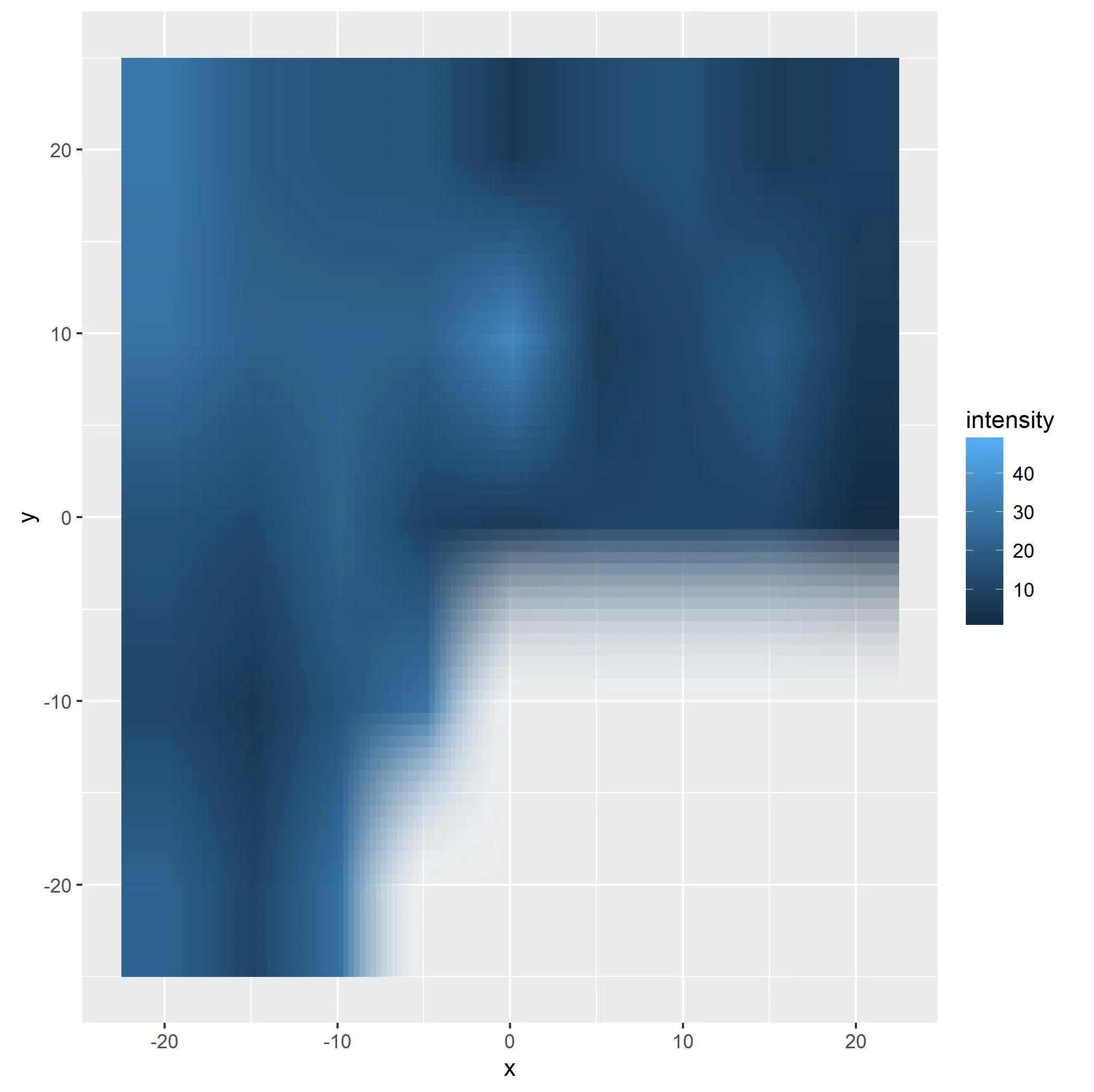
我有一个二维数据数组,其中包含一些缺失值。它有三列:
- x
- y
- intensity
我可以在 ggplot2 中将 x 与 y 绘制出来,用 intensity 作为颜色比例尺。
我想平滑颜色之间的过渡,并找到了 gstat 包中的 idw 函数。idw 旨在插值 2 维中的 NAs。它不应该外推,虽然从技术上讲它确实尊重数据的极限(±20 沿着两个方向),但它还试图填补图形边缘处的空隙,如下所示:
我想避免在我拥有的数据限制范围之外发生任何外推,包括第一张图中所示数据的右下角。
我该如何实现这一点?
编辑:这是一个示例数据集。它与上图显示的数据集不完全相同,但它再次包含一个大的缺失区域在右下角。
structure(list(x = c(10L, 15L, -10L, 0L, -5L, -10L, -15L, 0L,
-15L, 15L, 5L, 10L, -20L, -5L, -15L, -15L, -5L, 5L, 20L, -20L,
-15L, 20L, -15L, 5L, -5L, -20L, -5L, 15L, 0L, 0L, 15L, 10L, 0L,
20L, -10L, 5L, 5L, 0L, 20L, 5L, -15L, 5L, -5L, -5L, -15L, -10L,
-10L, -10L, -5L, -10L, 15L, 20L, 0L, 20L, -15L, 20L, -20L, -15L,
10L, 15L, 15L, -5L, 5L, 15L, 20L, 20L, -10L, -20L, -20L, 15L,
-10L, 10L, 5L, -20L, 20L, 10L, 0L, 10L, -10L, 0L, 10L, 10L, 10L,
-20L, 15L, -20L, 0L, -20L, -5L, 5L), y = c(0L, -10L, 0L, 20L,
0L, -10L, 0L, 0L, -20L, 20L, 0L, -10L, -10L, -10L, -10L, 20L,
10L, -10L, -20L, -20L, -10L, -10L, 0L, 10L, -20L, 20L, 0L, 0L,
0L, -20L, 0L, 0L, 10L, 10L, -20L, -20L, -10L, 20L, 10L, 20L,
10L, -20L, 20L, -10L, 20L, 20L, 10L, 10L, -20L, -10L, -10L, 20L,
-10L, -10L, -20L, 0L, -10L, 10L, -10L, 10L, -20L, 10L, 20L, 20L,
-20L, 20L, 0L, 10L, 10L, -20L, 20L, -20L, 10L, 0L, 0L, 10L, 10L,
-20L, -20L, -20L, 20L, 20L, 10L, 20L, 10L, -20L, -10L, 0L, 20L,
0L), intensity = c(12.9662, NA, 24.4379, 26.3923, 26.9449, 16.7372,
13.7691, 8.029, 11.922, 11.1967, 15.2792, NA, 14.4159, 20.6542,
22.0509, 17.356, 14.3841, NA, NA, 10.326, 6.0451, NA, 12.9515,
3.6745, NA, 18.1552, 9.9532, 9.9361, 7.0392, NA, 10.9814, 10.8351,
4.9017, 5.7864, 14.098, NA, NA, 6.3305, 6.4405, 49.2791, 19.9774,
NA, 25.1955, 28.5234, 20.2077, 20.3224, 12.688, 22.1371, NA,
17.5108, NA, 7.9351, NA, NA, 11.0975, 8.2349, 12.1194, 21.865,
NA, 10.7178, NA, 21.8222, 13.5971, 6.9751, NA, 8.8046, 22.0709,
14.2043, 27.8561, NA, 17.4329, NA, 7.4057, 15.2797, 1.0122, 11.1874,
35.5814, NA, 27.5919, NA, 11.8159, 15.8433, 12.297, 29.1978,
20.4151, 22.6336, NA, 16.0019, 16.9746, 10.8613)), .Names = c("x",
"y", "intensity"), row.names = c(NA, -90L), class = "data.frame")