好的,我找到了一个更简单的方法,通过注释原始ggplot盒图函数中的一些行并调用修改后的函数来完成。
我不是程序员,不知道这是否是一个好的或者健壮的做法,但目前看起来还可以。
以下是我正在使用的修改后的函数:
require(ggplot2)
geom_boxplot_noOutliers <- function (mapping = NULL, data = NULL, stat = "boxplot",
position = "dodge", outlier.colour = NULL,
outlier.shape = NULL, outlier.size = NULL,
notch = FALSE, notchwidth = .5, varwidth = FALSE,
...) {
GeomBoxplot_noOutliers$new(mapping = mapping, data = data, stat = stat,
position = position, outlier.colour = outlier.colour,
outlier.shape = outlier.shape, outlier.size = outlier.size, notch = notch,
notchwidth = notchwidth, varwidth = varwidth, ...)
}
GeomBoxplot_noOutliers <- proto(ggplot2:::Geom, {
objname <- "boxplot_noOutliers"
reparameterise <- function(., df, params) {
df$width <- df$width %||%
params$width %||% (resolution(df$x, FALSE) * 0.9)
if (is.null(params) || is.null(params$varwidth) || !params$varwidth || is.null(df$relvarwidth)) {
df$xmin <- df$x - df$width / 2
df$xmax <- df$x + df$width / 2
} else {
df$relvarwidth <- df$relvarwidth / max(df$relvarwidth)
df$xmin <- df$x - df$relvarwidth * df$width / 2
df$xmax <- df$x + df$relvarwidth * df$width / 2
}
df$width <- NULL
if (!is.null(df$relvarwidth)) df$relvarwidth <- NULL
df
}
draw <- function(., data, ..., fatten = 2, outlier.colour = NULL, outlier.shape = NULL, outlier.size = 2,
notch = FALSE, notchwidth = .5, varwidth = FALSE) {
common <- data.frame(
colour = data$colour,
size = data$size,
linetype = data$linetype,
fill = alpha(data$fill, data$alpha),
group = data$group,
stringsAsFactors = FALSE
)
whiskers <- data.frame(
x = data$x,
xend = data$x,
y = c(data$upper, data$lower),
yend = c(data$ymax, data$ymin),
alpha = NA,
common)
box <- data.frame(
xmin = data$xmin,
xmax = data$xmax,
ymin = data$lower,
y = data$middle,
ymax = data$upper,
ynotchlower = ifelse(notch, data$notchlower, NA),
ynotchupper = ifelse(notch, data$notchupper, NA),
notchwidth = notchwidth,
alpha = data$alpha,
common)
outliers_grob <- NULL
ggname(.$my_name(), grobTree(
outliers_grob,
GeomSegment$draw(whiskers, ...),
GeomCrossbar$draw(box, fatten = fatten, ...)
))
}
guide_geom <- function(.) "boxplot_noOutliers"
draw_legend <- function(., data, ...) {
data <- aesdefaults(data, .$default_aes(), list(...))
gp <- with(data, gpar(col=colour, fill=alpha(fill, alpha), lwd=size * .pt, lty = linetype))
gTree(gp = gp, children = gList(
linesGrob(0.5, c(0.1, 0.25)),
linesGrob(0.5, c(0.75, 0.9)),
rectGrob(height=0.5, width=0.75),
linesGrob(c(0.125, 0.875), 0.5)
))
}
default_stat <- function(.) StatBoxplot
default_pos <- function(.) PositionDodge
default_aes <- function(.) aes(weight=1, colour="grey20", fill="white", size=0.5, alpha = NA, shape = 16, linetype = "solid")
required_aes <- c("x", "lower", "upper", "middle", "ymin", "ymax")
})
我将它保存为一个r文件,并使用source加载它:
library(ggplot2)
library(scales)
source("D:/Eigene Dateien/Scripte/R-Scripte/myfunctions/geomBoxplot_noOutliers.r")
现在我可以使用
geom_boxplot_noOutliers绘制没有异常值的图形,即使有多个面板也可以正常工作 :-)
p1 <- ggplot(diamonds, aes(x=cut, y=price, fill=cut))
p1 + geom_boxplot_noOutliers() + facet_wrap(~clarity, scales="free")
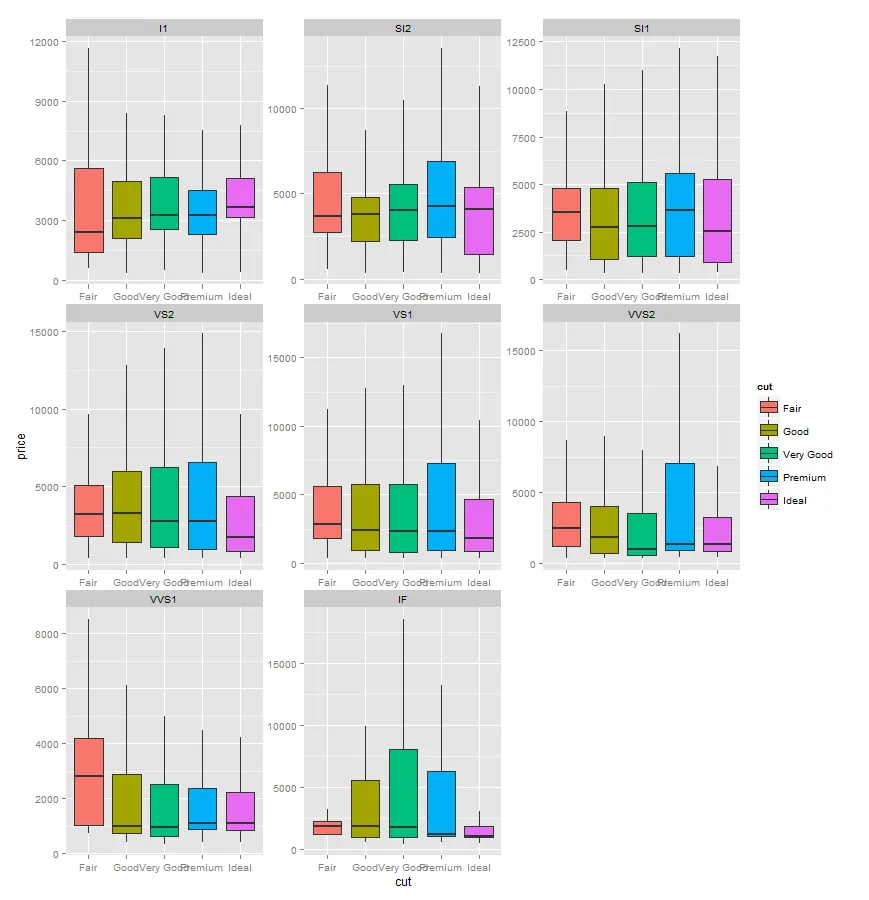
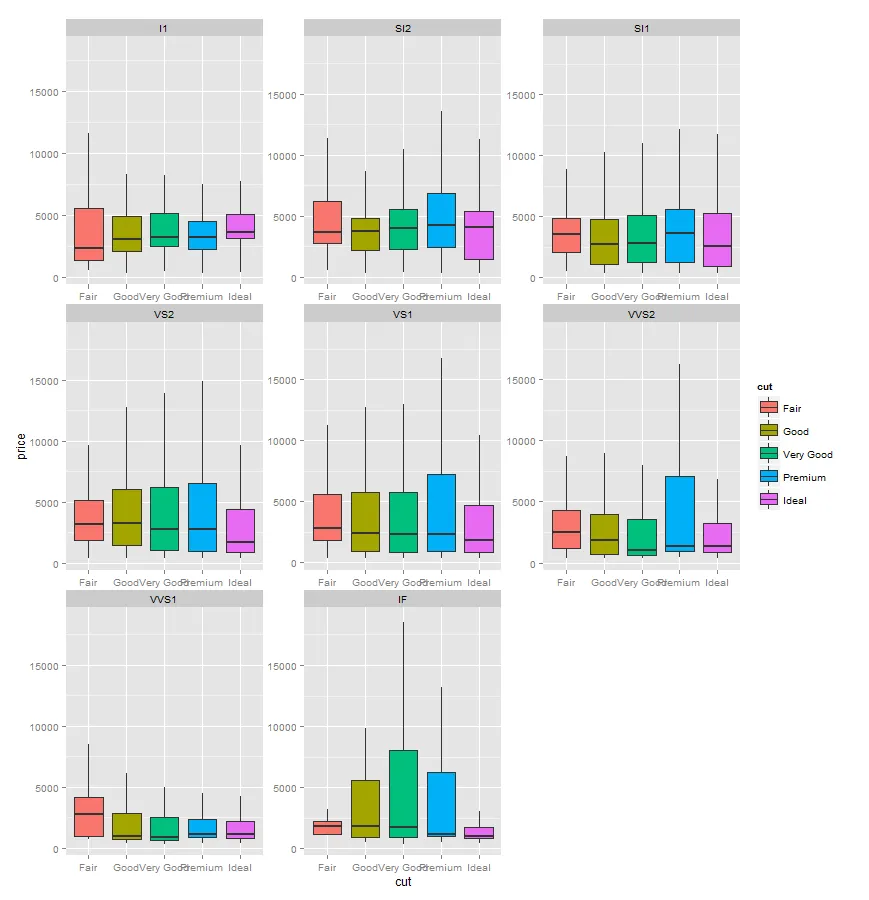
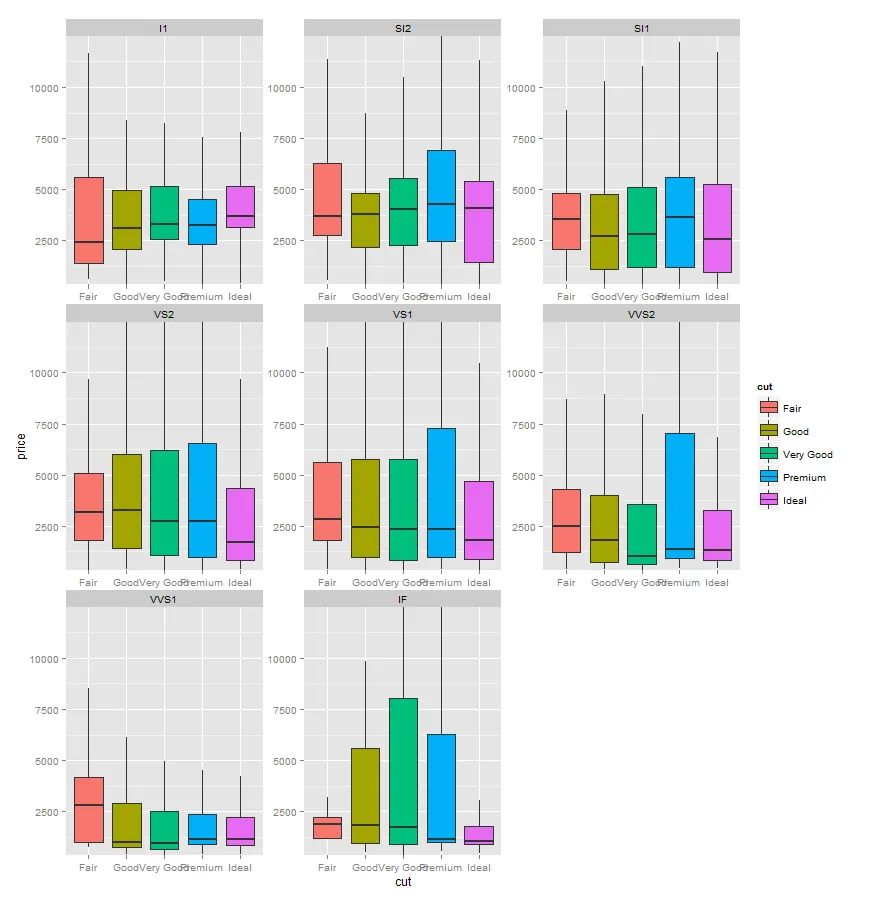
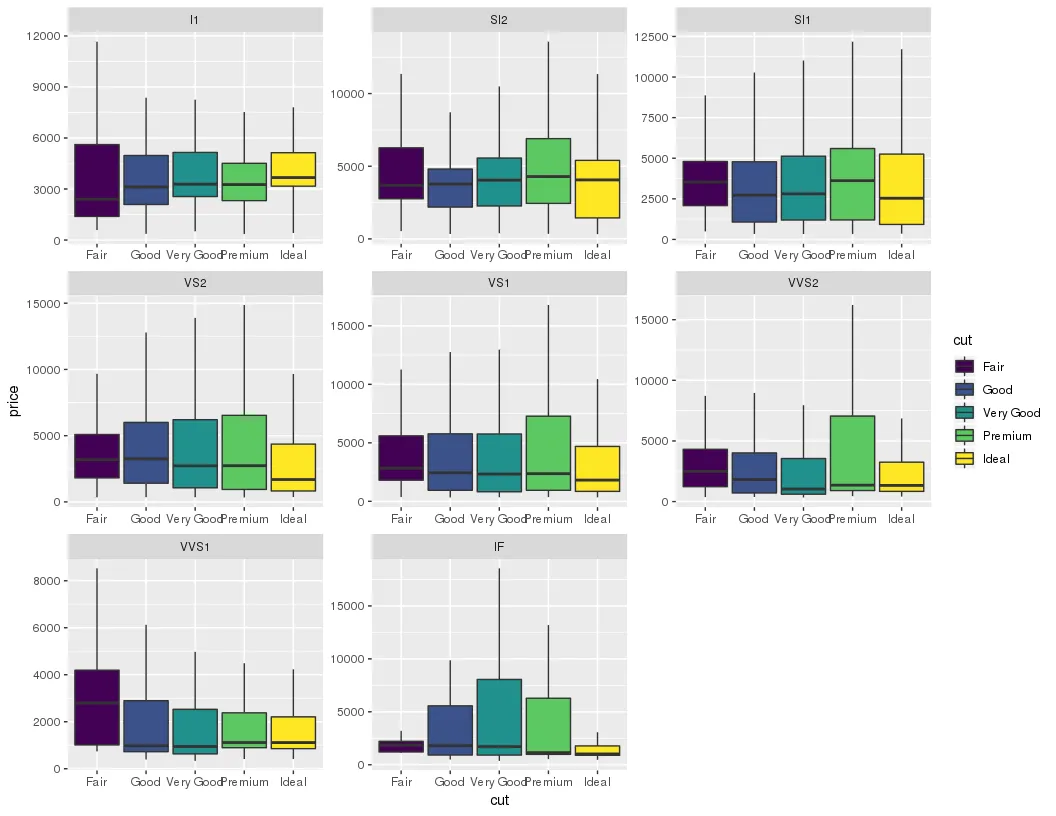
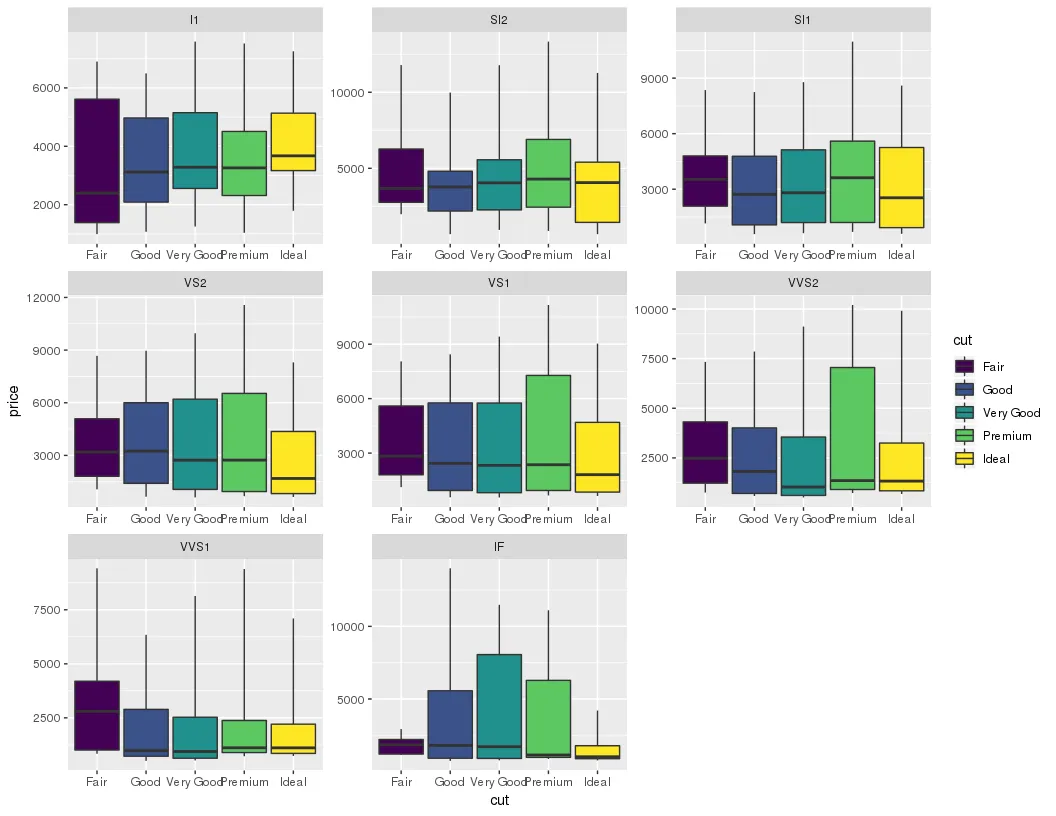
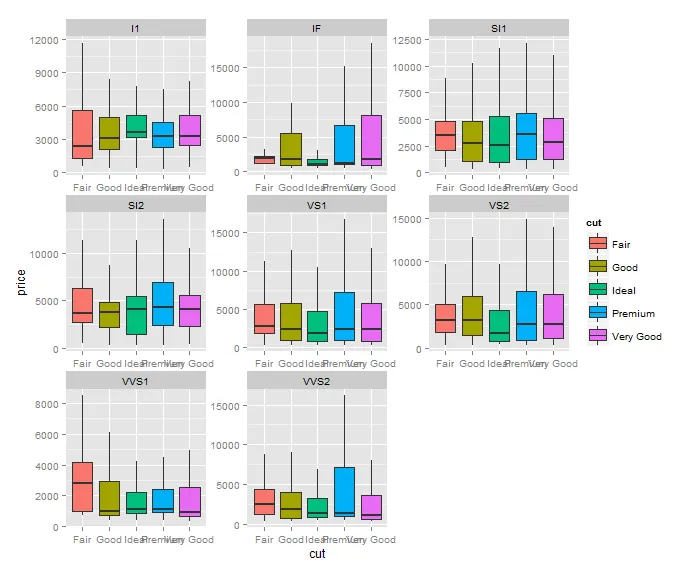

df$cut = factor(df$cut, levels = levels(diamonds$cut)))。 - aosmith