从你的问题本质来看,很可能是在进行多对多合并,其中每个学生在每个数据帧中都会出现多次。你可能需要检查每个学生出现的次数。如果每个学生在每个数据帧中均出现两次,则一个学生将生成4行。如果一个学生出现10次,则合并将增加100行。首先检查你将获得多少行,这是我用于此目的的函数:
count.rows <- function(x,y,v,all=FALSE){
tx <- table(x[[v]])
ty <- table(y[[v]])
val <- val <- names(tx)[match(names(tx),names(ty),0L) > 0L]
cts <- rbind(tx[match(val,names(tx))],ty[match(val,names(ty))])
colnames(cts) <- val
sum(apply(cts,2,prod,na.rm=all),na.rm=TRUE)
}
count.rows(DF1,DF2,"STUDENT.NAME")
如果你按照我所说的做(阅读R文档),你就会发现复杂度取决于答案的长度,这不是由于合并算法本身,而是由于所有结果的绑定。如果你真的想要更少占用内存的解决方案,你需要特别摆脱那个绑定。下面的算法可以帮助你做到这一点。我写出来是为了让你找到逻辑,并进行优化。请注意,它不会给出相同的结果,它会复制
两个数据框的
所有列。因此,你可能需要稍作调整。
mymerge <- function(x,y,v,count.only=FALSE){
ix <- match(v,names(x))
iy <- match(v,names(y))
xx <- x[,ix]
yy <- y[,iy]
ox <- order(xx)
oy <- order(yy)
xx <- xx[ox]
yy <- yy[oy]
nx <- length(xx)
ny <- length(yy)
val <- unique(xx)
val <- val[match(val,yy,0L) > 0L]
cts <- cbind(table(xx)[val],table(yy)[val])
dimr <- sum(apply(cts,1,prod),na.rm=TRUE)
idx <- vector("numeric",dimr)
idy <- vector("numeric",dimr)
ndx <- embed(c(which(!duplicated(xx)),nx+1),2)[unique(xx) %in% val,]
ndy <- embed(c(which(!duplicated(yy)),ny+1),2)[unique(yy) %in% val,]
count = 1
for(i in 1:nrow(ndx)){
nx <- abs(diff(ndx[i,]))
ny <- abs(diff(ndy[i,]))
ll <- nx*ny
idx[count:(count+ll-1)] <-
rep(ndx[i,2]:(ndx[i,1]-1),ny)
idy[count:(count+ll-1)] <-
rep(ndy[i,2]:(ndy[i,1]-1),each=nx)
count <- count+ll
}
x <- x[ox[idx],]
names(y) <- paste("y.",names(y),sep="")
x[names(y)] <- y[oy[idy],]
rownames(x) <- 1:nrow(x)
x
}
以下是一些测试代码,以便您可以看到它是如何工作的:
DF1 <- data.frame(
ID = 1:10,
STUDENT.NAME=letters[1:10],
SCORE = 1:10
)
id <- c(3,11,4,6,6,12,1,4,7,10,5,3)
DF2 <- data.frame(
ID = id,
STUDENT.NAME=letters[id],
SCORE = 1:12
)
mymerge(DF1,DF2,"STUDENT.NAME")
使用两个包含500,000行和4列的数据框进行相同操作,每个学生姓名最多有10个匹配项,返回一个包含5.8百万行和8列的数据框,并在内存上呈现以下图片:
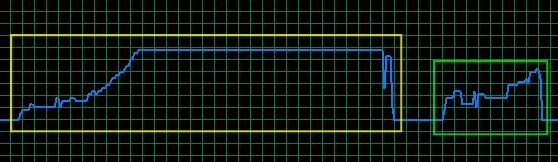
黄色框是合并调用,绿色框是mymerge调用。内存范围从2.3Gb到3.74Gb,因此merge调用使用1.45 Gb,mymerge略高于0.8 Gb。仍然没有“内存不足”错误......以下是测试代码:
Names <- sapply(
replicate(120000,sample(letters,4,TRUE),simplify=FALSE),
paste,collapse="")
DF1 <- data.frame(
ID10 = 1:500000,
STUDENT.NAME = sample(Names[1:50000],500000,TRUE),
FATHER.NAME = sample(letters,500000,TRUE),
SCORE1 = rnorm(500000),
stringsAsFactors=FALSE
)
id <- sample(500000,replace=TRUE)
DF2 <- data.frame(
ID20 = DF1$ID10,
STUDENT.NAME = DF1$STUDENT.NAME[id],
SCORE = rnorm(500000),
SCORE2= rnorm(500000),
stringsAsFactors=FALSE
)
id2 <- sample(500000,20000)
DF2$STUDENT.NAME[id2] <- sample(Names[100001:120000],20000,TRUE)
gc()
system.time(X <- merge(DF1,DF2,"STUDENT.NAME"))
Sys.sleep(1)
gc()
Sys.sleep(1)
rm(X)
gc()
Sys.sleep(3)
system.time(X <- mymerge(DF1,DF2,"STUDENT.NAME"))
Sys.sleep(1)
gc()
rm(X)
gc()

head(dataframe)),也许您可以得到更好的答案。 - kohske