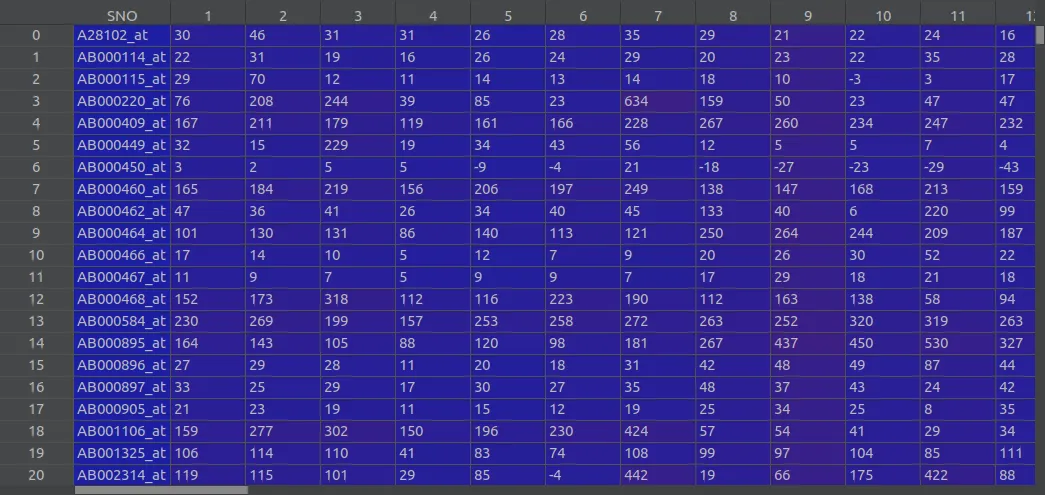
我有一个数据集,可以同时测量大量基因的表达水平。
这是我的数据框的部分内容
列0代表基因类型,其他列是患者样本。数据集中的样本代表患者。对于每个患者,为了将其疾病归类为以下几种情况之一:EPD、JPA、MED、MGL、RHB,测量了7070个基因的表达(值)。
我想要生成每个类别中具有最高绝对T值的前2、4、6、8、10、12、15、20、25和30个基因的子集。
我尝试使用scipy.stats.ttest_ind来进行每个可能的配对。
def calculate_t():
t_res = []
for cls in range(np.max(classes)):
samp = np.where(classes == cls)[0]
for gene in range(train.shape[1]):
for other_genes in range(gene, train.shape[1]):
t_res.append(ttest_ind(train[samp, gene], train[samp, other_genes])[:])
return t_res
我之前没有继续下去,因为我觉得这会花费太长时间。
如果有人有任何想法,我将非常感激。祝你有美好的一天。